用于显微镜的掩模自编码器是细胞生物学的可扩展学习
将显微镜图像特征化用于生物研究仍然是一个重大挑战,特别是对于跨越数百万张图像的大规模实验。这项工作探讨了弱监督分类器和自监督掩码自编码器(MAE),在使用越来越大的模型骨干和显微镜数据集进行训练时的缩放特性。结果表明,基于ViT的MAE在各种任务上的表现优于弱监督分类,在回忆从公共数据库中筛选出的已知生物关系时,相对提高了11.5%。此外,开发了一种新的通道无关MAE架构(CA-MAE),允许在推理时输入不同数量和通道顺序的图像。通过推断和评估在不同实验条件下生成的显微镜图像数据集(JUMP-CP),并使用与改进方法的预训练数据(RPI-93M)不同的通道结构,证明了CA MAE可以有效地进行泛化。研究结果激励继续在显微镜数据上扩展自监督学习,以创建强大的细胞生物学基础模型,这些模型有可能催化药物发现及其他领域的进步。
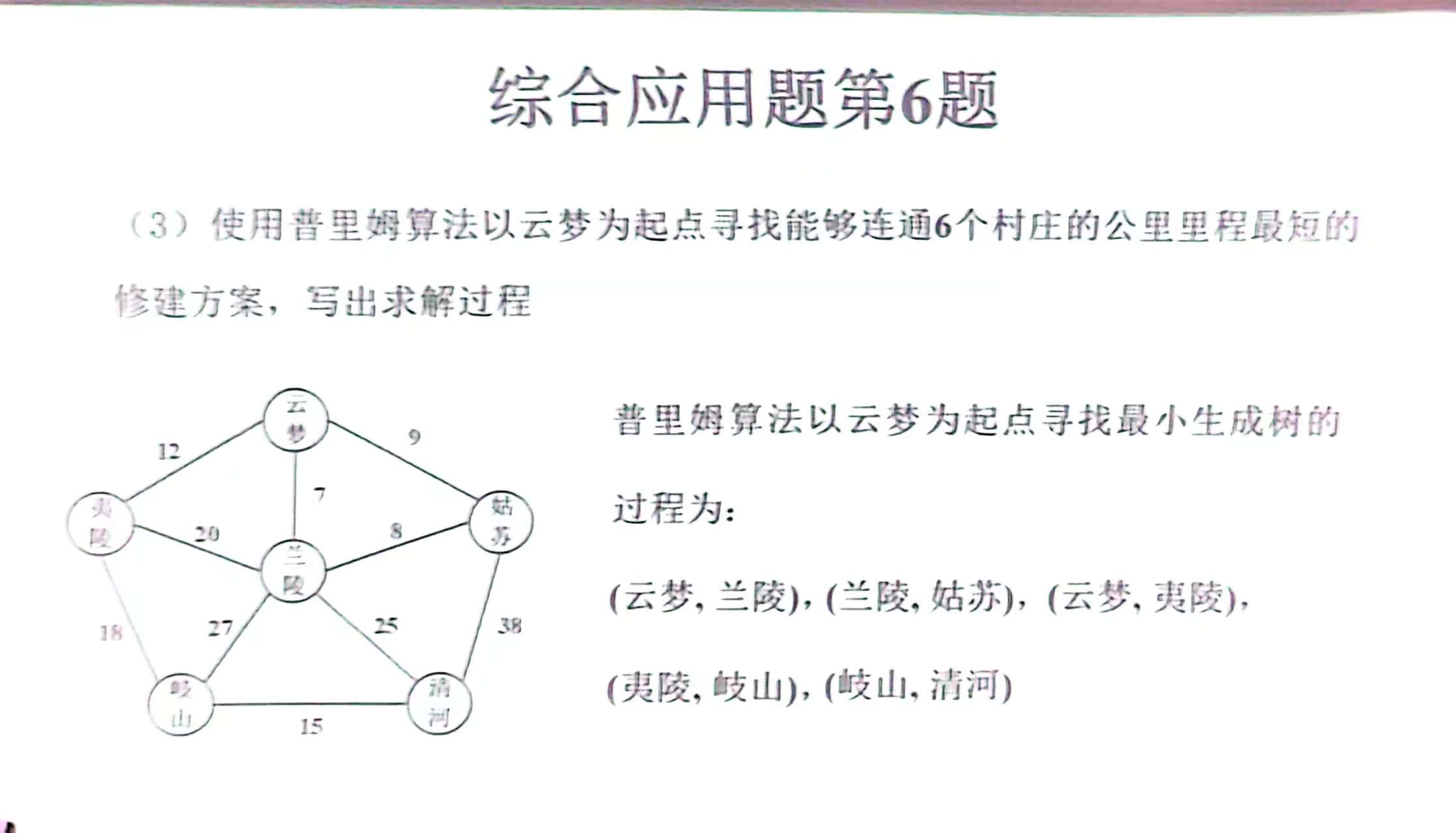
与渠道无关的MAE(CA-MAE),如图4-38所示。

图4-38 与渠道无关的MAE(CA-MAE)
在图4-38中,这种架构能够将使用MAE训练的ViT编码器从一组通道传输到另一组通道。左:CA-MAE训练(ViT-L/16+,85%掩码),其中输入张量被拆分为单独的通道,并对每个通道应用共享的线性投影(Tokenizer),然后为每个通道添加位置嵌入。右:训练好的ViT编码器可以通过使用类标记、对所有补丁嵌入进行平均,或分别对每个通道的补丁嵌入进行平均并将其连接起来,以便嵌入具有不同集合、顺序和/或通道数量(此处显示3个)的图像。




![VbaCompiler 1.6.4 注册分析[1]](https://img2023.cnblogs.com/blog/1145982/202412/1145982-20241219003311636-507062330.png)