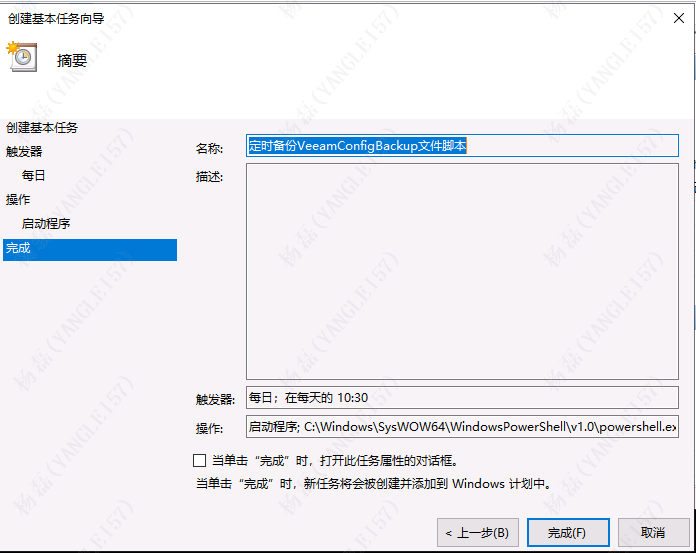
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
游离细胞DNA (Cell-free DNA,cfDNA)是血浆中游离的DNA片段,通常来源于正常细胞更新或病理状态下的细胞死亡。cfDNA已被广泛应用于癌症早期检测、胎儿遗传病诊断、器官移植评估等领域。然而,cfDNA在神经退行性疾病(如肌萎缩侧索硬化)中的应用仍处于探索阶段。肌萎缩侧索硬化(Amyotrophic lateral sclerosis ,ALS)是一种主要影响运动神经元的神经退行性疾病,导致肌肉无力、瘫痪,最终可能因呼吸衰竭而死亡。ALS诊断存在延迟,因为早期症状不特异,且缺乏可靠的生物标志物。
DNA甲基化是一种表观遗传修饰,能够影响基因表达,且具有组织和细胞特异性。在ALS患者中,中枢神经系统(CNS)组织和全血中已发现异常的DNA甲基化模式,但cfDNA甲基化组尚未被充分研究。
近日,埃默里大学医学院人类遗传学系Peng Jin团队在《Cell & Bioscience》杂志发表题为《Whole-genome bisulfite sequencing of cell-free DNA unveils age-dependent and ALS-associated methylation alterations》的研究论文,研究通过全基因组亚硫酸盐测序(WGBS),分析了血浆中的cfDNA甲基化模式,揭示了与年龄相关以及ALS相关的甲基化变化。并分析了cfDNA甲基化与ALS患者中枢神经系统基因表达之间的相关性。最后评估了cfDNA甲基化作为ALS生物标志物的潜力。

血浆中的cfDNA携带了其起源组织或细胞特异性的表观遗传学特征。循环cfDNA中异常的甲基化模式已成为非侵入性癌症检测、产前诊断和器官移植评估的有力工具。这些表观遗传学变化在诊断进展缓慢且具有长期无症状期的神经退行性疾病方面也显示出巨大潜力。然而,神经退行性疾病中cfDNA的全基因组甲基化变化尚未得到充分研究。
本研究通过对包括年轻和中年对照组以及与对照组匹配的ALS患者共30名个体的cfDNA进行全基因组亚硫酸盐测序(cfWGBS),分析了与年龄相关和ALS相关的甲基化特征。研究共鉴定出5223个与年龄相关的差异甲基化位点(DML),其中51.6%在老年个体中表现为低甲基化。本研究结果与一项大规模基于血液的表观基因组关联研究(EWAS)中鉴定的与年龄相关CpG位点有显著重叠。在ALS患者与对照组的比较中,共检测到1045个差异甲基化区域(DMRs),这些DMRs位于基因体(genebody)、启动子和基因间区域,且这些DMRs与ALS的关键相关通路(包括内吞作用和细胞黏附)相关。与脊髓转录组学数据整合分析显示,31%的DMR相关基因在ALS患者中表现出与对照组差异表达,其中超20个基因与疾病持续时间显著相关。此外,与已发表的ALS单核RNA测序(snRNA-Seq)数据比较表明,cfDNA甲基化变化揭示了ALS患者大脑中细胞类型特异性的基因失调,特别是在兴奋性神经元和星形胶质细胞中。cfDNA甲基化谱反卷积分析(deconvolution analysis)还提示ALS患者中免疫细胞和肝脏来源的cfDNA比例发生变化。
本研究结果表明,cfDNA甲基化是一种强大的工具,能够通过揭示受扰动的位点、基因以及不同组织/细胞对血浆的贡献比例,评估与年龄相关的改变和ALS特异性的分子失调。这种技术有望在广泛的神经退行性疾病中用于生物标志物发现的临床应用。
研究方法
样本收集:研究共纳入30名受试者,包括年轻和中年对照组,以及ALS患者和匹配的对照组。血浆样本通过非侵入性血液采样获得,cfDNA从血浆中提取。
cfWGBS测序:cfWGBS技术对cfDNA进行全基因组甲基化分析。该技术通过亚硫酸盐处理将未甲基化的胞嘧啶(C)转化为尿嘧啶(U),从而能够精确检测甲基化位点。
数据分析:比较年轻和中年对照组之间的甲基化差异,鉴定与年龄相关的差异甲基化位点。比较ALS患者和对照组之间的甲基化差异,识别差异甲基化区域。将cfDNA甲基化数据与脊髓转录组数据整合,分析ALS患者中基因表达变化。利用snRNA-Seq数据,分析cfDNA甲基化变化是否揭示ALS患者大脑中细胞类型特异性的基因调控。
组织/细胞溯源:通过反卷积分析,利用cfDNA的甲基化特征估计其组织和细胞来源。
结果图形
(1)cfDNA 中年龄相关的甲基化变化
研究发现5223个与年龄相关的差异甲基化位点,其中51.6%在老年个体中表现出低甲基化。这些位点与肌肉细胞的细胞骨架组织、轴突导向和钙信号通路相关。与大规模血液样本EWAS结果相比,研究中鉴定的差异甲基化位点与EWAS中鉴定的CpG位点有显著重叠。
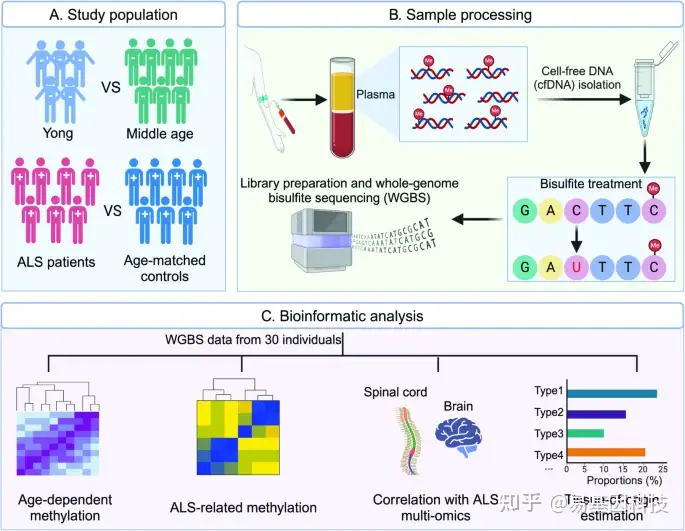
图1:利用血浆来源的 cfDNA 全基因组甲基化分析来鉴定年龄相关和ALS相关甲基化特征的研究工作流程

图2:鉴定 cfDNA 中与年龄相关的甲基化特征。
A.年轻和中年群体平均全基因组 mCG 水平条形图。组间未观察到甲基化水平的显著差异。
B. 两组之间 DML 的鉴定。
C. 两组之间鉴定的 DMLs 甲基化谱热图。
D. 已鉴定的 DML 的基因组注释到它们在每个基因组特征中的百分比。
E. DML 相关基因的 KEGG 分析以分析其生物学意义
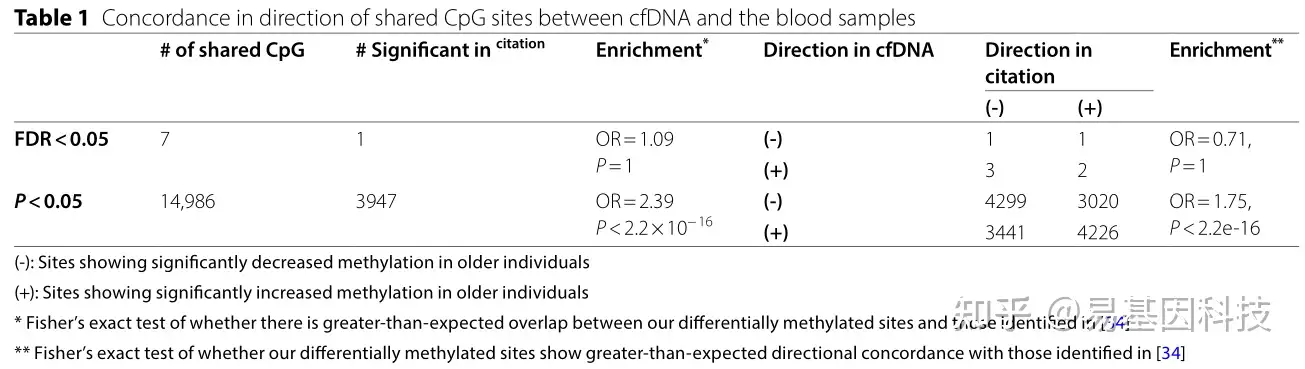
表1:cfDNA和血液样本之间共有 CpG 位点方向的一致性
(2)cfDNA中ALS相关的甲基化变化
在ALS患者和对照组之间鉴定出1045个DMRs,其中564个表现为低甲基化,481个表现为高甲基化。这些DMRs主要位于基因体、启动子和基因间区域,且与内吞作用和细胞黏附通路相关。与ALS相关的DMRs中,约21%位于CpG岛内。
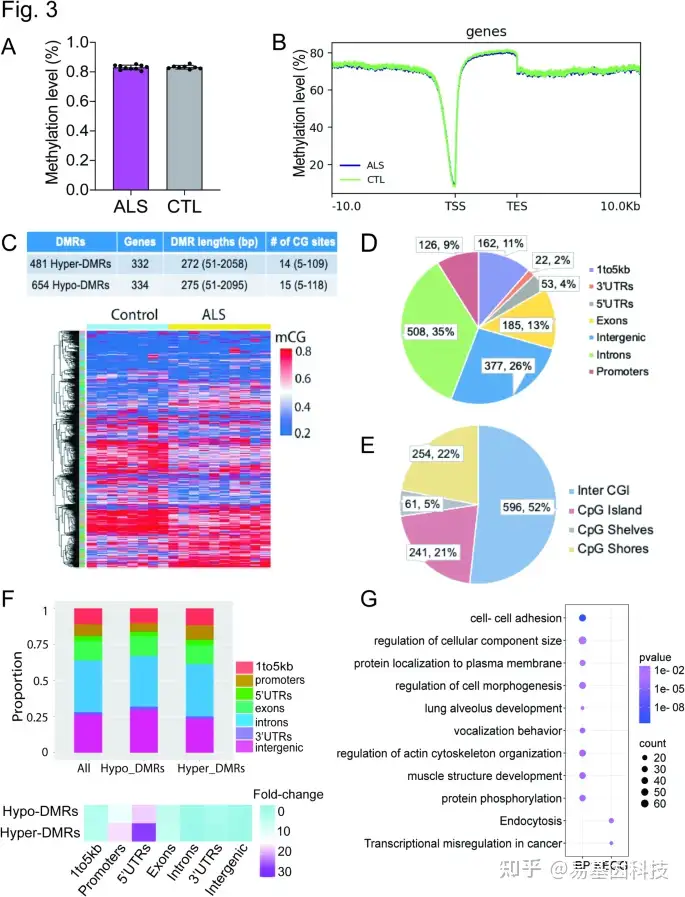
图3:ALS中cfDNA甲基化图谱特征。
- 条形图显示ALS组和对照组的全基因组mCG(甲基化胞嘧啶)平均水平。两组之间的甲基化水平未观察到显著差异。
- Metagene图显示在转录起始位点(TSS)、转录终止位点(TES)和RefSeq基因体的mCG(上)水平。
- ALS组和对照组之间的DMRs(上),并用热图(下)可视化所鉴定DMRs的甲基化谱。
- 对鉴定的DMRs进行基因组注释,显示其在每种基因组特征中的百分比。
- ALS相关DMRs在CpG岛(CGIs)、CpG架、CpG岸和其他基因组区域内的比例。
- 上:高甲基化DMRs和低甲基化DMRs在每种基因组特征中的百分比。下:DMRs相对于基因组背景的富集倍数。
- 对DMRs相关基因进行GO和KEGG通路分析,以探索其与ALS的功能相关性。
(3)cfDNA甲基化与基因表达的相关性
将ALS相关的DMRs与脊髓转录组数据整合,发现31%的DMR相关基因在ALS患者中表现出差异表达。进一步分析发现,与ALS进展相关的基因中,ARRB2和CYBA的表达水平与疾病持续时间显著相关。
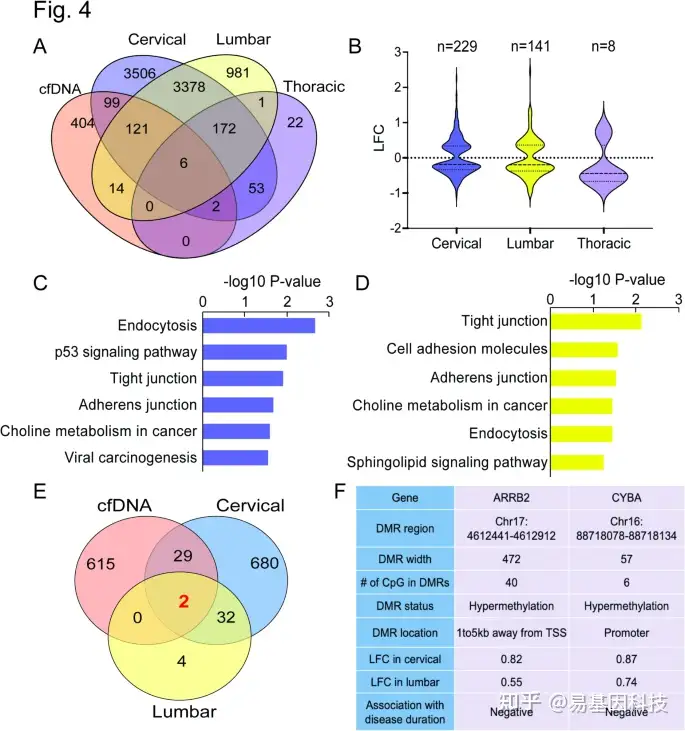
图4:ALS中cfDNA甲基化与脊髓基因表达的整合分析
- 维恩图展示cfDNA中DMR相关基因与ALS患者三个脊髓节段中鉴定的差异表达基因(DEGs)之间的重叠。
- 小提琴图显示与对照组相比,ALS患者脊髓中cfDNA差异甲基化的DEGs的log2倍数变化(LFC)。
C-D. 颈段和腰段脊髓中重叠基因的KEGG通路分析。
E. 维恩图展示与ALS疾病持续时间相关的基因与DMR相关基因之间的重叠。
F. 注释到ARRB2和CYBA的DMRs的详细信息,以及它们在脊髓中的基因表达统计信息。
(4)cfDNA甲基化揭示 ALS 中的细胞特异性基因调控
利用snRNA-Seq数据,研究发现cfDNA甲基化变化与ALS患者大脑中兴奋性神经元和星形胶质细胞的基因表达失调密切相关。在这些细胞类型中,DMR相关基因的差异表达比例较高,表明cfDNA甲基化可能反映细胞类型特异性的基因调控。
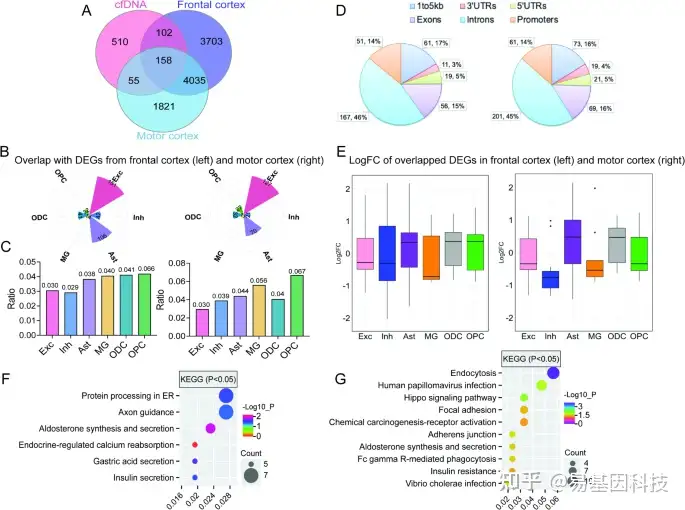
图5:ALS中cfDNA甲基化与snRNA-Seq数据的整合分析(靶向额叶皮层和运动皮层)
- 维恩图显示cfDNA甲基化DMR相关基因与ALS患者额叶皮层和运动皮层中鉴定的DEGs之间的重叠(与对照组相比)。
- 分别显示额叶皮层(左)和运动皮层(右)中六种主要细胞类型中与DMR相关基因重叠的DEGs数量。
- 条形图显示每种细胞类型中与cfDNA中的DMR相关的DEGs的百分比。
- 对重叠基因的DMR进行基因组注释,显示其在每种基因组特征中的百分比。
- 箱线图显示在额叶皮层(左)和运动皮层(右)中,每种主要细胞类型中cfDNA差异甲基化的DEGs的LogFC。
F-G. KEGG通路分析揭示重叠基因在ALS额叶皮层(左)和运动皮层(右)中的功能意义。
(5)cfDNA甲基化的组织/细胞溯源
反卷积分析显示,ALS患者中T细胞和B细胞来源的cfDNA比例降低,而肝脏来源的cfDNA比例增加。
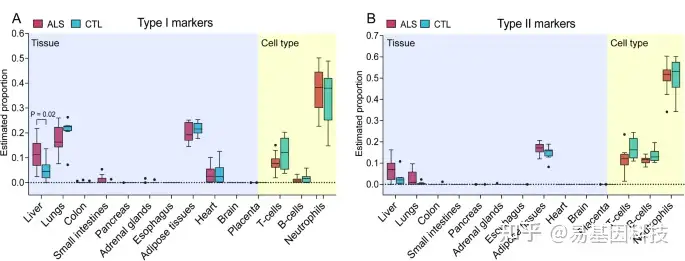
图6:基于WGBS数据的14种组织比例分析箱线图(使用I型和II型甲基化标记)。
ALS患者与对照组相比,来自T细胞和B细胞的cfDNA比例显著降低,而来自肝脏的cfDNA比例增加。
易小结
本研究通过cfWGBS技术揭示了cfDNA中与年龄和ALS相关的甲基化变化,并展示了cfDNA甲基化在反映ALS患者中枢神经系统基因表达失调方面的潜力。研究结果为cfDNA作为ALS生物标志物的应用提供了有力支持,并为未来的研究和临床应用奠定了基础。

易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化、cfDNA)和表观转录组学(m6A、m5C、m1A、m7G、ac4C、RNA与蛋白互作)、DNA与蛋白互作及染色质开放性技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
参考文献:
Jin Y, Conneely KN, Ma W, Naviaux RK, Siddique T, Allen EG, Guingrich S, Pascuzzi RM, Jin P. Whole-genome bisulfite sequencing of cell-free DNA unveils age-dependent and ALS-associated methylation alterations. Cell Biosci. 2025 Feb 20;15(1):26. pii: 10.1186/s13578-025-01366-1. doi: 10.1186/s13578-025-01366-1.
相关阅读:
1、游离细胞DNA(cfDNA)检测整体研究方案
2、干货:cfDNA甲基化测序实验怎么做,看完你就知道了
3、基于cfDNA甲基化的肿瘤液体活检如何开展?
4、课程回顾|表观遗传学--cfDNA甲基化疾病早筛研究思路分享
5、3文一览:基于cfDNA甲基化的液体活检在胰腺疾病中的研究进展
6、深度综述 | cfDNA甲基化诊断和监测肿瘤的研究进展与展望:胰腺癌
7、揭秘:cfDNA甲基化在器官和组织损伤检测中的强大力量
8、Nature子刊:cfDNA甲基化组多模式分析早期检测食管鳞状细胞癌和癌前病变
9、全基因组cfDNA甲基化分析提高了早期乳腺癌无创诊断成像的准确性
10、精准医学:新发现!全基因组cfDNA甲基化分析或可用于胰腺癌早期诊断应用
11、精准医学:结直肠癌诊断和监测的cfDNA甲基化标志物MYO1-G
12、前沿技术:cfDNA甲基化分析揭示造血细胞移植的所有主要并发症
13、新品:新型肿瘤标志物检测利器——cfDNA甲基化测序(cfDNA-RBS)
14、技术推介 | 微量cfDNA简化基因组甲基化测序(cfDNA-RBS)