上一期我们对孟德尔随机化做了一个简单的介绍,今天我们来复现一篇6分左右的使用了孟德尔随机化方法的文章,文章的题目是:Mendelian randomization analysis does not reveal a causal influence of mental diseases on osteoporosis(孟德尔随机分析没有揭示精神疾病对骨质疏松症的因果影响)

这是一篇我们国人写的文章,为什么选这篇文章呢?因为作者提供了文章的数据和主要流程的代码。我们可以跟着作者的思路进行一个复盘。
作者的研究是精神病(包括精神分裂症(SCH)、抑郁症(MDD)、情绪障碍、人格分裂和其他相关疾病)会不会对骨质疏松或骨折造成的影响。作者指出作为慢性疾病,精神病与骨代谢异常有关, 与一般人群相比,更有可能出现更低的骨密度和骨折风险,所以作者做了这项相关研究。最后作者的结论是:结果没有显示精神病(MDS)与骨质疏松症(OP)风险之间的因果关系。所以阴性结果也是能发文章的。
作者研究了精神分裂症、抑郁症、阿尔茨海默病、帕金森病等多个疾病对骨密度和骨折的影响,其中骨密度又分为前臂骨密度、腰椎骨密度、股骨颈骨密度等,骨折又分为手臂骨折、脊柱骨折、退步骨折等等。作者都做了一一比较,数据的工作量非常大。我抽取其中一部分内容:精神分裂症对骨密度(主要是上肢骨密度)的影响这一部分复现一下。
首先我们看看作者的给出的数据来源
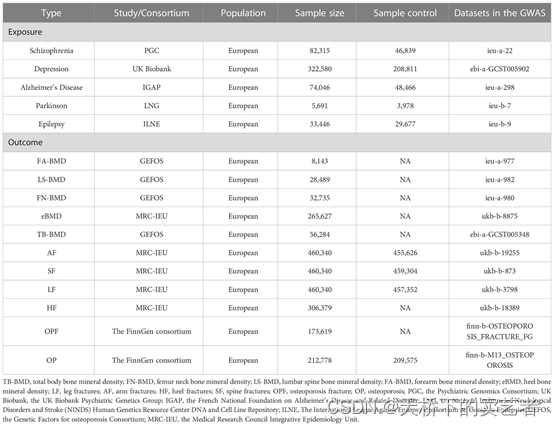
我们可以看到第一个精神分裂症(Schizophrenia)是来自GWAS的ieu-a-22,我们先查看一下对得上吗?ieu-a-22确实是精神病,样本量是82,315,和作者提供的对得上,我又拿它和作者提供的数据比对了一下,也是一样的。拿这个数据做暴露的数据应该是没问题。
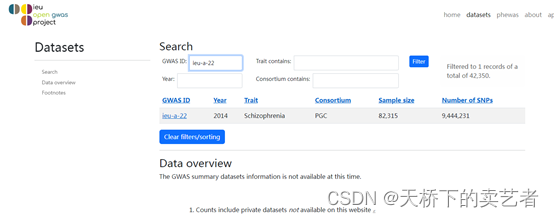
接下来看看结局变量前臂骨密度(FA-BMD)是来自GWAS的ieu-a-977,我们也像刚才一样检查一下,确实是前臂骨密度(Forearm bone mineral density),样本量8,143也对的上。这样两个数据就都对上了,

我们根据作者的代码来操作一下,先把R包导入
library(TwoSampleMR)
library(MRPRESSO)
先导入暴露变量数据(详细操作可以看上一章),作者在这里把暴露数据定义为aaa
aaa <- extract_instruments(outcomes='ieu-a-22',clump=TRUE, r2=0.001,kb=10000,access_token= NULL)
然后导入结果变量数据
abc <- extract_outcome_data(snps=aaa$SNP,outcomes='ieu-a-977',proxies = FALSE,maf_threshold = 0.01,access_token = NULL)
这样2个数据都生成好了
接下来按步骤进行:效应等位与效应量保持统一,这一步是必须的,
Mydata <- harmonise_data(exposure_dat=aaa,outcome_dat=abc,action= 2)
接下来就可以进行MR分析了,在这里作者定义了5种方法,包括固定效应和随机效应模型
res<-mr(Mydata, method_list=c("mr_ivw", "mr_ivw_fe", "mr_two_sample_ml","mr_egger_regression", "mr_weighted_median", "mr_penalised_weighted_median","mr_simple_mode", "mr_weighted_mode"))
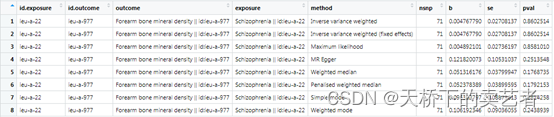
生成了结果,结论是一样的,精神病对前臂骨质疏松没关联。但是算出来和作者的不一样,我的OR和可信区间这里没有转换,但是我对照了P值不一样。
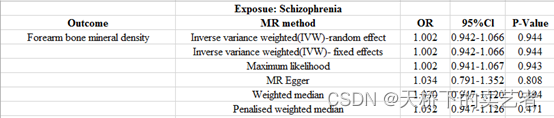
于是我看了一下作者提供的结果数据,我把它提取出来重新做一遍看看
这里顺便介绍一下怎么提取本地数据,使用read_excel这个函数提取数据很方便
Ins<-data <- read_excel("FA-BMD.outcome_dat.xlsx",1)

作者的提供的数据不是标准格式,我们需要对数据进行格式
outcome_dat<-format_data(Ins, type = "outcome", header = TRUE,phenotype_col = "Phenotype", snp_col = "SNPs", beta_col = "beta.outcome",se_col = "se.outcome", eaf_col = "eaf.outcome", effect_allele_col = "effect_allele.outcome",other_allele_col = "other_allele.outcome", pval_col = "pval.outcome")
进行数据效应等位与效应量保持统一,并行MR分析
Mydata <- harmonise_data(aaa, outcome_dat)res<-mr(Mydata, method_list=c("mr_ivw", "mr_ivw_fe", "mr_two_sample_ml","mr_egger_regression", "mr_weighted_median", "mr_penalised_weighted_median","mr_simple_mode", "mr_weighted_mode"))
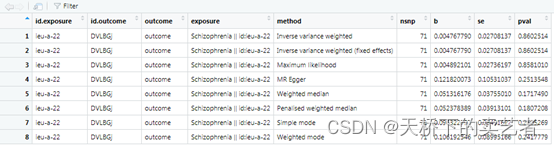
我无论自己提取数据还是使用作者的数据两次算出来的结果都是一样的(使用了作者的代码),但是和作者算出来的结论虽然一样,P值却不一样。
接下来作者还进行了一个(MR-PRESSO)检验,这个也是多水平效应检验,P值应该要大于0.05
mr_presso(BetaOutcome ="beta.outcome", BetaExposure = "beta.exposure", SdOutcome ="se.outcome", SdExposure = "se.exposure", OUTLIERtest =TRUE,DISTORTIONtest = TRUE, data =Mydata, NbDistribution = 1000, SignifThreshold = 0.05)

接下来就是异质性检验,和作者文章中结论一样,是没有异质性的。
mr_heterogeneity(Mydata, method_list=c("mr_egger_regression", "mr_ivw"))

多水平校验,这里是没有多水平效应的,和文章一致
pleio <- mr_pleiotropy_test(Mydata)
pleio

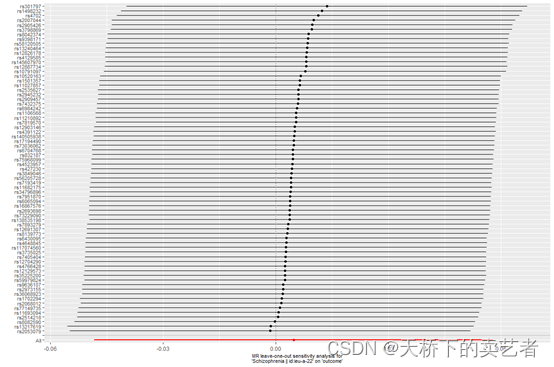
接下来是Leave-one-out analysis,Leave-one-out analysis是指逐步剔除SNP后观察剩余的稳定性,理想的是剔除后变化不大,这和我们的meta分析剔除法很相似。这张图和作者做的几乎一样。
single <- mr_leaveoneout(Mydata)
mr_leaveoneout_plot(single)
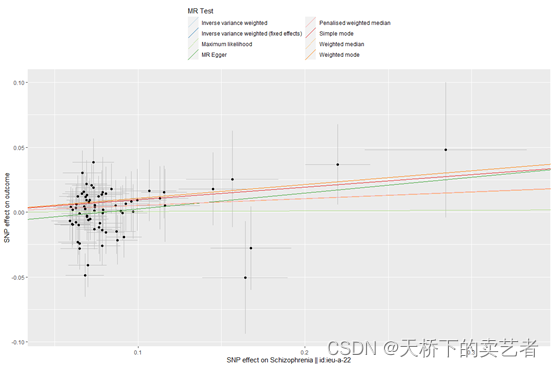
散点图
mr_scatter_plot(res,Mydata)
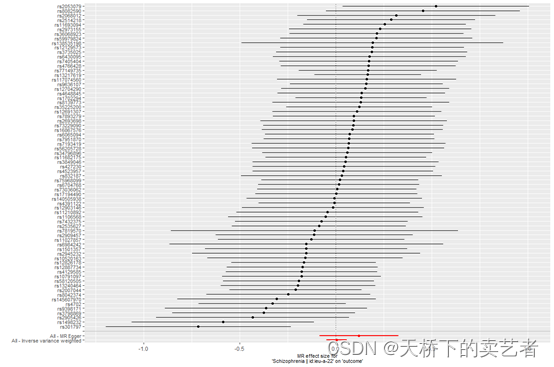
绘制森林图
res_single <- mr_singlesnp(Mydata)
mr_forest_plot(res_single)
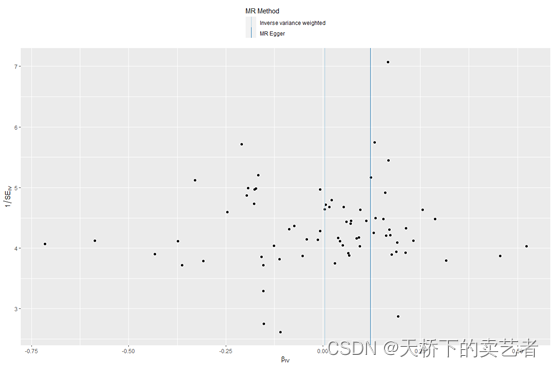
绘制漏斗图,主要是看蓝线周围的散点是否对称
mr_funnel_plot(res_single)
最后这个是生成OR和可信区间,我们可以看到虽然P值不一样,但是OR和可信区间非常接近,表明咱们的方法应该基本上是没啥问题的,毕竟数据和作者的一致,又是使用作者给的代码。
out<-generate_odds_ratios(res)
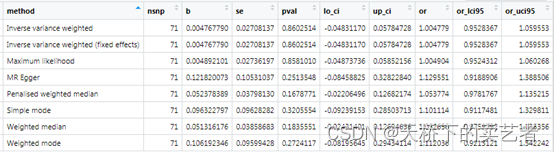

最后总结一下,咱们对作者的文章部分结果进行了复盘(自己提取数据和使用作者提供的数据),虽然数据和代码都对得上,结论也是正确的,但是OR和P值和原作者有轻微差别,我觉得本次对数据和方法还有结果是没有问题的,我大胆猜一下,有没有一种可能是作者数据太多,贴错了
到此咱们已经介绍了R包软件提取数据和手动提取数据,然后进行分析,趁着风口,大家还不自己来一篇。
欢迎斧正!