比对参考基因组
在构建文库的过程中需要将DNA片段化,因此测序得到的序列只是基因组的部分序列。为了确定测序reads在基因组上的位置,需要将reads比对回参考基因组上,这个步骤叫做比对,即文献中所提到的alignment或mapping。包括基因组比对和转录组比对目前比对的工具有很多,这里用的是hisat2。
1.HISAT2官网下载index
hisat2和其他比对软件一样,需要先建立索引,建立索引比较复杂且耗费时间,好在官网有已经建立好的索引,我们只需要到官网下载使用即可。此处下载的是人源。
wget -O hg38.tar.gz https://cloud.biohpc.swmed.edu/index.php/s/hg38/download
2.hisat2比对得到sam文件
例子:
hisat2 -t -p 8 -x ~/my_project/airway/reference/index/hg38/genome -1 ~/my_project/wangll/raw/con1_raw_1.fq.gz -2 ~/my_project/wangll/raw/con1_raw_2.fq.gz -S con1.sam
批量处理
#!/bin/bash
raw=~/my_project/wangll/raw
index=~/my_project/airway/reference/index/hg38/genome
outdir=~/my_project/wangll/alignment
ls *_raw_1.fq.gz|while read id; do id=${id/_raw_1.fq.gz/} #将字符串 id 中的_raw_1.fq.gz 部分替换为空字符串,即将_raw_1.fq.gz删除hisat2 -t -p 8 -x $index -1 $raw/${id}_raw_1.fq.gz -2 $raw/${id}_raw_2.fq.gz -S $outdir/${id}.sam;
done#挂服务器后台处理
nohup bash align.sh &
处理完之后查看nohup.out文件即可查看比对率,如下图的95.14%

3.sam文件转bam文件
sam(Sequence Alignment/Map)格式是一种通用的比对格式,用来存储reads到参考序列的比对信息。sam主要应用于测序序列mapping到基因组上的结果表示,分为注释信息(header section)和比对结果(alignment section)两部分。bam是sam的二进制文件,占用的磁盘空间比sam文本文件小;利用bam二进制文件的运算速度快。
要先安装samtools,可以用conda,如果不行的话就用wegt
mkdir ~/biosoft/samtools
cd ~/biosoft/samtools
wget -c https://github.com/samtools/samtools/releases/download/1.9/samtools-1.9.tar.bz2
#服务器上GitHub太慢了,我就在本地下载之后scp再传到服务器
tar jxvf samtools-1.9.tar.bz2 #解压
cd samtools-1.9
./configure --prefix=/home/data/t230436/biosoft/samtools/samtools-1.9
make
make install
./samtools --help #查看使用帮助,顺便确认安装成功了没有
#如果想要全局调用,可以将samtools路径放到.bashrc配置文件中
批量处理把sam文件转为bam文件
#!/bin/bash
ls *.sam|while read id;do id=${id/.sam/}samtools view -bhS -q 30 ${id}.sam > ${id}.bamsamtools sort ${id}.bam -o ${id}.sorted.bamsamtools index ${id}.sorted.bam
done
#挂服务器后台处理
nohup bash sam2bam.sh &
运行完毕之后就会得到以下文件

过程中遇到的报错:
1)

解决:加上-o参数samtools sort con1.bam -o con1.sorted.bam
2)
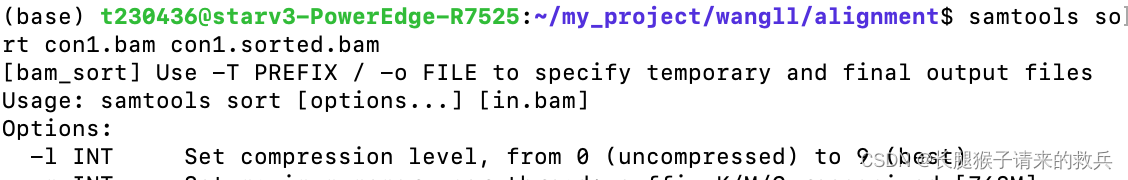
不知道为什么会莫名出错,然后我尝试了samtools sort -n con1.bam -o con1_sorted.bam可以运行,但是后面samtools index又出现了报错,搜索了一下发现命令按理说没有问题,于是退出终端之后重试了几次结果又可以运行不报错了,有点懵逼
3)samtools index: failed to create index for “con1_sorted.bam”: No such file or directory
解决:可能是因为前面sort出错(我前面用了-n参数),后面重新运行samtools sort con1.bam -o con1.sorted.bam之后再运行samtools index就没出现这个报错了
长腿猴子请来的救兵
写于2023年11月25日 马课摸鱼写的


![[BJDCTF 2020]easy_md5](https://img-blog.csdnimg.cn/5a03271839a04f769b87dc789e711e92.png)