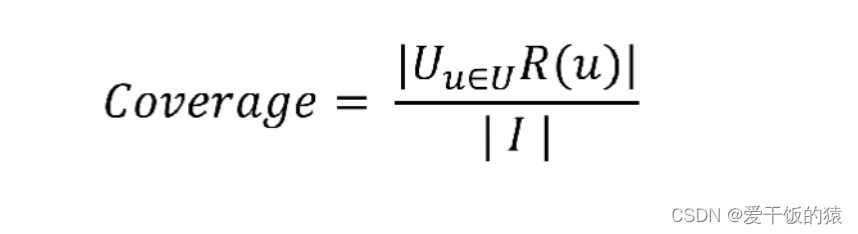在学习聚类算法的过程中,学习到的聚类算法大部分都是针对n维的,针对一维数据的聚类方式较少,今天就来学习下如何给一维的数据进行聚类。
方案一:采用K-Means对一维数据聚类
Python代码如下:
- from sklearn.cluster import KMeans
- import numpy as np
- x = np.random.random(10000)
- y = x.reshape(-1,1)
- km = KMeans()
- km.fit(y)
核心的操作是y = x.reshape(-1,1),含义为将一维数据变成只有1列,行数不知道多少(-1代表根据剩下的维度计算出数组的另外一个shape属性值)。
方案二:采用一维聚类方法Jenks Natural Breaks
Jenks Natural Breaks(自然断点分类)。一般来说,分类的原则就是差不多的放在一起,分成若干类。统计上可以用方差来衡量,通过计算每类的方差,再计算这些方差之和,用方差和的大小来比较分类的好坏。因而需要计算各种分类的方差和,其值最小的就是最优的分类结果(但并不唯一)。这也是自然断点分类法的原理。另外,当你去看数据的分布时,可以比较明显的发现断裂之处,这些断裂之处和Jenks Natural Breaks方法算出来也是一致的。因而这种分类法很“自然”。
Jenks Natural Breaks和K Means在一维数据时,完全等价。它们的目标函数一样,但是算法的步骤不完全相同。K Means是先设定好K个初始随机点。而Jenks Breaks则是用遍历的方法,一个点一个点地移动,直到达到最小值。
Natural Breaks算法又有两种:
- Jenks-Caspall algorithm(1971),是Jenks和Caspall发明的算法。原理就如前所述,实现的时候要将每种分类情况都计算一遍,找到方差和最小的那一种,计算量极大。n个数分成k类,就要从n-1个数中找k-1个组合,这个数目是很惊人的。数据量较大时,如果分类又多,以当时的计算机水平根本不能穷举各种可能性。
- Fisher-Jenks algorithm(1977),Fisher(1958)发明了一种算法提高计算效率,不需要进行穷举。Jenks将这种方法引入到数据分类中。但后来者几乎只知道Jenks而不知Fisher了。
具体算法实现:
- Jenks-Caspall algorithm:https://github.com/domlysz/Jenks-Caspall.py
- Fisher-Jenks algorithm:https://github.com/mthh/jenkspy
和K-Means一样,使用Jenks Natural Breaks需要先确定聚类数量K值。常见的方法是:GVF(The Goodness of Variance Fit)。GVF,翻译过来是“方差拟合优度”,公式如下:
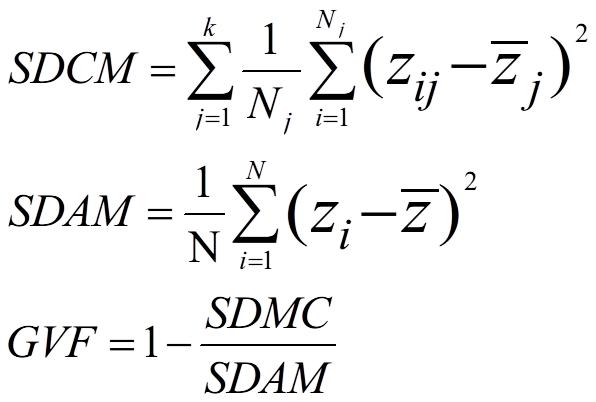
其中,SDAM是the Sum of squared Deviations from the Array Mean,即原始数据的方差;SDCM是the Sum of squared Deviations about Class Mean,即每一类方差的和。显然,SDAM是一个常数,而SDCM与分类数k有关。一定范围内,GVF越大,分类效果越好。SDCM越小,GVF越大,越接近于1。而SDCM随k的增大而大,当k等于n时,SDMC=0,GVF=1。
GVF用于判定不同分类数的分类效果好坏。以k和GVF做图可得:

随着k的增大,GVF曲线变得越来越平缓。特别是在红线处(k=5),曲线变得基本平坦(之前起伏较大,之后起伏较小),k(5)也不是很大,所以可以分为5类。一般来说,GVF>0.7就可以接受了,当然越高越好,但一定要考虑k不能太大。显然,这是一个经验公式,但总比没有好吧。
代码示例:
- from jenkspy import jenks_breaks
- import numpy as np
- def goodness_of_variance_fit(array, classes):
- # get the break points
- classes = jenks_breaks(array, classes)
- # do the actual classification
- classified = np.array([classify(i, classes) for i in array])
- # max value of zones
- maxz = max(classified)
- # nested list of zone indices
- zone_indices = [[idx for idx, val in enumerate(classified) if zone + 1 val] for zone in range(maxz)]
- # sum of squared deviations from array mean
- sdam = np.sum((array - array.mean()) 2)
- # sorted polygon stats
- array_sort = [np.array([array[index] for index in zone]) for zone in zone_indices]
- # sum of squared deviations of class means
- sdcm = sum([np.sum((classified - classified.mean()) 2) for classified in array_sort])
- # goodness of variance fit
- gvf = (sdam - sdcm) / sdam
- return gvf
- def classify(value, breaks):
- for i in range(1, len(breaks)):
- if value < breaks[i]:
- return i
- return len(breaks) - 1
- if name ‘main’:
- gvf = 0.0
- nclasses = 2
- array = np.random.random(10000)
- while gvf < .8:
- gvf = goodness_of_variance_fit(array, nclasses)
- print(nclasses, gvf)
- nclasses += 1
参考链接:
- https://en.wikipedia.org/wiki/Jenks_natural_breaks_optimization
- https://macwright.org/2013/02/18/literate-jenks.html
方案三:核密度估计Kernel Density Estimation
所谓核密度估计,就是采用平滑的峰值函数(“核”)来拟合观察到的数据点,从而对真实的概率分布曲线进行模拟。核密度估计更多详细内容,可以参考先前的Mean Shift聚类中的相关说明。
使用示例:
- import numpy as np
- from scipy.signal import argrelextrema
- import matplotlib.pyplot as plt
- from sklearn.neighbors.kde import KernelDensity
- a = np.array([10, 11, 9, 23, 21, 11, 45, 20, 11, 12]).reshape(-1, 1)
- kde = KernelDensity(kernel=‘gaussian’, bandwidth=3).fit(a)
- s = np.linspace(0, 50)
- e = kde.score_samples(s.reshape(-1, 1))
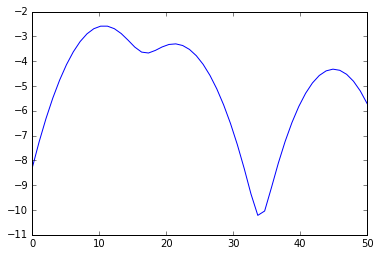
- plt.plot(s, e)
- plt.show()
- mi, ma = argrelextrema(e, np.less)[0], argrelextrema(e, np.greater)[0]
- print(“Minima:”, s[mi])
- print(“Maxima:”, s[ma])
- print(a[a < mi[0]], a[(a >= mi[0]) * (a <= mi[1])], a[a >= mi[1]])
- plt.plot(s[:mi[0] + 1], e[:mi[0] + 1], ‘r’,
- s[mi[0]:mi[1] + 1], e[mi[0]:mi[1] + 1], ‘g’,
- s[mi[1]:], e[mi[1]:], ‘b’,
- s[ma], e[ma], ‘go’,
- s[mi], e[mi], ‘ro’)
- plt.show()
输出内容:
- Minima: [17.34693878 33.67346939]
- Maxima: [10.20408163 21.42857143 44.89795918]
- [10 11 9 11 11 12] [23 21 20] [45]

参考链接:
- https://en.wikipedia.org/wiki/Kernel_density_estimation
- http://scikit-learn.org/stable/auto_examples/neighbors/plot_kde_1d.html
- https://jakevdp.github.io/blog/2013/12/01/kernel-density-estimation/
![[VSCode] Java开发环境配置](https://img-blog.csdnimg.cn/direct/a606654342694f43b7505ebf39c9eca5.png#pic_center)