Title: High-Throughput Field Plant Phenotyping: A Self-Supervised Sequential CNN Method to Segment Overlapping Plants
Abstract: High-throughput plant phenotyping—the use of imaging and remote sensing to record plant growth dynamics—is becoming more widely used. The first step in this process is typically plant segmentation, which requires a well-labeled training dataset to enable accurate segmentation of overlapping plants. However, preparing such training data is both time and labor intensive. To solve this problem, we propose a plant image processing pipeline using a self-supervised sequential convolutional neural network method for in-field phenotyping systems. This first step uses plant pixels from greenhouse images to segment nonoverlapping in-field plants in an early growth stage and then applies the segmentation results from those early-stage images as training data for the separation of plants at later growth stages. The proposed pipeline is efficient and self-supervising in the sense that no human-labeled data are needed. We then combine this approach with functional principal components analysis to reveal the relationship between the growth dynamics of plants and genotypes. We show that the proposed pipeline can accurately separate the pixels of foreground plants and estimate their heights when foreground and background plants overlap and can thus be used to efficiently assess the impact of treatments and genotypes on plant growth in a field environment by computer vision techniques. This approach should be useful for answering important scientific questions in the area of high-throughput phenotyping.
Keywords: NONE
题目:高通量田间植物表型:一种用于分割重叠植物的自监督序列CNN方法
摘要:高通量植物表型利用成像和遥感记录植物生长动态正得到越来越广泛的应用。这个过程的第一步通常是植物分割,这需要一个标记良好的训练数据集来实现重叠植物的精确分割。然而,准备这样的训练数据既费时又费力。为了解决这个问题,我们提出了一种用于现场表型系统的植物图像处理通道,该通道使用自监督序列卷积神经网络方法。第一步使用温室图像中的植物像素来分割早期生长阶段的不重叠的田间植物,然后将这些早期图像中的分割结果用作后期生长阶段植物分离的训练数据。在不需要人工标记数据的意义上,所提出的通道是有效的和自我监督的。然后,我们将这种方法与功能主成分分析相结合,以揭示植物生长动态与基因型之间的关系。我们表明,当前景植物和背景植物重叠时,所提出的通道可以准确地分离前景植物的像素并估计它们的高度,因此可以通过计算机视觉技术有效地评估处理和基因型对田间环境中植物生长的影响。这种方法应该有助于回答高通量表型领域的重要科学问题。
关键词:无
文章出处:Plant Phenomics
影响因子:6.961
作者: Xingche Guo1, Yumou Qiu1*, Dan Nettleton1, and Patrick S. Schnable2,3
作者单位:
1Department of Statistics, Iowa State University, Ames, IA, USA.
2Plant Sciences Institute, Iowa State University, Ames, IA, USA.
3Department of Agronomy, Iowa State University, Ames, IA, USA.
1.引言
高通量表型(High-throughput phenotyping)能够大规模收集温室和田地中的植物图像和传感器数据。例如,通过在每排植物前面放置相机,可以在田地里的数百到数千种作物的整个生长期同时连续地拍摄它们的侧视图像。为了利用这些丰富的数据进行植物表型的统计分析,需要随后的图像处理来提取性状。植物目标分割是植物特征提取的基本步骤。现有许多植物图像处理工具,如Scanalyzer[11]、CropSight[12]和Leaf GP[13]。这些工具通常应用简单的阈值方法[14,15]进行植物分割,该方法适用于背景均匀的温室图像。然而,对于背景复杂的图像,尤其是田间植物图像,它不能产生令人满意的植物分割结果。神经网络方法,如U-net[18]和SoySegNet[19],可以更准确地分割具有噪声背景的植物图像。然而,这些方法是基于人类标记数据的监督学习或人类标记数据和无监督数据的混合的半监督学习。准备一个足够大的训练数据集既耗时又劳动密集。郭等人[8]和Adams等人[20]提出了两步自监督的图像分割方法,其中首先通过对具有干净背景的温室图像进行K-means聚类来产生训练数据(植物像素的标签),然后基于这些自动生成的训练数据来训练神经网络模型,以对温室和田间种植的植物进行分割。他们证明了他们的方法比传统的阈值方法更准确、更稳健。
植物分割对于现场图像来说尤其具有挑战性,因为背景由泥土、设备、植物阴影等组成。图1A至D显示了爱荷华州立大学一排田间表型系统中一台相机随时间拍摄的植物照片序列。不仅有复杂的背景,而且随着植物的生长,前景行中的目标植物与背景植物重叠,因此即使用人眼也很难分离植物行。郭等人[8]提出的图像分析的K-means辅助训练(KAT4IA)程序可以产生良好的分割图像,去除大部分背景噪声(见图E至H中的分割结果)。基于分割的图像,KAT4IA可以为处于早期生长阶段的植物提供有效的高度测量,当前景行中的植物与背景行中的作物不重叠时,如图E和F所示。然而,在目标植物与背景植物重叠后,KAT4IA无法分离目标植物(例如,见图H)。

图1.对植物生长期间拍摄的图像进行分割。(A至D)爱荷华州立大学田间表型系统的一排中,一台相机随时间拍摄的植物照片序列。(E至H)使用[8]中的KAT4IA程序分割的相应图像。
尽管现有的方法可以准确地估计早期生长阶段的田间植物高度,但当植物重叠时,它们就失败了,并且不能提供完整的生长曲线估计。由于试验田的空间限制,植物行之间的距离通常需要很小;因此,在许多实验中,植物在生长过程中很快开始重叠。因此,重要的是找到一种自动机器学习方法来分离重叠的植物,而不需要人类标记的训练数据。
为了解决这个问题,我们提出了一种自监督序列卷积神经网络(SS-CNN)来分离前景植物像素和背景植物像素。我们构建了一个计算通道来提取植物高度数据,并估计每个分离植物的整个生长曲线。关键思想是在植物生长过程中以顺序的方式使用植物重叠之前的图像的分割结果作为植物重叠的图像的训练数据。我们的策略依赖于这样一种假设,即前景植物和背景植物重叠之前的前景植物的像素强度与重叠之后,但玉米生理成熟阶段之前的前景作物的像素强度足够相似,此时作物开始变黄。
具体来说,我们首先使用温室图像来训练郭等人[8]提出的田间种植植物的植物分割方法;该方法旨在分割所有植物像素,去除非植物背景噪声。然后,利用每个早季图像行中植物像素的比例,从具有不重叠植物的图像中识别前景植物和背景植物像素。通过这种方式,可以自动有效地构建关于植物像素的自监督训练数据,并将其用于生长后期重叠植物的分离。这种自我监督的方法避免了准备训练数据的昂贵的手动标记过程。
区分前景植物和背景植物像素比区分植物和背景像素更具挑战性。因此,需要每个像素的邻域信息来获得良好的分类结果。我们基于矩形邻域的像素强度构建了一个卷积神经网络(CNN)模型,该模型利用植物的几何结构来更好地分离前景植物和背景植物像素。然后可以基于分割的前景植物图像来测量植物高度。通过将所提出的方法的生长后期高度测量值与KAT4IA程序计算的生长早期高度测量值相结合,可以获得植物在整个生长期的完整生长曲线。所提取的生长曲线可以用于随后的生物学分析。在结果中,我们介绍了功能主成分分析(FPCA)[21,22]的结果,以研究和比较基于提取高度的不同基因型的生长动态,揭示了基因型对植物生长模式的影响。
2.方法
我们的主要目标是开发一种通道,自动将前景植物像素与背景植物像素分离,如图1所示。并从照片序列中提取所有前景植物的高度,以估计植物生长曲线。主要步骤如下。每个步骤的详细程序将在后面的小节中进行解释。
1.从背景分割:KAT4IA算法[8]从相机随时间拍摄的田地图像序列开始执行,以获得植物的分割图像。通过将所有分类的非植物像素的RGB强度替换为0来去除图像中的背景。这将保持植物像素的原始RGB强度,并将所有背景像素着色为黑色;产生的图像被称为“背景去除图像”。当前景植物开始与背景植物重叠时,就确定了时间点。
2.植物分离训练数据的自动构建:在植物重叠之前,在去除背景的图像中分离前景和背景植物。通过分别用1和0标记所有前景和背景植物像素来创建训练数据。对于每个标记的像素,像素强度被包括在其邻域中,大小为(2r+1)×(2c+1)作为该像素的相关特征,其中r和c是以标记像素为中心的邻域矩形的半宽半高。
3.CNN学习:使用从第二步获得的训练数据对CNN进行训练,以分离后期图像中的前景和背景植物,其中前景植物和背景植物重叠。
4.超像素分类结果的后处理:使用Achanta等人提出的简单线性迭代聚类(SLIC)超像素,从第三步细化前景和背景植物像素的分类结果[23]。为每个超级像素中的所有像素分配了一个公共标签。
5.高度测量:根据生长季节的一系列图像计算每个前景植物的植物高度。使用非参数回归估计植物生长曲线,平均值不降低[24,25]。
所有步骤均由R语言和R中的API Keras实现。训练模型建立在单个个人计算机上,基于训练模型的野外图像分割是在并行计算的高性能计算集群中进行的。
3.图像数据和背景植物分割
在这个项目中,我们使用了爱荷华州州立大学植物科学研究所的研究人员2017年在内布拉斯加州格兰特附近的一块雨养(即无灌溉)田地中拍摄的GPS坐标(40.94,−101.77)的田地图像。该地有2个重复种植区,分别有103个和101个玉米基因型。每一次复制中的每一行最多包括6个单一基因型的植物,每行植物前面安装一个摄像头。行长、行间距和行内株间距分别为182.9、304.8和36.6cm。我们在每排植物前面使用了NIKON COOLPIX S3700相机,焦距为4.5毫米,传感器尺寸为6.17毫米×4.55毫米。相机到行的距离为213.4厘米,相机的高度约为120至150厘米,安装在每根杆子的顶部。两次复制中,每台相机拍摄的平均照片数量分别为1719张和1650张,白天每15分钟拍摄一次。我们使用6月中旬至8月初拍摄的照片。现场照片是RGB图像,每个像素的红色、绿色和蓝色通道的强度值在0到255之间。像素强度通过将其除以255来归一化,从而产生0和1之间的浮点数。我们将图像分辨率从5152×3864重新缩放到1000×750,以提高计算效率。
在我们的训练通道中,我们首先应用郭等人[8]提出的KAT4IA程序来获得田间图像的植物分割图像(图1)。KAT4IA使用温室植物图像来训练田间分割模型(更多详细信息,请参见[8])。使用分割结果,我们通过用零值替换所有非植物像素的RGB强度来从图像中去除背景(图3B)。这导致了“背景去除图像”。对于每一排植物,我们使用以下方法自动检测前景和背景植物重叠的时间点。首先,计算每个图像行中植物像素的比例。然后,行比例中的第一峰值(从图像的底部到顶部)通过选择每个峰值的上边界和下边界作为小于峰值中心以上和以下的峰值最大值的小百分比(例如5%)的第一像素行来识别。行比例的第一个峰值的边界识别具有前景植物的图像行。上面提到的行切割算法有助于定位前景植物(见图2B)。然后,将变化点检测方法应用于随时间变化的图像序列中识别的第一个峰值的宽度,以估计田地中每行的前景植物和背景植物重叠的时间(图2A)。
采用这种方法,当植物开始重叠时,第一个峰值的宽度将急剧增加到更大的值。当前景植物和背景植物很好地分离时,行切割算法可以准确地识别生长早期前景植物的图像行。然而,一旦前景植物开始与背景植物重叠,行切割算法倾向于分割包含前景和背景植物的图像行,这导致分割行的宽度跳跃(见图2C中的示例)。此跳跃对应于前景植物开始与背景中的植物重叠的日期。图1C提供了一个示例。
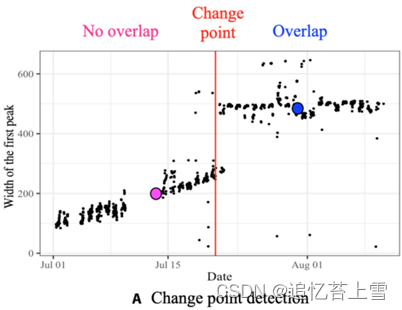
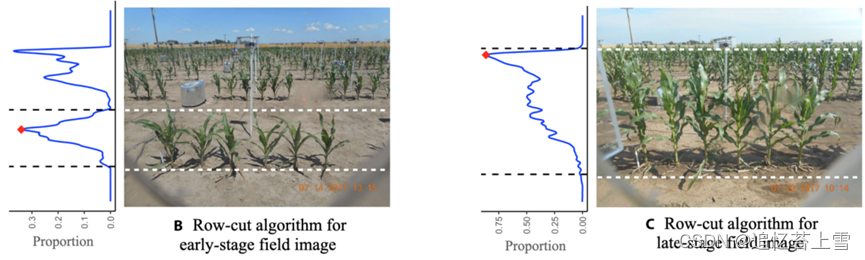
图2.行切割算法如何分离前景植物的示例。
(A)图像序列中识别的第一个峰值的宽度随时间的散点图。红线是前景植物和背景植物开始重叠的时间点(“变化点”)。
(B)用于早期生长阶段场图像的行切割算法的结果;(A)中的粉红色点表示(B)中的第一峰值的估计宽度。分割图像的行比例曲线显示在场图像的左侧。2条虚线是从底部开始的第一个峰的上边界和下边界。
(C)生长后期场图像的行切割算法的结果;(A)中的蓝点表示(C)中的第一峰值的估计宽度。

图3.用于构建训练数据集的工作流。
(A)早期生长阶段现场图像示例。
将KAT4IA算法应用于(A)给出了“背景去除图像”(B)。
使用行切割算法分离前景和背景植物,得到“标记的分割图像”(C)。
(D和E)使用蓝色矩形从(B)中标记的3个示例背景植物像素和使用粉色矩形在(B)中标记的3个实例前景植物像素裁剪的迷你图像。
4.训练数据的自动构建
为了分离重叠的前景和背景植物,需要大量的训练数据来构建机器学习算法。然而,获得这样的训练数据通常需要对每个实验的大图像集中的每个植物像素进行手动注释和标记。由于图像的高分辨率和植物的不规则形状,这种标记过程既耗时又劳动密集。我们通过使用生长早期非重叠植物的植物像素来构建自我监督训练数据,以分离生长后期重叠的前景和背景植物,从而应对这一挑战。具体而言,对于田间图像中的早期生长阶段,可以使用前一步骤中的行切割算法简单地分离前景和背景植物,因为如果前景和背景作物不重叠,则在分割图像的行比例的曲线中存在尖锐的波谷。因此,我们可以让算法简单地将截止线上方的所有植物像素定义为背景植物像素(0),将截止线下方的所有植物象素定义为前景植物像素(1)(见图3C中的示例)。
此外,为了构建生长后期植物的更具代表性的训练数据,我们使用了重叠开始前的图像。例如,图1C比图1A和1B更适合构建训练数据,因为图1C中的植物结构与生长后期的植物具有更相似的特征。由于邻域像素包含关于植物的几何信息,它们可以帮助区分目标植物。对于前景和背景植物的每个标记像素,以邻域(2r+1)×(2c+1)为中心的裁剪迷你图像被用作输入特征,其中r和c是以标记像素为中心的邻域矩形的半宽半高。直观的假设是,同一类别中的裁剪迷你图像更有可能共享相似的特征。这类似于经典的CNN方法,其中使用邻域信息的卷积来预测响应类别。构建训练数据集的工作流程如图3所示。背景和前景植物特征的示例分别如图D和E所示。
5.利用CNN分离前景和背景植物像素
接下来,我们使用植物重叠前图像生成的训练数据训练CNN,使用R中的API Keras来分离重叠的前景和背景植物。对于训练数据中的每个标记像素,其(2r+1)×(2c+1)邻域中像素的RGB(红、绿和蓝)强度被用作输入特征,其中r=c=16。这导致每个训练像素的特征空间的尺寸为33×33×3,其中3是红色、绿色和蓝色强度的通道数量,33×33是每个通道的分辨率。我们还尝试了r=c=8、r=c=12和r=c=20的CNN模型。从训练结果中,我们发现验证精度随着r和c的增加而增加,并且在r和c达到16后变得稳定。因此,我们选择邻域大小为r=c=16,因为较小的邻域大小导致较少的训练时间。具体而言,在CNN模型中,有4个卷积层,其大小如下:(33,33,32)、(33,32,32)和(16,16,64),和(16,16,64)。其中第一个是输入层,其中3是通道的数量,33×33是每个通道的分辨率。使用具有相同填充和ReLU激活函数的A 3×3滤波器核来计算卷积层。在第二和第三卷积层之间应用没有填充的A 2×2最大池化。在最后一个卷积层之后应用另一个2×2最大池化,这导致大小为(8,8,64)的最大池化层。最后,使用具有一个隐藏层的多层感知器(MLP)来计算属于前景植物类的特定像素的预测概率。最大池化层的平坦化给出了MLP的输入层,该层具有4096个节点。隐藏层有128个神经元。MLP的输入层和隐藏层之间以及隐藏层和输出层之间的丢失率被设置为0.3。ReLU激活函数用于MLP的输入层和隐藏层之间。sigmoid激活函数用于基于MLP的隐藏层来预测每个像素的前景概率。使用具有Adam优化算法和0.001的学习率的二进制交叉熵损失函数来评估网络模型。最后,将批量大小为1000的100个时期用于训练,并保留5%的训练数据作为验证集。最后50个时期的平均训练和验证准确率分别为97.7%和94.3%,表明植物CNN具有良好的拟合结果。图4显示了所提出的CNN的结构。训练CNN对生长后期具有重叠植物的图像的前景和背景植物像素进行分类,并计算每个植物像素属于前景植物类的概率。图5E显示了示例图像的预测概率。
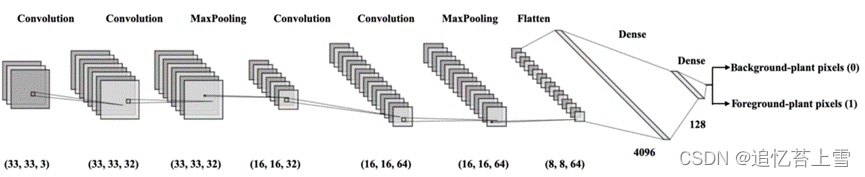
图4.CNN的结构,用于对生长后期具有重叠植物的图像的前景和背景植物像素进行分类。
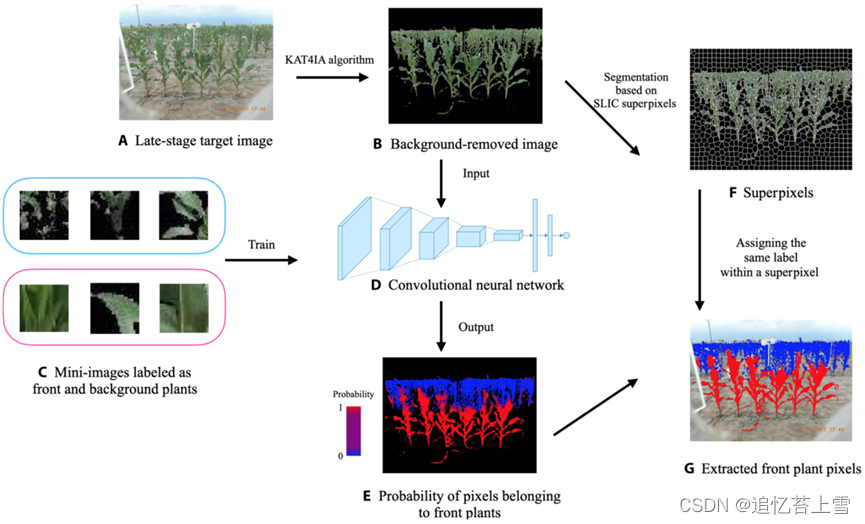
图5. 植物像素分离和后处理工作流程。(A)目标生长后期场图像。
目标是从图像中提取前景植物。将KAT4IA算法应用于(A)给出了“背景去除图像”(B)。
(B)的超像素如(F)所示。
收集与2个植物类别相关的修剪的早期生长阶段迷你图像作为训练数据(C),以训练CNN(D)
对于(B)中的每个植物像素,裁剪邻域迷你图像,然后将(D)应用于每个迷你图像给出每个像素属于前景植物类的概率。
概率在(E)中可视化,其中红色表示分配了1,蓝色表示分配了0。
通过首先对每个超像素内的预测概率进行平均,然后使用截止阈值对前景植物和背景植物超像素进行分类来获得最终图像(G)。
6.使用超像素对CNN结果进行后处理
所提出的自监督方法基于使用给定截止值从CNN模型生成的概率对去除背景的图像中的每个植物像素进行分类。如图5E所示,我们训练的CNN相当好地将前景植物与背景植物分离。然而,仍然存在一些分类错误:图E中的图像包括蓝色点(已识别的背景像素)中的几个红色点(已标识的前景像素)和被红色点包围的几个蓝色点。请注意,CNN分别预测每个像素的类标签。这可能导致目标植物的非光滑分割结果。因此,我们认为,通过利用图像几何信息,例如周围像素的颜色和像素的空间接近度,应该可以细化CNN的分类结果。
为此,我们在通道中引入了一个过程,在该过程中,在去除背景的图像上形成SLIC超像素[23],以基于像素坐标和RGB强度将像素分组为具有感知意义的原子区域(例如,图5F)。超像素可以被解释为共享相似信息的每个图像的几何微聚类。为了从相邻像素借用信息,计算每个超像素内前景类的平均概率。然后使用0.5的截止阈值来在超像素级别对前景和背景植物进行分类。如果平均概率分别大于或小于0.5,则超像素内的所有像素被预测为前景类或背景类。这样,同一超级像素中的所有像素都被分类到一个公共类中。为了加速计算,基于来自该超像素的Niα 个像素的随机样本来估计第i个超像素中前景类的平均概率,其中Ni 是第i个超级像素中的像素总数。图5总结了使用自监督CNN进行植物像素分离和使用超像素进行后处理的工作流程,超像素可以高精度识别前景植物。
7. 株高测量和生长曲线拟合
对于生长早期的图像,在植物重叠之前,可以使用行切割算法轻松地分离前景植物(图2)。然后使用类似于行切割算法的列切割算法来分离单独的前景植物。然后,基于分割图像中的顶部和底部植物像素来测量每个植物的高度。KAT4IA通道[8]详细说明了早期植物的这一过程。
对于生长后期的图像,一旦识别并分离了前景植物,就可以使用类似的高度测量算法。首先,创建一个二值图像,其中前景植物像素被标记为1,所有其他像素都被标记为0。然后计算行平均值,给出图像每行中前景植物像素的百分比。其次,使用R中的黄土函数,使用局部平滑器对行平均曲线进行平滑。计算行平均值的最大值,并将行平均值曲线中峰值的上边界和下边界识别为平均值分别小于峰值最大值的7.5%和2.5%的第一行。这标识了图像中与前景植物相对应的行。第三,将KAT4IA管道中使用的相同柱切割和高度测量算法应用于分离单个前景植物并测量它们的高度。图6A、C和E显示了前景植物分离的示例,图B、D和F显示了这些图像中每个目标植物的高度测量结果。所提出的自监督CNN算法能够以较低的假阳性率恢复前景植物的大部分,并且高度测量算法基于前景分离结果对每个目标植物的高度提供了合理的估计。



图6.识别前景植物,并确定它们的高度。(A、C和E)来自一台摄像机的3幅示例图像的前景和背景植物识别结果。(B、D和F)相应的高度测量结果。
使用从生长季节的所有植物图像中提取的植物高度,可以使用具有非递减趋势的非参数回归拟合田地每行中每棵植物的生长曲线[24,25]。图6所示前景植物的拟合生长曲线如图7所示。粉红色点和青蓝色点是生长早期和晚期图像的高度测量值,分别对应于不重叠的植物和重叠的植物。测量的高度也如图7所示,颜色不同。
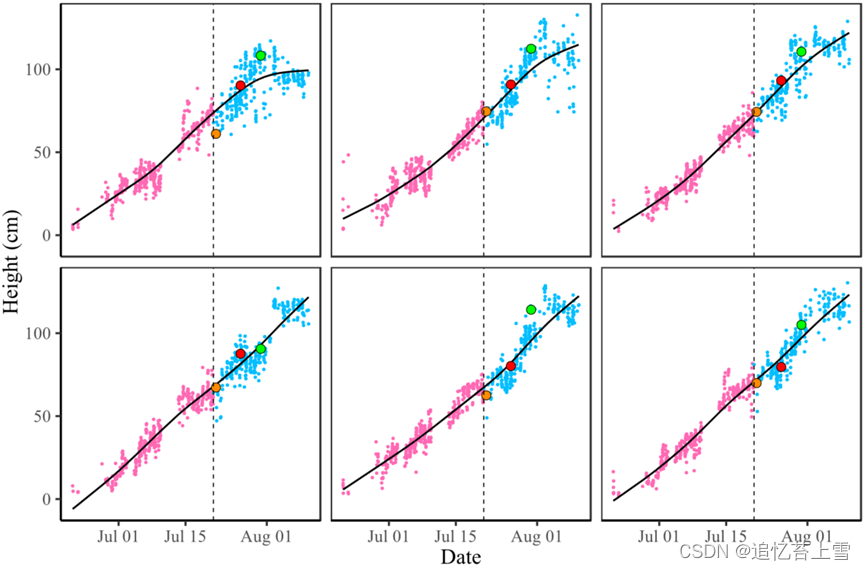
图7. 生长曲线被拟合到来自一台相机的一组图像中每株植物的高度测量值。
粉红色点是使用KAT4IA管道从早期生长阶段图像中提取的植物高度。
青蓝色点是使用我们提出的方法从生长后期图像中提取的植物高度。
非递减拟合增长曲线以黑线显示。
图6中3幅图像的测量高度用不同的颜色突出显示。橙色圆点表示图B的高度,红色圆点表示图D的高度,绿色圆点表示图F的高度。
8. 结果
在本节中,我们详细介绍了将我们的SS-CNN通道应用于2017年干田数据的首次重现(replication)的结果。具体来说,我们计算了第一次重现中103个基因型中每一个基因型在植物生长季节的平均植物高度,并提供了每个基因型的生长曲线估计。增长曲线估计如图8A所示,其中红色曲线表示增长曲线估计的平均值。值得注意的是,干旱地区的大多数植物在2017年8月初的一场风暴中受损。因此,我们决定使用8月之前的野外图像来拟合生长曲线。了研究基因型对植物生长模式的影响,我们将FPCA[21]应用于103生长曲线估计。根据Karhunen–Loève定理,第i个基因型(Yi(t),i=1,…,103)的生长曲线可以很好地近似为:
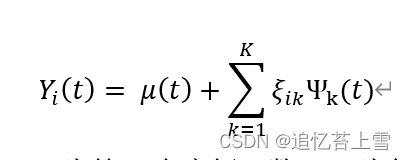
其中μt 是均值函数,Ψk(t) 为第k个本征函数,ξik 为第i个基因型的第k个函数主成分(FPC)得分。前两个FPC解释了增长曲线估计总方差的95%以上。请注意,我们只在8月之前测量了植物的高度,当时大多数植物仍在生长。因此,增长曲线并没有变平。为了拟合最终变平的完整增长曲线,我们可以通过包含2个以上的FPC来增加模型的复杂性。
前2个本征函数如图8B所示。第一个本征函控制着总体增长率;对应于第一本征函数的正FPC分数导致相对高于平均值的增长率。同时,第二个本征函数控制着增长率随时间的变化;对应于第二本征函数的正FPC得分意味着生长速率在植物生长季节增加。
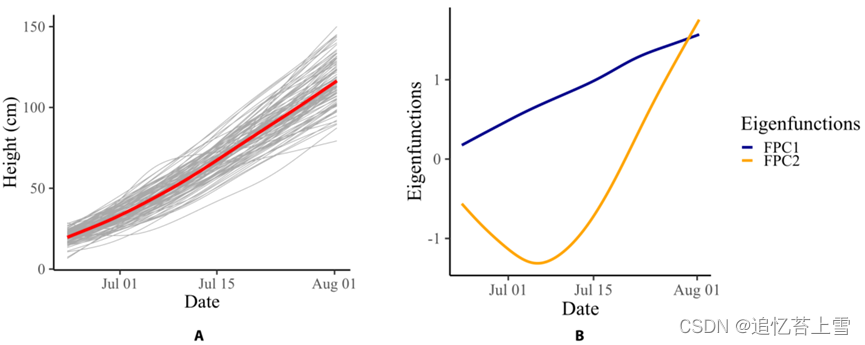
图8.估算了103个基因型的生长曲线。(A)103种基因型的生长曲线估计;红色曲线表示增长曲线估计值的平均值。(B)前2个估计的本征函数。
图9提供了更清晰的解释。图9A显示了每个基因型的第二次FPC评分与第一次FPC得分的散点图。PHB47×PHW30和B73×PHN82基因型具有相似的第二FPC评分;然而,PHB47×PHW30的第一次FPC评分为阳性,而B73×PHN82的第一次FP评分为阴性。因此,PHB47×PHW30的总体增长率高于B73×PHN82(图B)。基因型PHB47×LH123HT和LH198×PHN82的首次FPC得分均接近0;然而,PHB47×LH123HT的第二FPC得分>0,而LH198×PHN82的第二FPC得分<0。因此,两种基因型的总体生长速率都接近平均生长速率,但PHB47×LH123HT在生长早期的生长速率较低,在生长后期的生长速率较高,而LH198×PHN82的生长速率随着时间的推移而下降。
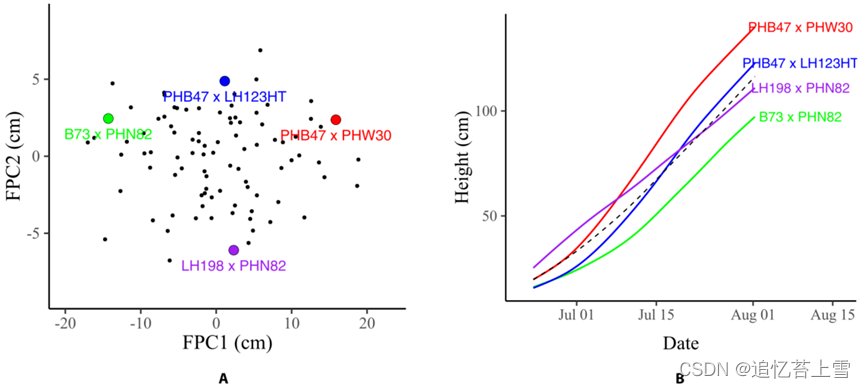
图9.增长曲线对应于一对FPC分数。(A)每个基因型的第二个FPC分数与第一个FPC得分的散点图。(B)4种基因型的生长曲线估计。每个生长曲线对应于在(a)中使用相同颜色标记的一对FPC分数。黑色虚线曲线是估计的平均增长曲线。
9.讨论
处理图像数据和提取植物特征是当前表型研究和应用的主要问题之一。尽管在一些图像中,将目标植物与背景分离的人工注释是可能的,但这种标记过程通常是乏味和耗时的。所提出的自我监督方法使用计算机自动准备训练数据。这允许机器根据在适应光照条件和环境变化的田地中拍摄的图像有效地测量植物特征。我们的结果表明,所提出的程序可以对植物高度进行准确可靠的测量,下面的功能数据分析能够揭示基因型对植物生长曲线的影响。未来,我们将在田间成像机器人上部署所提出的分析管道,使机器人能够在田间工作时自动提取植物特征并实时进行统计分析。在所提出的通道中,我们使用序列图像来准备重叠植物的训练数据。分割结果依赖于重叠前的早期植物和重叠后的晚期植物之间的相似性。如果来自这两个阶段的图像的植物像素的颜色非常不同,则分割结果将不好。幸运的是,在我们的图像数据分析示例中并非如此。自动创建训练数据的另一种方法是使用来自具有干净背景的温室图像的分割植物,并将它们覆盖在具有田地背景的图像上。这可以为前景植物的像素创建具有已知位置的合成田间植物图像。然后,可以构建与所提出的管道中使用的CNN类似的CNN或U-net模型[18],用于植物分割。
从图像中提取的高度的验证是一个重要的问题。然而,我们还没有从这个实验中手动测量植物的高度。因此,我们无法将提取的高度与地面实况进行比较。然而,如果植物分割是准确的,则从图像中提取的高度应该是准确的。通过比较分割图像和原始图像,可以直观地检查所提出的分割过程的有效性,如图6所示。在未来的实验中,我们将收集人工测量的植物高度,作为所提出程序的验证数据集。
10.数据可用性
拟议通道的R代码、样本图像数据(包括6张用于训练的现场照片和52张由我们的一台相机拍摄的用于测试的现场照片示例)和描述可在Github上获得,网址为GitHub - xingcheg/Plant-Traits-Extraction-2023。