欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://spike.blog.csdn.net/article/details/135568438
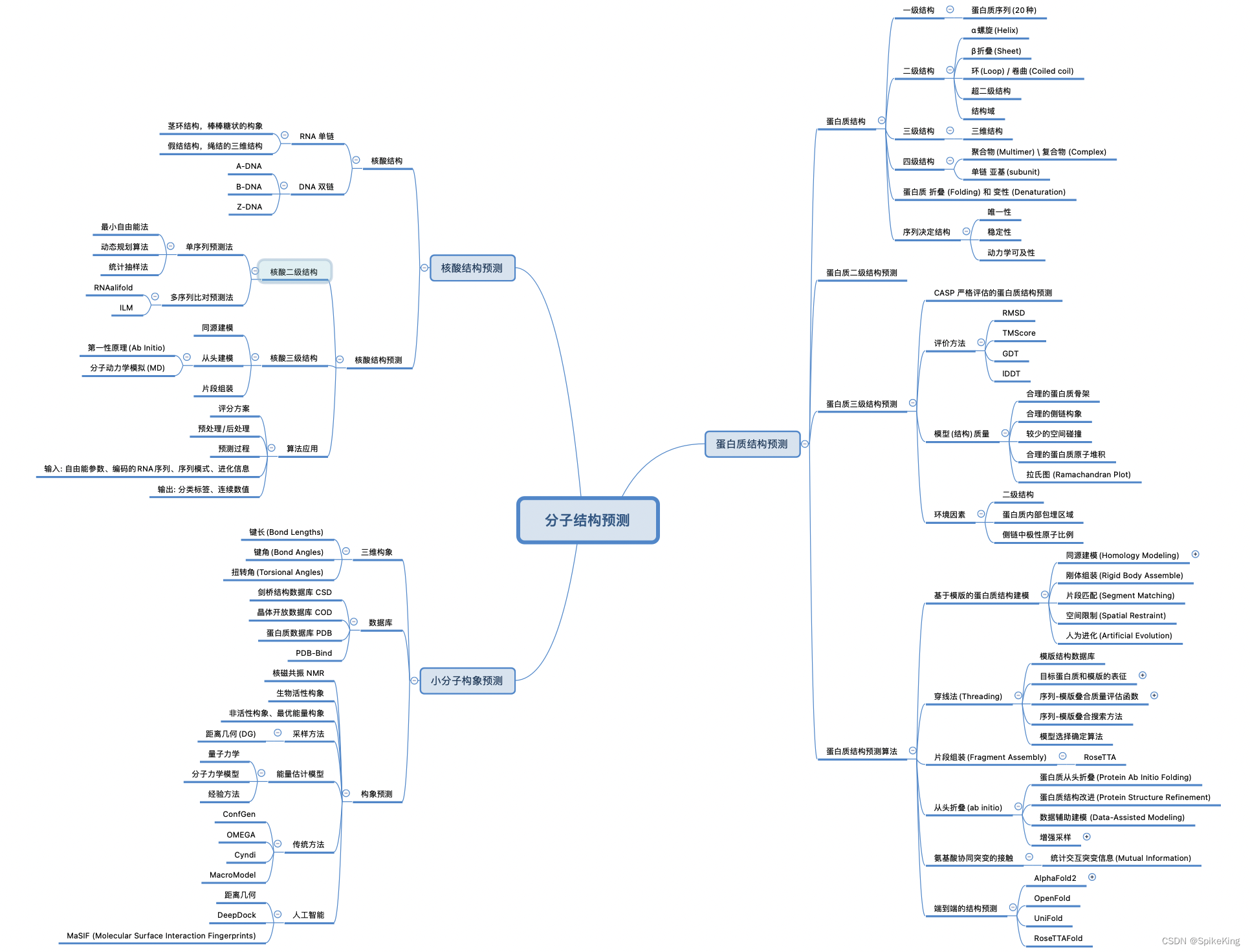
生物结构预测是指根据生物分子的序列信息,推断其在空间中的三维形状和排列。生物结构预测对于理解生物分子的功能、相互作用、进化和设计具有重要意义。生物结构预测的对象主要包括蛋白质、核酸和小分子。
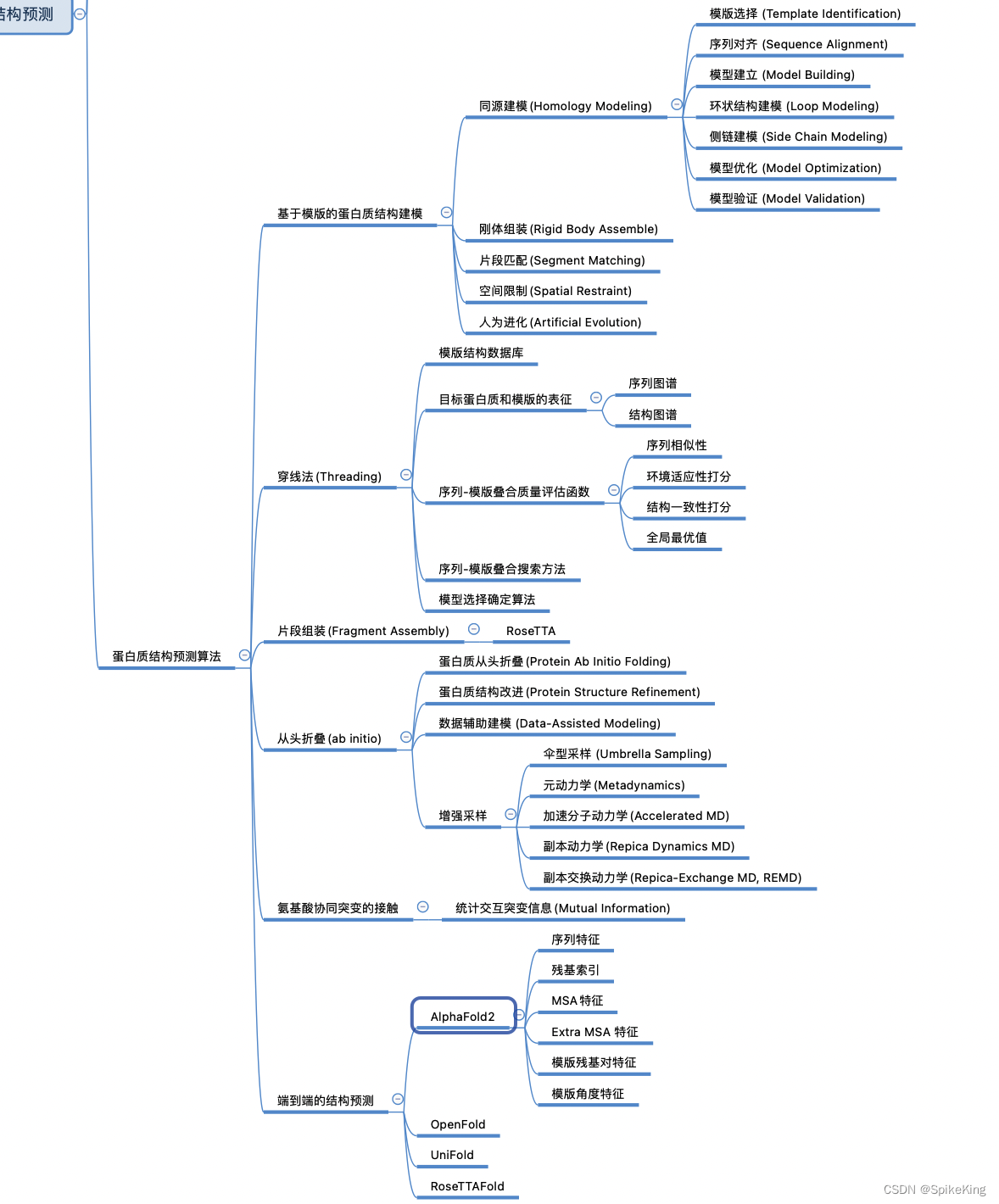
- 蛋白质结构预测是指根据蛋白质的氨基酸序列,预测其二级结构、三级结构和四级结构。蛋白质结构预测的方法主要有两类:同源建模和折叠识别。同源建模是指利用已知结构的蛋白质作为模板,根据序列相似性和结构保守性,构建目标蛋白质的结构。折叠识别是指利用已知的蛋白质折叠类型,根据序列和结构的特征,识别目标蛋白质的折叠类别,并生成其结构。一些常用的蛋白质结构预测的在线工具有SWISS-MODEL、PSIPRED和Phyre2等。
- 核酸结构预测是指根据核酸的碱基序列,预测其二级结构和三级结构。核酸结构预测的方法主要有两类:热力学方法和统计方法。热力学方法是指利用核酸的自由能模型,计算不同结构的稳定性,选择最稳定的结构作为预测结果。统计方法是指利用核酸的序列和结构的统计规律,计算不同结构的概率,选择最可能的结构作为预测结果。一些常用的核酸结构预测的在线工具有RNAfold、Mfold和InterPro等。
- 小分子构象预测是指根据小分子的化学结构,预测其在空间中的可能的形状和能量。小分子构象预测的方法主要有两类:确定性方法和随机方法。确定性方法是指利用小分子的键长、键角和扭转角等几何参数,系统地搜索所有可能的构象,选择最低能的构象作为预测结果。随机方法是指利用小分子的势能函数,随机地生成和优化构象,选择满足一定条件的构象作为预测结果。一些常用的小分子构象预测的在线工具有TorsionNet、DGSM和CONFGF等。
蛋白质结构预测算法: