全基因组关联(GWA)研究扫描整个物种基因组,寻找多达数百万个SNPs与特定感兴趣特征之间的关联。值得注意的是,感兴趣的性状实际上可以是归因于群体的任何类型的表型,无论是定性的(例如疾病状态)还是定量的(例如身高)。本质上,给定p个SNP和n个样本或个体,GWA分析将拟合p个独立的单变量线性模型,每个模型基于n个样本,使用每个SNP的基因型作为感兴趣特征的预测因子。每个P检验中的关联显著性(P值)由相应SNP的系数估计确定(从技术上讲,关联的显著性为)。请注意,由于这些测试是独立的且数量众多,因此在设置并行GWA分析时有很大的计算优势(我们将很快进行)。非常合理的是,有必要使用多种假设检验方法,如Bonferroni、Benjamini Hochberg或错误发现率(FDR)来调整所得的P值。GWA研究现在在许多不同物种的遗传学中很常见。
读取数据
让我们首先从三个民族中的每一个导入PLINK转换的.ded、.fam和.bim Illumina文件。我们将使用snpStats包中的函数read.plink,并在本教程的其余部分中处理生成的对象。此函数读取.bed、.fam和.bim,并创建一个包含三个元素的列表——$genesis、$fam和$map。第一个包含从所有样本中确定的所有SNP,第二个包含关于谱系和性别的信息,第三个包含SNP的基因组坐标。目前,我们共有323人(110名中国人、105名印度人和108名马来人)和2527458个SNPs。接下来,我们将把$genetype中存储的Illumina SNP ID更改为更传统的rs ID,这将使我们能够在USCS基因组浏览器中放大候选SNP周围的基因组区域。我准备了一个表来建立两者之间的对应关系,这样我们就可以轻松地切换ID。
library(snpStats)
load("conversionTable.RData")pathM <- paste("Genomics/108Malay_2527458snps", c(".bed", ".bim", ".fam"), sep = "")
SNP_M <- read.plink(pathM[1], pathM[2], pathM[3])pathI <- paste("Genomics/105Indian_2527458snps", c(".bed", ".bim", ".fam"), sep = "")
SNP_I <- read.plink(pathI[1], pathI[2], pathI[3])pathC <- paste("Genomics/110Chinese_2527458snps", c(".bed", ".bim", ".fam"), sep = "")
SNP_C <- read.plink(pathC[1], pathC[2], pathC[3])# Merge the three SNP datasets
SNP <- rbind(SNP_M$genotypes, SNP_I$genotypes, SNP_C$genotypes)# Take one bim map (all 3 maps are based on the same ordered set of SNPs)
map <- SNP_M$map
colnames(map) <- c("chr", "SNP", "gen.dist", "position", "A1", "A2")# Rename SNPs present in the conversion table into rs IDs
mappedSNPs <- intersect(map$SNP, names(conversionTable))
newIDs <- conversionTable[match(map$SNP[map$SNP %in% mappedSNPs], names(conversionTable))]
map$SNP[rownames(map) %in% mappedSNPs] <- newIDs接下来,我们导入并合并三个脂质数据集(存储为.txt),并确定SNP和脂质数据集中都存在哪些样本。在以下分析中,我们将使用两个平台中介绍的样本子集,共计319个。最后,我们创建一个列表genData,存储合并的SNP数据($SNP),这是三个$map中的一个,因为它们都是相同的($map)和合并的脂质数据($LIP)。最后,让我们为后续步骤保存RAM,并在将genData保存到之后删除所有文件。RData文件。
# Load lipid datasets & match SNP-Lipidomics samples
lipidsMalay <- read.delim("Lipidomic/117Malay_282lipids.txt", row.names = 1)
lipidsIndian <- read.delim("Lipidomic/120Indian_282lipids.txt", row.names = 1)
lipidsChinese <- read.delim("Lipidomic/122Chinese_282lipids.txt", row.names = 1)
all(Reduce(intersect, list(colnames(lipidsMalay),
colnames(lipidsIndian),
colnames(lipidsChinese))) == colnames(lipidsMalay)) # TRUE
lip <- rbind(lipidsMalay, lipidsIndian, lipidsChinese)matchingSamples <- intersect(rownames(lip), rownames(SNP))
SNP <- SNP[matchingSamples,]
lip <- lip[matchingSamples,]genData <- list(SNP = SNP, MAP = map, LIP = lip)
save(genData, file = "PhenoGenoMap.RData")# Clear memory
rm(list = ls())预处理
让我们重新加载genData并对其进行清理。简而言之,数据的预处理包括
丢弃呼叫率<1或MAF<0.1的SNPs丢弃呼叫率<100%、IBD亲缘系数>0.1或近交系数|F|>0.1的样本呼叫率是进行基因分型的SNPs(或样本)的比例。例如,特定SNP(样本)的呼叫率为0.95意味着丢失了5%的值。因为我们有这么多的SNP,我们可以通过对SNP和样本施加1的呼叫率阈值,在$SNP矩阵中绝对没有遗漏值。如果你想放宽门槛并容忍缺失的值,请记住,你需要运行一个完整的程序来对这些值进行指责。Reed等人,2015描述了一种基于PCA的插补方法,该方法利用1000基因组计划作为代理,以防您感兴趣。
次要等位基因频率(MAF)表示每个SNP的最不常见等位基因的比例。当然,很难检测出与罕见变异的关联,这就是为什么我们选择低MAF值。我读过的大多数GWA研究通常报告MAF阈值为0.05。在这里,我选择了更严格的0.1,因为我们有大量的数据,而且由于所有这些都是为了说明目的,我们希望进行快速的GWA分析。
library(snpStats)
library(doParallel)
library(SNPRelate)
library(GenABEL)
library(dplyr)
source("GWASfunction.R")
load("PhenoGenoMap.RData")# Use SNP call rate of 100%, MAF of 0.1 (very stringent)
maf <- 0.1
callRate <- 1
SNPstats <- col.summary(genData$SNP)maf_call <- with(SNPstats, MAF > maf & Call.rate == callRate)
genData$SNP <- genData$SNP[,maf_call]
genData$MAP <- genData$MAP[maf_call,]
SNPstats <- SNPstats[maf_call,]接下来,我们需要考虑表现出过度杂合性的样本——从技术上讲,偏离Hardy-Weinberg平衡(HWE),
# Sample call rate & heterozygosity
callMat <- !is.na(genData$SNP)
Sampstats <- row.summary(genData$SNP)
hetExp <- callMat %*% (2 * SNPstats$MAF * (1 - SNPstats$MAF)) # Hardy-Weinberg heterozygosity (expected)
hetObs <- with(Sampstats, Heterozygosity * (ncol(genData$SNP)) * Call.rate)
Sampstats$hetF <- 1-(hetObs/hetExp)
# Use sample call rate of 100%, het threshold of 0.1 (very stringent)
het <- 0.1 # Set cutoff for inbreeding coefficient;
het_call <- with(Sampstats, abs(hetF) < het & Call.rate == 1)
genData$SNP <- genData$SNP[het_call,]
genData$LIP <- genData$LIP[het_call,]最后,我们将使用基于血统认同的亲属系数(IBD)来研究样本之间的相关性。请注意,SNPRelate包中的这些函数需要GDS文件。出于这个原因,我们首先需要将来自三个群体的.bed、.fam和.bim文件聚合到convertGDS中。函数snpgdsBED2GDS2创建这部分分析所需的GDS。为了确定样本对之间的亲缘关系系数,我们将使用不相关SNPs的子集,以便进行无偏估计。为此,我们将使用连锁不平衡(LD)作为标记之间相关性的衡量标准。LD的范围从0到1,其值越高,两个SNP就越有可能共同分离并因此相关。在这里,我们将利用LD<0.2(p~12000)的SNPs子集来确定IBD亲缘系数。我用函数snpgdsLDpruning计算LD大约花了2个小时,所以要耐心。接下来,根据Reed等人,2015,我们将排除所有亲缘系数>0.1的样本。
# LD and kinship coeff
ld <- .2
kin <- .1
snpgdsBED2GDS(bed.fn = "convertGDS.bed", bim.fn = "convertGDS.bim",
fam.fn = "convertGDS.fam", out.gdsfn = "myGDS", cvt.chr = "char")
genofile <- snpgdsOpen("myGDS", readonly = F)
gds.ids <- read.gdsn(index.gdsn(genofile, "sample.id"))
gds.ids <- sub("-1", "", gds.ids)
add.gdsn(genofile, "sample.id", gds.ids, replace = T)
geno.sample.ids <- rownames(genData$SNP)
# First filter for LD
snpSUB <- snpgdsLDpruning(genofile, ld.threshold = ld,
sample.id = geno.sample.ids,
snp.id = colnames(genData$SNP))
snpset.ibd <- unlist(snpSUB, use.names = F)
# And now filter for MoM
ibd <- snpgdsIBDMoM(genofile, kinship = T,
sample.id = geno.sample.ids,
snp.id = snpset.ibd,
num.thread = 1)
ibdcoef <- snpgdsIBDSelection(ibd)
ibdcoef <- ibdcoef[ibdcoef$kinship >= kin,]# Filter samples out
related.samples <- NULL
while (nrow(ibdcoef) > 0) {
# count the number of occurrences of each and take the top one
sample.counts <- arrange(count(c(ibdcoef$ID1, ibdcoef$ID2)), -freq)
rm.sample <- sample.counts[1, 'x']
cat("Removing sample", as.character(rm.sample), 'too closely related to', sample.counts[1, 'freq'],'other samples.\n')# remove from ibdcoef and add to list
ibdcoef <- ibdcoef[ibdcoef$ID1 != rm.sample & ibdcoef$ID2 != rm.sample,]
related.samples <- c(as.character(rm.sample), related.samples)
}
genData$SNP <- genData$SNP[!(rownames(genData$SNP) %in% related.samples),]
genData$LIP <- genData$LIP[!(rownames(genData$LIP) %in% related.samples),]预处理后,我们留下316个样本(110个中国人、100个印度人和106个马来人),其特征在于795668个SNP标记和282个脂质物种。请注意,由于LD修剪过程是随机的,因此您的样本量可能略有不同。
分析
主成分分析
既然我们已经完成了预处理,那么使用主成分分析(PCA)检查基因型数据中最大的变异源并找出异常值或聚类模式可能是个好主意。因为我们使用的是S4对象,所以我们将使用SNPRelate中的PCA函数snpgdsPCA。让我们绘制前两个主要组件(PC)。
# PCA
pca <- snpgdsPCA(genofile, sample.id = geno.sample.ids, snp.id = snpset.ibd, num.thread = 1)
pctab <- data.frame(sample.id = pca$sample.id,
PC1 = pca$eigenvect[,1],
PC2 = pca$eigenvect[,2],
stringsAsFactors = F)origin <- read.delim("countryOrigin.txt", sep = "\t")
origin <- origin[match(pca$sample.id, origin$sample.id),]pcaCol <- rep(rgb(0,0,0,.3), length(pca$sample.id)) # Set black for chinese
pcaCol[origin$Country == "I"] <- rgb(1,0,0,.3) # red for indian
pcaCol[origin$Country == "M"] <- rgb(0,.7,0,.3) # green for malaypng("PCApopulation.png", width = 500, height = 500)
plot(pctab$PC1, pctab$PC2, xlab = "PC1", ylab = "PC2", col = pcaCol, pch = 16)
abline(h = 0, v = 0, lty = 2, col = "grey")
legend("top", legend = c("Chinese", "Indian", "Malay"), col = 1:3, pch = 16, bty = "n")
dev.off()不出所料,795668个SNP标记清楚地描绘了这三个群体。研究结果还表明,与印度人相比,中国人和马来人更接近彼此(这一观察结果可以通过分层聚类等方法更好地解决)。此外,前两个PC没有发现明显的异常值。一切都很好,可以继续进行GWA。
全基因组联合
最后,抵抗力。从282种脂质中,我选择了最熟悉的胆固醇作为感兴趣的特征之一。您可以完全自由地选择不同的脂质种类并继续操作。我们将使用Reed等人2015年提供的GWA函数,并进行一些小的修改。我建议您打开脚本GWAS函数。R,然后浏览一遍。这是一个优秀的、文档丰富的并行化实现。注意,glm函数用于确定每个SNP与感兴趣的性状之间的关联的显著性。如果你想知道,glm比lm更通用,因为它可以进行高斯、泊松、二项和多项回归/分类,这取决于你感兴趣的特征是如何分布的(表型文件中的所有脂质都是高斯的)。该GWA函数不会创建变量,而是编写一个.txt汇总表,列出每个SNP的系数估计\β,t和相应的P值,以及相应的基因组坐标。2012年年中,我使用MacBook Pro(8Gb,2.9 GHz英特尔酷睿i7)运行GWA功能花了大约1.5个小时。
# Choose trait for association analysis, use colnames(genData$LIP) for listing
# NOTE: Ignore the first column of genData$LIP (gender)
target <- "Cholesterol"phenodata <- data.frame("id" = rownames(genData$LIP),
"phenotype" = scale(genData$LIP[,target]), stringsAsFactors = F)# Conduct GWAS (will take a while)
start <- Sys.time()
GWAA(genodata = genData$SNP, phenodata = phenodata, filename = paste(target, ".txt", sep = ""))
Sys.time() - start # benchmark一旦完成,我们可以使用所谓的曼哈顿图来可视化结果。我们所需要的只是加载在前一步中编写的.txt汇总表,添加一个带有-\log_{10}(P)的列,并根据所有SNP的基因组坐标绘制这些显著性估计。
# Manhattan plot
GWASout <- read.table(paste(target, ".txt", sep = ""), header = T, colClasses = c("character", rep("numeric",4)))
GWASout$type <- rep("typed", nrow(GWASout))
GWASout$Neg_logP <- -log10(GWASout$p.value)
GWASout <- merge(GWASout, genData$MAP[,c("SNP", "chr", "position")])
GWASout <- GWASout[order(GWASout$Neg_logP, decreasing = T),]png(paste(target, ".png", sep = ""), height = 500,width = 1000)
GWAS_Manhattan(GWASout)
dev.off()此外,一条完整的(分别为虚线)线表示1000000次(分别为10000次)测试中Bonferroni调整的\alpha=0.05的水平。
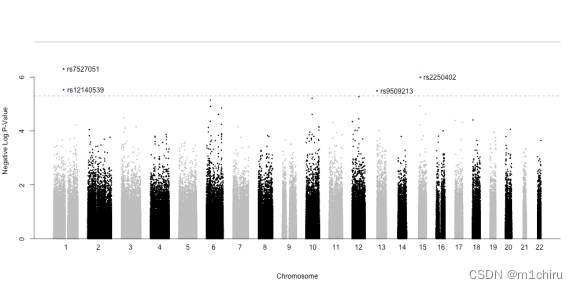
我们发现,共有四个SNP通过了“宽松”的Bonferroni阈值(没有一个通过“硬”阈值)。这些是SNPs rs7527051(Chr.1)、rs12140539(与第一个Chr.1共定位)、rs9509213(Chr.13)和rs2250402(Chr.15)。
在进行这四次点击之前,将所得P值的分布与偶然预期的分布进行对比是有帮助的,以确保没有混淆的系统性偏差。这很容易用分位数-分位数(Q-Q)图来解决。顾名思义,它根据分位数比较两种分布。在目前的情况下,我们想将我们的t统计数据的分布与偶然获得的分布进行对比。如果两个分布的形状相同,Q-Q图将显示一条x=y线。因此,来自可靠GWA分析的Q-Q图将显示一条x=y线,其中只有很少的偏差值表明存在关联。否则,如果线向上或向下移动,GWA分析可能会受到混杂因素的影响。我们将使用GenABEL包中的函数estlambda绘制Q-Q图。
# QQ plot using GenABEL estlambda function
png(paste(target, "_QQplot.png", sep = ""), width = 500, height = 500)
lambda <- estlambda(GWASout$t.value**2, plot = T, method = "median")
dev.off()
由此得到的Q-Q图清楚地描绘了一条与x=y(黑色)重叠的趋势线(λ=0.99,红色)和右尾部的轻微偏差,因此我们可以对我们的结果充满信心。
候选标记的功能
见解最后,我们将尝试使用USCS基因组浏览器(输入基因组浏览器,在文本框中插入SNP ID,输入并缩小),通过搜索其附近的基因来找到这四个候选SNP的功能相关性。我发现rs9509213(Chr.13)正好落在CRYL1(晶体λ1,内含子序列)上,使其成为后续研究的有趣候选者。
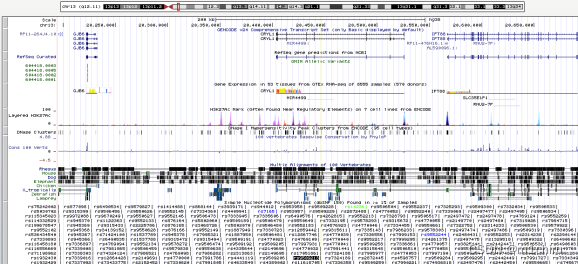
SNP rs9509213在图的底部显示为一个黑色文本框,其坐标由一条垂直的黄线突出显示。CRYL1基因模型显示在染色体模型(最上面)下方的顶部。
有趣的是,最近有一份出版物发现,“POE、HP和CRYL1都与阿尔茨海默病有关,阿尔茨海默病的病理学涉及脂质和胆固醇途径。”。
最后,我想谈谈对候选基因进行实验验证的必要性,以验证由此产生的SNP特征关联。在这种特殊的情况下,可以测试在小鼠的胆固醇水平中过表达或敲除CRYL1的直系同源物的效果。


![[java基础揉碎]do..while循环控制](https://img-blog.csdnimg.cn/direct/52824073c9534084abe66fd4399d8b0d.png)
![[C++]使用纯opencv部署yolov8旋转框目标检测](https://img-blog.csdnimg.cn/direct/e509302a23f34c52aeb473fab2d4a092.jpeg)