| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|
文章目录
前言
本文是UCSF DOCK的使用案例分享,包括DOCK 6.11的de novo Design 模块包含的三项功能:
(1)Generic de novo design
(2)De Novo Refinement
(3)用户自定义生成片段库并实现Focused De Novo Design
一、 软件及操作环境
操作环境:Ubuntu 22.04
软件版本:UCSF DOCK 6.11,安装可以参考这篇博文;UCSF Chimera 1.17.3,UCSF ChimeraX 1.7.1,安装可以参考这篇博文。
二、研究目的
NX-2127是一款具有双重活性的口服小分子靶向蛋白降解剂,可以布鲁顿氏酪氨酸激酶(BTK)蛋白。在与BTK结合的同时,NX-2127还可以招募E3泛素连接酶,使BTK蛋白“泛素化”,从而BTK被蛋白酶体降解。
在本实例中,我们将使用DOCK的通用片段库,从头开始为我们的受体构建新的配体:
首先,展示通用的Generic de novo design,随后是基于anchor的优化生成De Novo Refinement,最后是基于用户自定义生成片段库的Focused De Novo Design。
三、结构文件准备
grid文件来自于前一篇案例分享:UCSF DOCK 分子对接详细案例(02)-并行用于高通量虚拟筛选
也可以在此下载本案例需要的输入文件:下载
新建本案例需要的目录:
cd DOCK_workdir
mkdir 009_denovo_generic 010_denovo_refine 011_denovo_focused
现在,DOCK_workdir 内容如下:

四、 DOCK中 de novo design
4.1 generic de novo design
cd ./009_denovo_generic
将此前建立的格点文件.bmp和.nrg文件复制到本目录:
cp ../003_gridbox/grid.bmp ../003_gridbox/grid.nrg ./
创建参数输入文件generic.in,输入以下内容:
conformer_search_type denovo
dn_fraglib_scaffold_file $DOCKHOME/parameters/fraglib_scaffold.mol2
dn_fraglib_linker_file $DOCKHOME/parameters/fraglib_linker.mol2
dn_fraglib_sidechain_file $DOCKHOME/parameters/fraglib_sidechain.mol2
dn_user_specified_anchor no
dn_torenv_table $DOCKHOME/parameters/fraglib_torenv.dat
dn_name_identifier 8u2e_generic
dn_sampling_method graph
dn_graph_max_picks 30
dn_graph_breadth 3
dn_graph_depth 2
dn_graph_temperature 100.0
dn_pruning_conformer_score_cutoff 100.0
dn_pruning_conformer_score_scaling_factor 2.0
dn_pruning_clustering_cutoff 100.0
dn_mol_wt_cutoff_type soft
dn_upper_constraint_mol_wt 550.0
dn_lower_constraint_mol_wt 0.0
dn_mol_wt_std_dev 35.0
dn_constraint_rot_bon 15
dn_constraint_formal_charge 2.0
dn_heur_unmatched_num 1
dn_heur_matched_rmsd 2.0
dn_unique_anchors 1
dn_max_grow_layers 9
dn_max_root_size 25
dn_max_layer_size 25
dn_max_current_aps 5
dn_max_scaffolds_per_layer 1
dn_write_checkpoints yes
dn_write_prune_dump no
dn_write_orients no
dn_write_growth_trees no
dn_output_prefix 8u2e_generic
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
use_database_filter no
orient_ligand yes
automated_matching yes
receptor_site_file ../002_surface_spheres/selected_spheres.sph
max_orientations 1000
critical_points no
chemical_matching no
use_ligand_spheres no
bump_filter yes
bump_grid_prefix grid
max_bumps_anchor 2
max_bumps_growth 2
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix grid
minimize_ligand yes
minimize_anchor yes
minimize_flexible_growth yes
use_advanced_simplex_parameters no
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_anchor_max_iterations 500
simplex_grow_max_iterations 250
simplex_grow_tors_premin_iterations 0
simplex_random_seed 0
simplex_restraint_min no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_de_novo.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
编写slurm运行脚本generic.sh,写入以下内容:
#! /bin/sh
#SBATCH --time=5:00:00
#SBATCH --nodes=1
#SBATCH --ntasks=10
#SBATCH --job-name=BTK_generic
#SBATCH --output=BTK_genericdock6 -i generic.in
运行:
sbatch cart_min.sh
大约运行30 min。
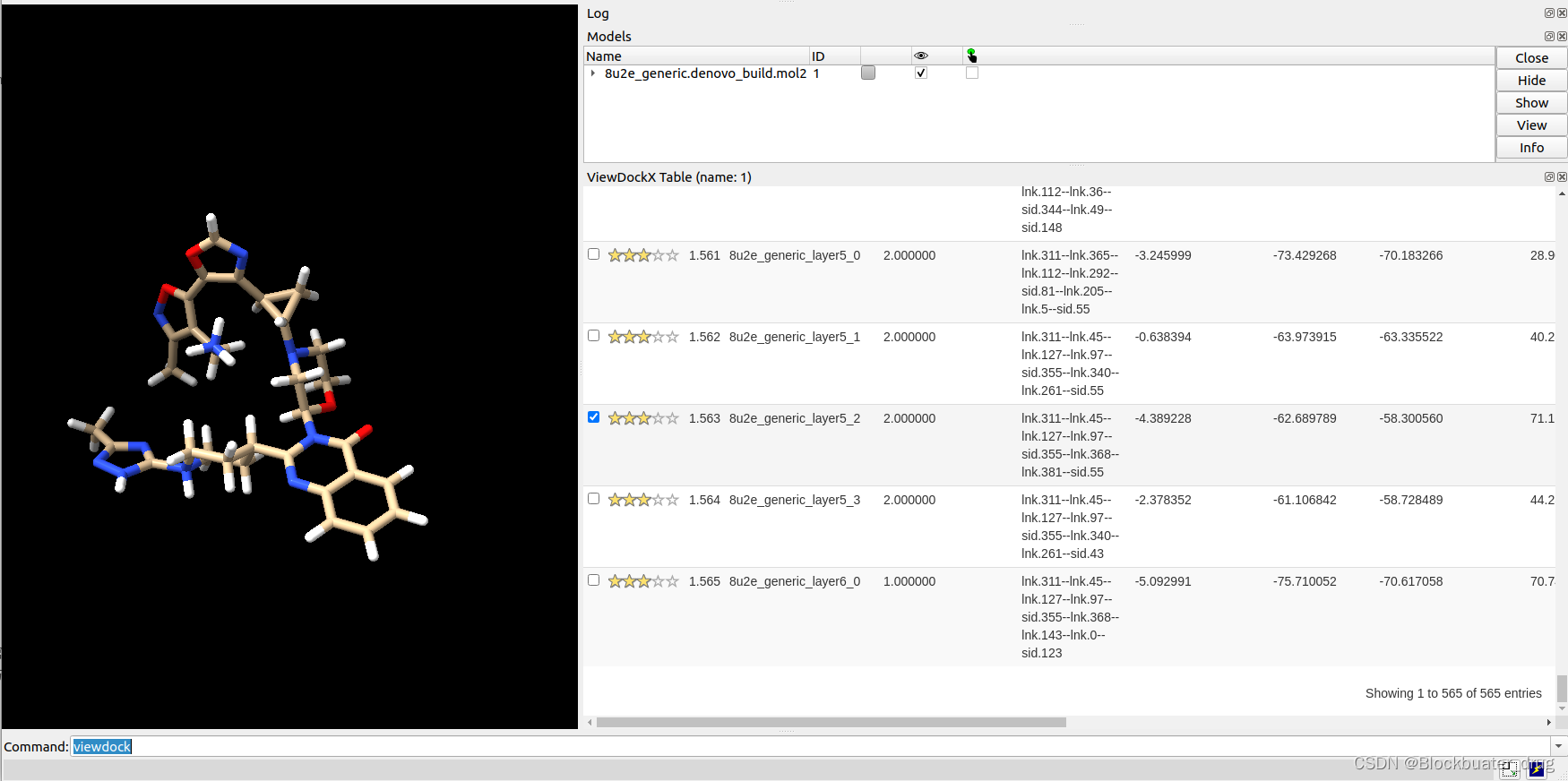
结果文件:8u2e_generic.denovo_build.mol2,生成565个分子,可以通过Chimera或者ChimeraX的ViewDock查看:
4.2 Rescoring the Outputs对输出重新评分
通过4.1中的方式产生的分子已经通过grid score进行评分,可以进一步做rigid docking能量最小化优化,以此更加准确评价生成分子与受体之间结合能力。
vim generic_min.in
conformer_search_type rigid
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
ligand_atom_file 8u2e_generic.denovo_build.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd no
use_database_filter no
orient_ligand yes
automated_matching yes
receptor_site_file ../002_surface_spheres/selected_spheres.sph
max_orientations 1000
critical_points no
chemical_matching no
use_ligand_spheres no
bump_filter yes
bump_grid_prefix grid
max_bumps_anchor 2
max_bumps_growth 2
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix ../003_gridbox/grid
minimize_ligand yes
simplex_max_iterations 1000
simplex_tors_premin_iterations 0
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_random_seed 0
simplex_restraint_min no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_de_novo.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix generic_min
write_orientations no
num_scored_conformers 1
rank_ligands no
运行:
dock6 -i generic_min.in
或者,编写slurm运行脚本generic_min.sh,写入以下内容:
#! /bin/bash
#SBATCH --time=5:00:00
#SBATCH --nodes=1
#SBATCH --ntasks=10
#SBATCH --job-name=BTK_generic.min
#SBATCH --output=BTK_generic.mindock6 -i generic_min.in
slurm运行:
sbatch generic_min.sh
大约运行5 min。
结果文件:generic_min_scored.mol2,对生成565个分子优化,可以通过Chimera或者ChimeraX的ViewDock查看:
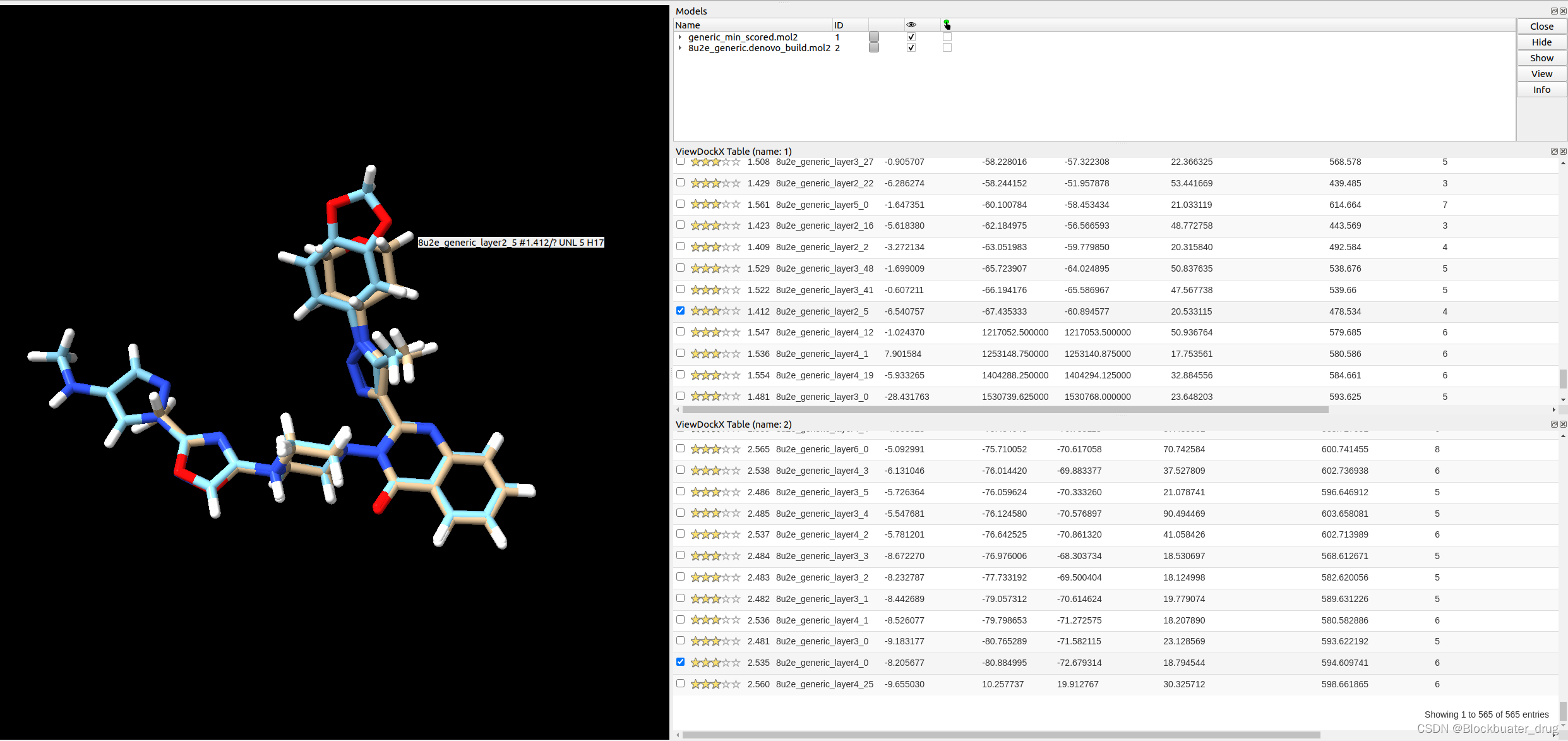 通过对比可以看出,优化后排名靠前的分子与未优化时靠前的分子,结构相近且具有较小的片段,因此该分子或者说该分子系列可能具有很好的受体结合能力。
通过对比可以看出,优化后排名靠前的分子与未优化时靠前的分子,结构相近且具有较小的片段,因此该分子或者说该分子系列可能具有很好的受体结合能力。
五、 De Novo Refinement
cd ../010_denovo_refine
如何在固有配体的指引下生成新的结构,并且与受体依然有良好的结合是药物设计的核心任务。该项工作往往是对配体受体充分了解后开展的,没特定的模式可供套用。
如下图所示,N-甲基哌嗪处于口袋外的溶剂区域,如果采用fast-follow结构修饰的研究策略,那单独改造这个部分是保持活性的同时,重点研究的片段之一。如果考虑采用de novo生成方法,获得全新骨架系列分子的抑制剂,以上方法将不再适用。
一般而言,口袋内部的结合对配体-受体的结合起着非常重要的作用,结合熵变与结合焓变可能都是非常重要的结合贡献力量。保持溶剂区片段不变,寻找具有新型结构的在口袋内部结合的片段,是一项具有挑战性但意义巨大的工作。
本实例是基于已知的药效分子与靶点BTK的结合模式,利用DOCK De Novo Refinement功能,产生新的具有潜在结合的配体分子,保留溶剂区片段(可以尝试,同时保留另外一个靠近口袋外的片段,此处不再展示)。分为三部分:
准备配体
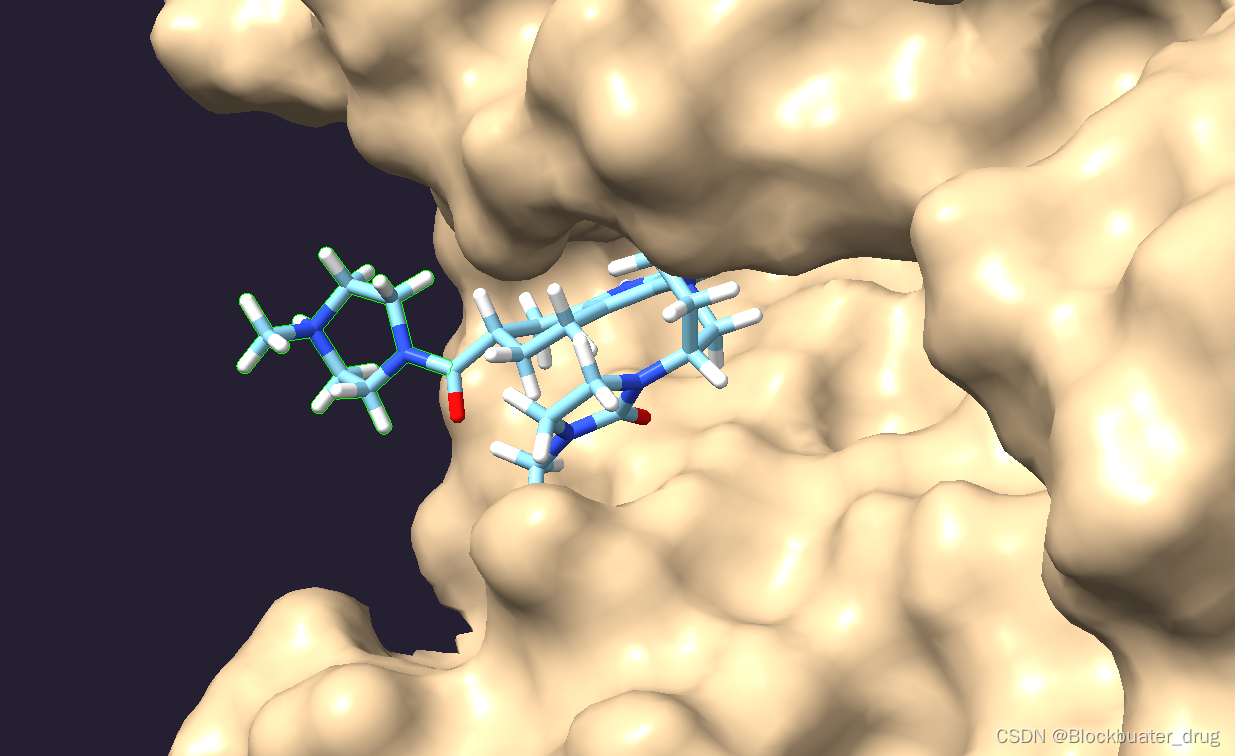 如上图所示,删除配体分子溶剂区片段以外的结构,保存分子为.mol2格式,查看删除分子的连接原子编号(这里是C24),在mol2分子中修改其原子类型为Du,并标记原子名字为Du1,这个位置是产生新结构的起始位置。如下:
如上图所示,删除配体分子溶剂区片段以外的结构,保存分子为.mol2格式,查看删除分子的连接原子编号(这里是C24),在mol2分子中修改其原子类型为Du,并标记原子名字为Du1,这个位置是产生新结构的起始位置。如下:
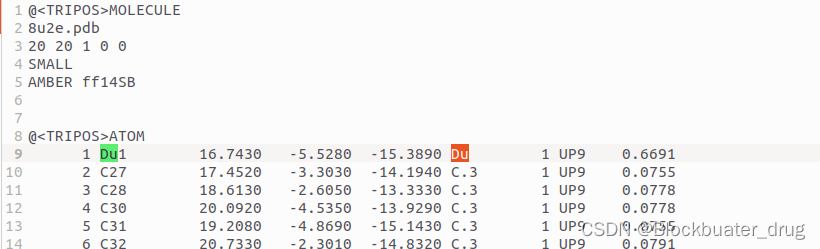
保存文件,名称Chopped_ligand_for_denovo.mol2。
运行Refinement
保存运行参数文件:de_novo_refine.in,内容如下:
conformer_search_type denovo
dn_fraglib_scaffold_file $DOCKHOME/parameters/fraglib_scaffold.mol2
dn_fraglib_linker_file $DOCKHOME/parameters/fraglib_linker.mol2
dn_fraglib_sidechain_file $DOCKHOME/parameters/fraglib_sidechain.mol2
dn_user_specified_anchor yes
dn_fraglib_anchor_file Chopped_ligand_for_denovo.mol2
dn_torenv_table $DOCKHOME/parameters/fraglib_torenv.dat
dn_name_identifier 8u2e_refine
dn_sampling_method graph
dn_graph_max_picks 30
dn_graph_breadth 3
dn_graph_depth 2
dn_graph_temperature 100.0
dn_pruning_conformer_score_cutoff 100.0
dn_pruning_conformer_score_scaling_factor 2.0
dn_pruning_clustering_cutoff 100.0
dn_mol_wt_cutoff_type soft
dn_upper_constraint_mol_wt 550.0
dn_lower_constraint_mol_wt 0.0
dn_mol_wt_std_dev 35.0
dn_constraint_rot_bon 15
dn_constraint_formal_charge 2.0
dn_heur_unmatched_num 1
dn_heur_matched_rmsd 2.0
dn_unique_anchors 1
dn_max_grow_layers 9
dn_max_root_size 25
dn_max_layer_size 25
dn_max_current_aps 5
dn_max_scaffolds_per_layer 1
dn_write_checkpoints yes
dn_write_prune_dump no
dn_write_orients no
dn_write_growth_trees no
dn_output_prefix 8u2e_refine
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
use_database_filter no
orient_ligand no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix ../003_gridbox/grid
minimize_ligand yes
minimize_anchor no
minimize_flexible_growth yes
use_advanced_simplex_parameters no
simplex_max_cycles 1
simplex_score_converge 0.1
simplex_cycle_converge 1.0
simplex_trans_step 1.0
simplex_rot_step 0.1
simplex_tors_step 10.0
simplex_grow_max_iterations 250
simplex_grow_tors_premin_iterations 0
simplex_random_seed 0
simplex_restraint_min yes
simplex_coefficient_restraint 10.0
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_de_novo.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
关于de_novo_refine.in一些参数的说明:
-dn_user_specified_anchor 设置为no时,如果希望运行从头设计运行,其中他们不指定具有虚拟原子的输入锚,DOCK将使用片段库中的部分作为锚点来构建,允许尝试多个不同的锚点和方向。
-dn_sampling_method 可以是“graph”、“random”或“exhaustive”。“graph”将选择与先前选择的改善网格得分的部分相似的片段,因此图将试图以促进改善配体的方式偏向未来的片段选择。术语“random”将导致片段被随机选择。“exhaustive”将确保在每个可能的位置尝试每个可能的片段,会显著增加所需的计算时间。
-dn_graph_max_picks 和dn_num_random_picks(后者在上面未示出)控制每个生长层每个虚原子尝试多少片段。
-dn_max_grow_layers 控制DOCK将从初始锚上的每个虚拟原子向外生长多少个片段。8-9层,通常用于从头开始生长配体而不设置锚。
-minimize_ligand 当设置为yes时,将尝试在每个片段选择后使配体能量最小化。
-minimize_anchor 当设置为yes时,将尝试在连接片段之前使锚能量最小化,而不管锚是由用户提供还是随机选择。对于从头运行,其中锚是以已知方向结合的配体的一部分,最好将此参数设置为“no”。否则,DOCK 可能会在尝试用新的片段生长之前改变你的锚的方向,产生的分子可能不能反映配体通常如何结合。
-simplex_restraint_min 应设置为yes时,该参数基本上将拉伸系绳应用于初始锚,允许其为了能量最小化而稍微偏离其起始位置,但影响RMSD。
运行:
dock6 -i de_novo_refine.in
查看结果
生成8u2e_refine.denovo_build.mol2文件,可以用ChimeraX ViewDock查看。
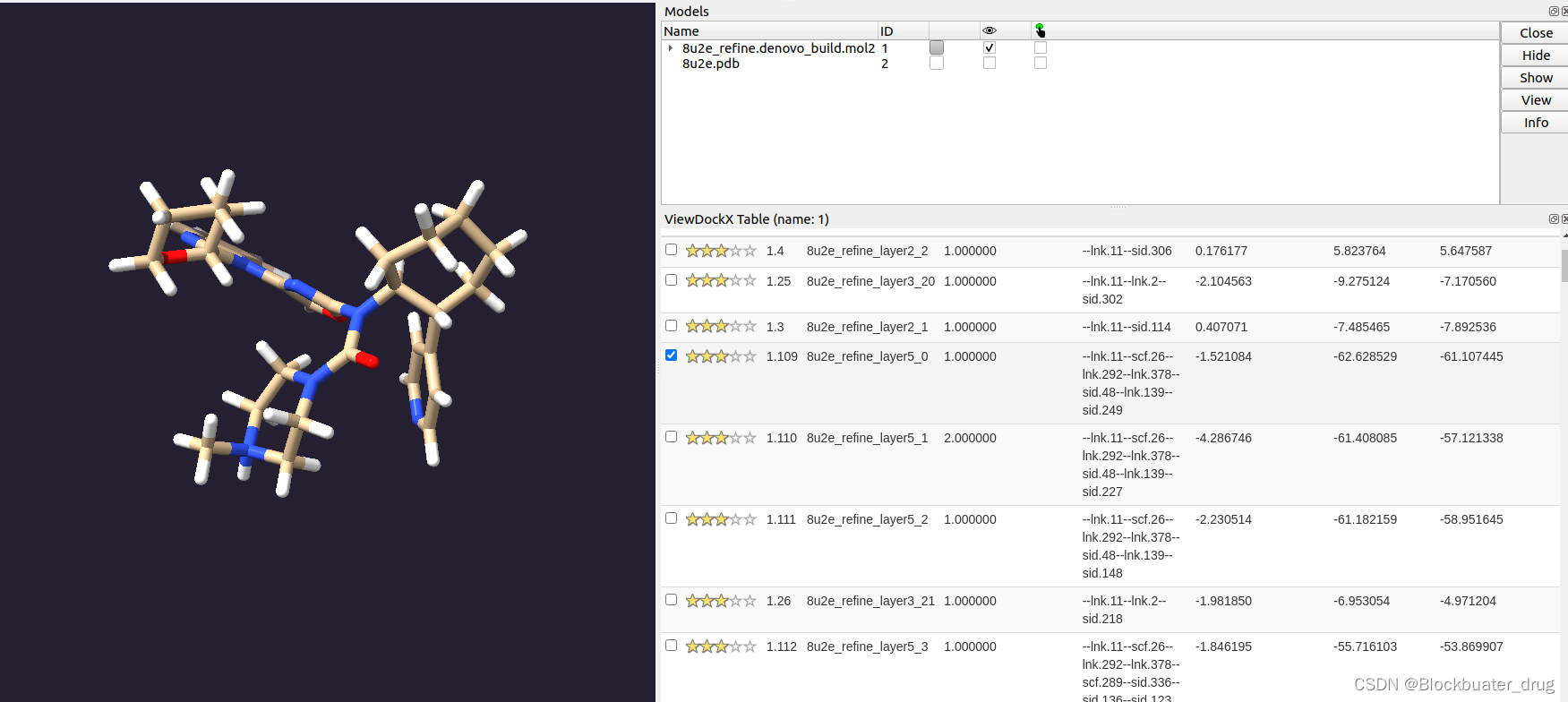
六、 Focused De Novo Design
cd ../011_denovo_focused
生成片段库
DOCK可以读取包含有一个或者多个分子的mol2文件,生成片段库。以输入up9分子为例。
新建片段生成的输入参数文件frag_gen.in,输入以下内容:
conformer_search_type flex
write_fragment_libraries yes
fragment_library_prefix up9_fraglib
fragment_library_freq_cutoff 1
fragment_library_sort_method freq
fragment_library_trans_origin yes
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
ligand_atom_file ../001_structure/up9_rec_withH.mol2
limit_max_ligands no
skip_molecule no
read_mol_solvation no
calculate_rmsd no
use_database_filter no
orient_ligand no
bump_filter no
score_molecules no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_de_novo.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
ligand_outfile_prefix up9_trash
write_orientations no
num_scored_conformers 1
rank_ligands no
运行:
dock6 -i frag_gen.in -o frag_gen.out
生成以下6个文件,可以使用ChimeraX查看。

frag_gen.in输入参数的说明:
-fragment_library_freq_cutoff 作为一种排序机制,只允许出现指定次数的片段进入库。当设置为1时,这允许将出现一次的任何片段添加到库中。
-fragment_library_transs_origin 设置为yes时,会将空间中的所有片段平移到一个位置,这样当在Chimera或ChimeraX等程序中查看时,用户可以查看各种片段,而无需在每次切换正在查看的片段时调整位置。
Fragment Library Merging
可以将DOCK自带的片段库与用户生成的片段库合并使用。
将自带库拷贝进工作目录:
cp $DOCKHOME/parameters/fraglib_s*.mol2 $DOCKHOME/parameters/fraglib_linker.mol2 $DOCKHOME/parameters/fraglib_torenv.dat ./
cat *linker* >> combined_fraglib_linker.mol2
cat *sidechain* >> combined_fraglib_sidechain.mol2
cat *scaffold* >> combined_fraglib_scaffold.mol2
合并torsion files:
python $DOCKHOME/bin/combine_torenv.py fraglib_torenv.dat up9_fraglib_torenv.dat
以上便得到片段和torsion数据库。
Running the Focused De Novo Design
新建Focused De Novo Design的输入参数文件focused.in,输入以下内容:
conformer_search_type denovo
dn_fraglib_scaffold_file combined_fraglib_scaffold.mol2
dn_fraglib_linker_file combined_fraglib_linker.mol2
dn_fraglib_sidechain_file combined_fraglib_sidechain.mol2
dn_user_specified_anchor yes
dn_fraglib_anchor_file ../010_denovo_refine/Chopped_ligand_for_denovo.mol2
dn_torenv_table fraglib_torenv.dat
dn_name_identifier focused_8u2e.combined
dn_sampling_method graph
dn_graph_max_picks 30
dn_graph_breadth 3
dn_graph_depth 3
dn_graph_temperature 100.0
dn_pruning_conformer_score_cutoff 100.0
dn_pruning_conformer_score_scaling_factor 2.0
dn_pruning_clustering_cutoff 100.0
dn_mol_wt_cutoff_type soft
dn_upper_constraint_mol_wt 550.0
dn_lower_constraint_mol_wt 0.0
dn_mol_wt_std_dev 35.0
dn_constraint_rot_bon 15
dn_constraint_formal_charge 2.0
dn_heur_unmatched_num 1
dn_heur_matched_rmsd 2.0
dn_unique_anchors 1
dn_max_grow_layers 5
dn_max_root_size 25
dn_max_layer_size 25
dn_max_current_aps 5
dn_max_scaffolds_per_layer 1
dn_write_checkpoints yes
dn_write_prune_dump no
dn_write_orients no
dn_write_growth_trees no
dn_output_prefix 8u2e_focused
use_internal_energy yes
internal_energy_rep_exp 12
internal_energy_cutoff 100.0
use_database_filter no
orient_ligand no
bump_filter no
score_molecules yes
contact_score_primary no
grid_score_primary yes
grid_score_rep_rad_scale 1
grid_score_vdw_scale 1
grid_score_es_scale 1
grid_score_grid_prefix ../003_gridbox/grid
minimize_ligand no
atom_model all
vdw_defn_file $DOCKHOME/parameters/vdw_de_novo.defn
flex_defn_file $DOCKHOME/parameters/flex.defn
flex_drive_file $DOCKHOME/parameters/flex_drive.tbl
运行:
dock6 -i focused.in -o focused.out
查看结果:
在以上演示条件下,生成1061个分子,可以通过ViewDock查看。
实际使用,片段库和生成参数均需要摸索和优化,才能生成需要的新分子,是一个不断优化和探索的过程。
总结
在本实例中,我们使用DOCK的通用片段库,从头开始为我们的受体构建新的配体;也使用DOCK片段数据库构建功能建立片段数据库用于分子生成。主要分为三个部分:首先,展示通用的Generic de novo design,随后是基于anchor的优化生成De Novo Refinement,最后是基于用户自定义生成片段库的Focused De Novo Design。
参考资料
- https://ringo.ams.stonybrook.edu/index.php/Main_Page
- https://dock.compbio.ucsf.edu/DOCK_6/dock6_manual.htm
- https://pubs.acs.org/doi/pdf/10.1021/acs.jcim.3c01031
| 欢迎浏览我的CSND博客! Blockbuater_drug …点击进入 |
|---|


![BUUCTF---[极客大挑战 2019]LoveSQL1](https://img-blog.csdnimg.cn/direct/ed89ce4c71244b55a6e4cd9735263eab.png)