rm(list = ls())
library(Rmisc)
library(ggplot2)
library(dplyr)data <- iris
x<-iris$Sepal.Length
#求置信区间方法1##
confidence_interval <- t.test(x)$conf.int
print(confidence_interval)
#[1] 5.709732 5.976934
#attr(,"conf.level")
#[1] 0.95#求置信区间方法2##
CI(x,ci=0.95)##基于library(Rmisc)##密度图##
hist(x,##重要性特征col='black',border='yellow',main='重要性',#xlab='',freq = F)#纵坐标为组距x频率
lines(density(x))##添加曲线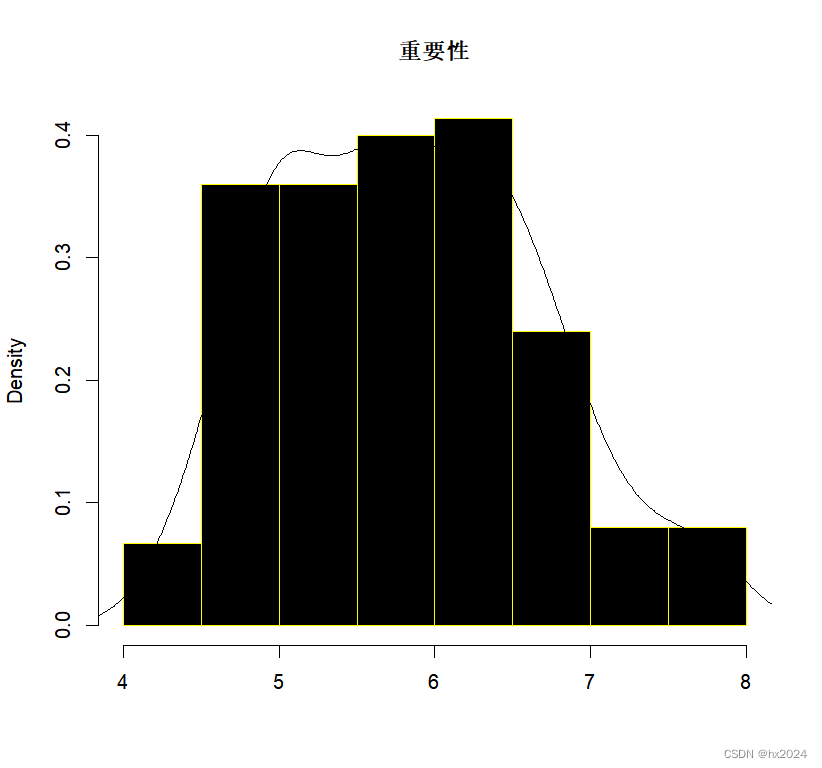
根据P值进行特征筛选
#结果相当于P值越小,所计算的特征越大#
#结果相当于P值越小,所计算的特征越大#
df$feature1 <- -log10(df$adj.P.Val)#这两个是对等的
df$feature2 <- log10(1/df$adj.P.Val)#这两个是对等的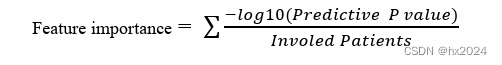
随后可以根据正态分布频率区间筛选95%区间外的重要特征数据
参考:
R语言计算一组数据的置信区间的简单小例子 - 知乎 (zhihu.com)
Tumor microenvironment evaluation promotes precise checkpoint immunotherapy of advanced gastric cancer
![code摘录日记[矩阵变元素,变列向量,3D表面图,table行列设置] Matlab](https://img-blog.csdnimg.cn/direct/3569472143f047b5a87d93d60f561e42.png)