前记
PopLDdecay是一款用于进行种群遗传学和关联分析的软件。它可以在全基因组水平上进行基因型数据的相关性和衰减分析,帮助研究人员探索种群间的遗传差异和突变选择的模式。
使用PopLDdecay可以实现以下功能:
- 遗传距离的计算:可以计算遗传距离,包括最小二乘法估计和Cockerham和Weir的方法。
- 关联分析:可以计算遗传连锁块、相关关系、平衡和不平衡的情况。
- 衰减分析:可以计算将遗传相关性与遗传距离之间的关系进行衰减分析。
PopLDdecay支持多种数据格式的输入,包括VCF、HapMap、PLINK和BEAGLE等格式。它还提供了直观的可视化功能,可以生成遗传距离和衰减图,帮助用户更好地理解和解释结果。
此外,PopLDdecay还具有高效的计算能力和并行处理功能,可以加快分析速度,提高效率。
总之,PopLDdecay是一款功能强大且易于使用的软件,适用于基因型数据的遗传相关性和衰减分析,在种群遗传学和关联研究中具有重要的应用价值。
一、软件的安装与配置
PopLDdecay官网地址如下:GitHub - BGI-shenzhen/PopLDdecay: PopLDdecay: a fast and effective tool for linkage disequilibrium decay analysis based on variant call format(VCF) files![]() https://github.com/BGI-shenzhen/PopLDdecay
https://github.com/BGI-shenzhen/PopLDdecay
软件的安装和配置代码如下:
#安装
git clone https://github.com/BGI-shenzhen/PopLDdecay.git
cd PopLDdecay
chmod 755 configure
./configure
make
mv PopLDdecay ./bin/#环境变量配置
vim ~/.bashrc
export PATH="/public/home/gguo/biosoft/PopLDdecay/bin:$PATH"
source ~/.bashrc二、LD衰减距离的计算
使用VCF文件进行LD计算,代码如下:
#计算LD
cd /public/home/
PopLDdecay -InVCF sample.vcf -OutStat LDdecay#绘制LD衰减图
perl Plot_OnePop.pl -inFile LDdecay.stat.gz -output Fig第一行代码运行后会产生LDdecay.stat.gz文件,即为LD计算结果。
使用第二行代码进行衰减图的绘制,会产生PDF和PNG两种格式文件。
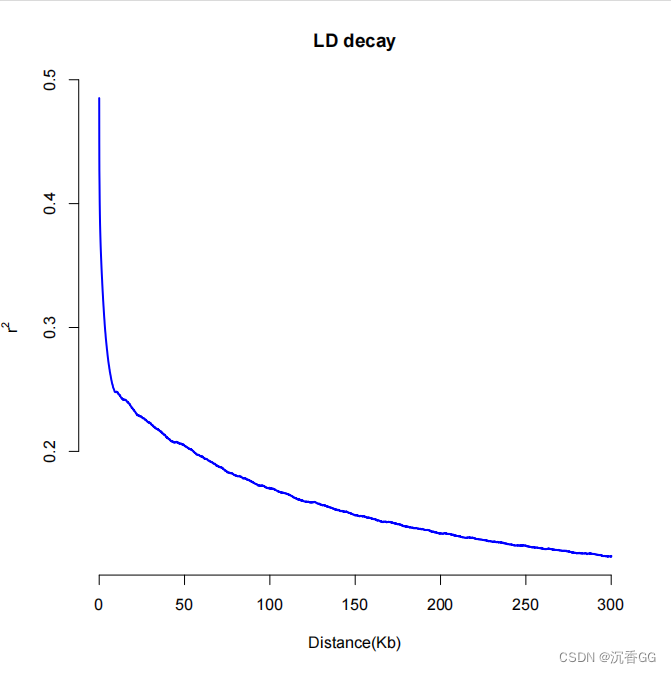
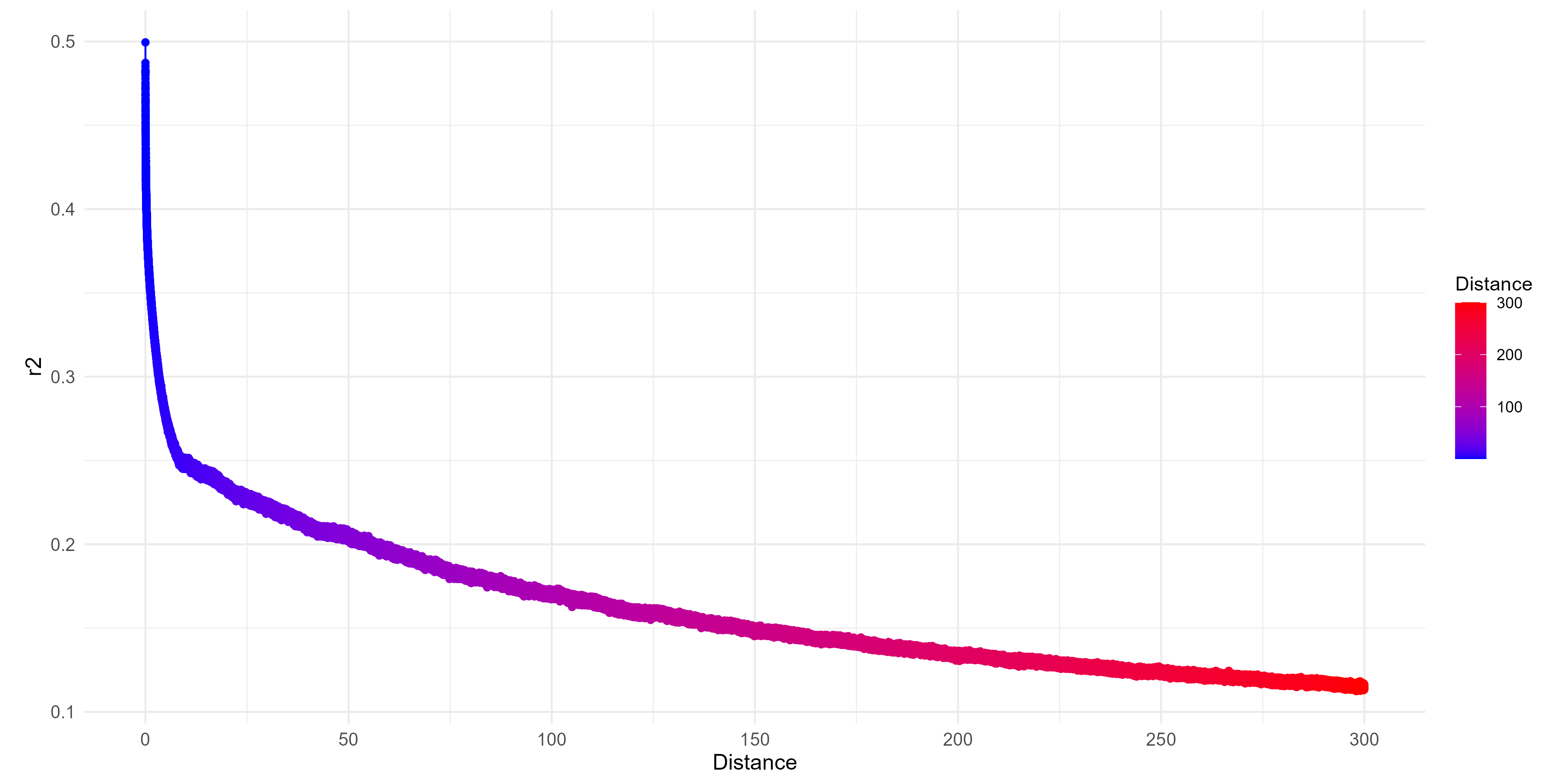
三、R可视化LD衰减图
主要使用ggplot2包,代码如下:
######### 绘制LD衰减距离图 ###########
rm(list = ls()) # 清除工作环境中的变量
setwd("D:/Working-Folder/GWAS data/")
library(ggplot2)ld_data <- read.table("LDdecay.txt", header = TRUE, sep = "\t")
Distance <- ld_data$Distance..Kb.
r2 <- ld_data$r.2# 检查数据
head(distance)
head(average_r2)# 创建LD衰减图
ld_plot <- ggplot(data = ld_data, aes(x = Distance, y = r2, color = Distance)) +geom_line(size = 0.5) + # 增加线条粗细scale_color_gradient(low = "blue", high = "red") + # 渐变色theme_minimal() +theme(legend.position = "right", # 将图例放在图的右侧plot.title = element_text(face = "bold", size = 14, hjust = 0.5),axis.title.x = element_text(size = 12),axis.title.y = element_text(size = 12),axis.text.x = element_text(size = 10),axis.text.y = element_text(size = 10),plot.margin = margin(5.5, 14, 5.5, 14))# 增加图形边距ld_plot <- ld_plot +geom_point(size = 1.5, shape = 19, stroke = 0.5, fill = "white") # 改变点的形状、大小和颜色ld_plot <- ld_plot +scale_x_continuous(breaks = seq(0, max(distance), by = 50), labels = seq(0, max(distance), by = 50)) # 调整x轴刻度# 显示图形
print(ld_plot)#保存
ggsave("LD_decay_plot.png", ld_plot, width = 12, height = 6, dpi = 300)
参考文献
Zhang C, Dong SS, Xu JY, He WM, Yang TL. PopLDdecay: a fast and effective tool for linkage disequilibrium decay analysis based on variant call format files. Bioinformatics. 2019 May 15;35(10):1786-1788. doi: 10.1093/bioinformatics/bty875. PMID: 30321304.
后记
以上是PopLDdecay的简单使用方法,简单记录一下。
2024.3.29
----CXGG
千里之行,始于足下。