基于多数据源融合的医疗知识图谱框架构建研究
- 提出背景
- 医学数据源
- 医学数据获取方法
- 知识图谱的构建
提出背景
论文:基于多数据源融合的医疗知识图谱框架构建研究
本文以医疗领域的实际应用需求为出发点,从医疗大数据获取、医疗实体及关系标注、医疗实体识别、医疗实体链接、医疗实体关系挖掘、
中文医疗知识图谱表示和存储等关键技术入手,提出了多数据源融合的医疗知识图谱构建的理论框架。
国外已经建成了 医 疗 领 域 资 源 库 一 体 化 医 学 语 言 系 统
( UMLS ) 、 医 学 系 统 命 名 法—临 床 术 语(SNOMED CT) 知识库等资源。
医学数据源
医疗大数据通常包括规范的电子病历、医疗健康社区中的用户生成内容(UGC)、医疗词典资源和相关政策文件等,这些数据根据其表现形式,可以被分为结构化数据、半结构化数据和非结构化数据三类。
- 结构化医疗数据:主要包括医疗疾病词典、中医药词典等标准化资源,例如国际疾病分类手册ICD11。
- 半结构化医疗数据:主要涵盖电子病历和医学文献等,这些数据虽然具有一定的格式,但仍保留了大量的自由文本信息。
- 非结构化医疗数据:包括医疗相关的百科词条、医疗论坛上的文本数据等,这些数据通常以自由形式文本存在,缺乏固定格式。
在构建医疗知识图谱时,需要重点考虑以下数据资源:
- 医疗词典:如ICD11,提供专业性强的数据源。
- 电子病历:临床医生的病程记录,是非常重要的数据源。
- 医学文献:科研成果的表现形式,是高质量的医疗数据源。
- 互联网上的用户生成内容:随着信息技术的发展,这类数据量大且质量逐渐提高,成为医疗知识图谱重要的补充数据。
现有的医疗知识库依赖于单一数据源和专家知识,没有充分利用这些多样的医疗大数据资源。
通过融合多方数据资源,可以显著提高医疗知识图谱的实际应用价值,特别是对于临床辅助决策和医疗问答系统等应用场景。
医学数据获取方法
获取医疗知识图谱的数据源主要涉及以下几个步骤和方法:
-
确定数据源种类:首先需要确定你的医疗知识图谱需要哪些类型的数据源。如前所述,这些数据源通常分为结构化数据(如医疗词典、疾病分类手册),半结构化数据(如电子病历、医学文献摘要),以及非结构化数据(如医疗论坛上的文本、用户生成内容)。
-
获取结构化和半结构化数据:
- 医疗词典和分类手册:可以从官方医疗机构或国际医疗标准组织获取,例如世界卫生组织(WHO)发布的国际疾病分类(ICD)手册。
- 电子病历:这些数据通常由医院和其他医疗机构掌握,获取这些数据需要与这些机构建立合作关系,遵循相关的隐私保护和数据使用规定。
- 医学文献:可以通过访问医学数据库和图书馆获取,如PubMed、ScienceDirect等,这些资源通常提供大量的医学研究文章和文献摘要。
-
获取非结构化数据:
- 互联网论坛和社区内容:可以通过网络爬虫技术自动收集相关网站上的用户生成内容,但需要注意遵守网站的爬虫协议和用户隐私保护政策。
- 社交媒体:同样可以使用网络爬虫技术从各大社交媒体平台收集关于医疗健康的讨论和帖子。
-
数据清洗和预处理:收集到的数据需要进行清洗和预处理,包括格式统一、去除噪声数据、数据分词和词性标注等,以提高数据质量,为后续的实体识别和关系抽取打好基础。
-
遵守法律法规和伦理准则:在获取和使用医疗数据时,必须严格遵守相关的法律法规,尤其是关于个人隐私保护和数据安全的规定,以及伦理审查的要求。
获取医疗知识图谱数据是一个复杂且细致的过程,需要充分考虑数据来源的可靠性和合法性,以及数据处理过程中的技术细节。
知识图谱的构建
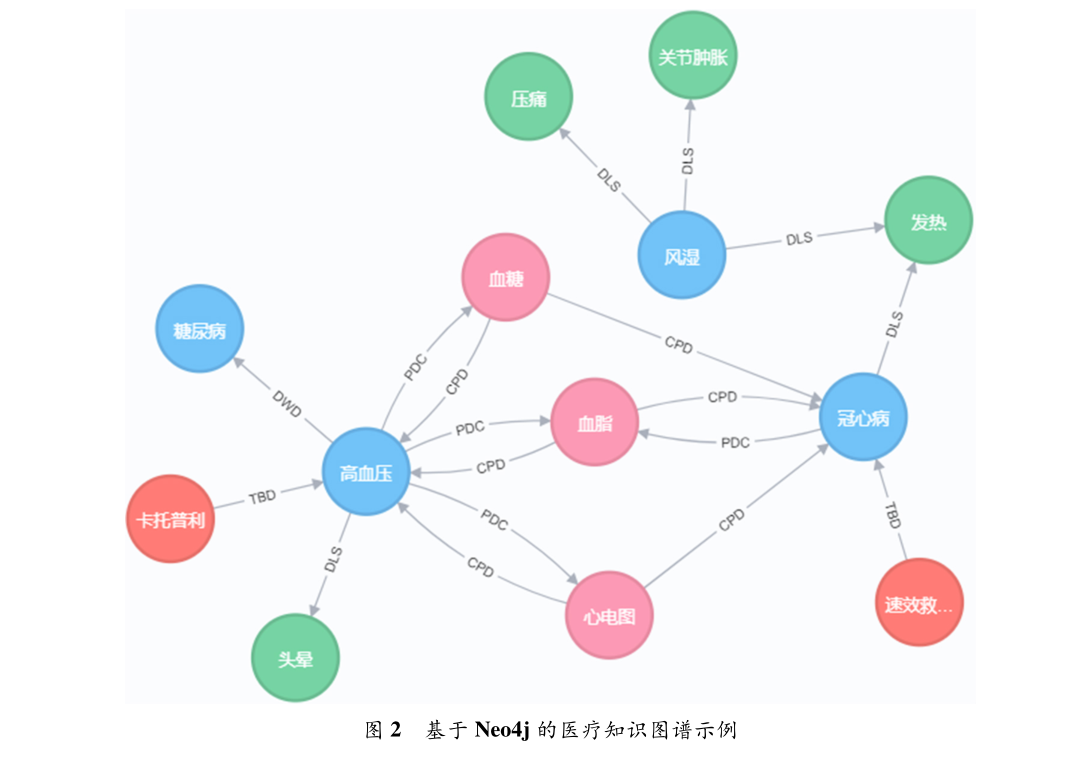
中文医疗知识图谱的构建过程,包括多数据源融合、医疗文本数据处理、医疗实体识别、实体及实体关系标注、实体链接与知识融合、实体关系抽取、知识图谱表示及存储和图谱的动态构建等关键技术环节。
-
多数据源融合:首先通过不同渠道收集医疗文本大数据,然后进行格式统一、数据清洗、分词和词性标注,采用机器学习方法进行医疗实体和实体关系的标注。
-
医疗文本数据处理:包括数据格式统一、数据清洗和筛选,以及分词和词性标注。分词中,考虑到医疗领域词汇的专业性和新词的频繁出现,需要高质量的医学词典支持。
-
医疗实体识别:包括基于医学词典的方法、基于规则的方法和基于机器学习的方法。机器学习方法,尤其是CRF和基于深度学习的方法,在实体识别中表现较好。
-
医疗实体及实体关系标注:通过特定的标识符对医疗实体进行标注,并定义了一系列实体间的关系类型,如疾病与症状之间的关联。
-
医疗实体链接与知识融合:通过实体链接技术将不同数据源中的同一实体关联起来,提升了医疗知识图谱的覆盖度和准确性。
-
医疗实体关系抽取:采用基于模式匹配、基于特征和基于机器学习的方法来挖掘实体间的关系,这是构建知识图谱的关键环节。
之所以用实体识别和关系抽取子解法,是因为数据结构化特征。
将非结构化的文本数据转化为可用于构建知识图谱的结构化数据。
例子:使用自然语言处理技术识别文本中的医疗实体(如疾病、症状)及它们之间的关系。
-
知识图谱表示及存储:介绍了利用RDF和图数据库等技术对知识图谱进行表示和存储,以及如何通过URI实现实体数据间的链接。
-
动态构建知识图谱:利用Spark技术平台对大数据进行快速处理,并通过实时更新技术确保知识图谱的时效性和准确性,以满足实际应用的需求。
整个构建过程强调了从数据采集、处理、到知识提取和存储的每个环节,明确了在中文医疗领域构建知识图谱的技术路径和方法,旨在提高临床辅助诊断和健康问答系统的知识基础。
以糖尿病为例,构建中文医疗知识图谱的具体过程可以分解为以下步骤:
-
多数据源融合:
- 从医疗数据库和文献中收集关于糖尿病的数据,如PubMed、中国知网等。
- 获取电子病历中关于糖尿病的诊断、治疗和管理信息。
- 收集社交媒体和医疗论坛上患者和医生讨论糖尿病的经验和见解。
-
医疗文本数据处理:
- 将收集到的数据统一格式,如将文本转换为XML格式。
- 对数据进行清洗,去除无关信息,如广告、重复内容等。
- 使用医疗领域的分词工具和医学词典进行分词和词性标注,确保糖尿病及其相关术语如“高血糖”、“胰岛素”等被正确识别。
-
医疗实体识别:
- 采用基于规则和机器学习的方法,识别糖尿病及其相关实体,如症状(多饮、多尿)、并发症(视网膜病变、肾病)等。
- 使用CRF、深度学习等技术提高实体识别的准确性。
【医学实体识别】从糖尿病论文和临床指南中,做关键信息分类
-
医疗实体及实体关系标注:
- 标注实体及其属性,例如,将“糖尿病”标注为疾病实体,将“多饮”、“多尿”标注为症状实体。
- 定义实体间的关系,如“糖尿病”导致“视网膜病变”。
-
医疗实体链接与知识融合:
- 将不同数据源中关于糖尿病的实体链接起来,形成统一的知识视图。
- 使用实体链接技术解决实体歧义问题,确保不同数据源中的相同实体被正确识别和合并。
-
医疗实体关系抽取:
- 使用模式匹配和机器学习方法从文本中抽取糖尿病的实体关系,如糖尿病与其并发症之间的关系。
- 根据抽取到的实体和关系构建知识图谱中的边。
-
知识图谱表示及存储:
- 使用RDF和图数据库如Neo4j存储知识图谱,将糖尿病及其相关实体和关系以图的形式表示。
- 每个实体和关系都分配唯一的URI,方便链接和查询。
-
动态构建知识图谱:
- 利用Spark等技术平台处理新增的医疗大数据,定期更新糖尿病知识图谱,保持信息的时效性。
- 实时更新知识图谱,反映最新的研究成果和临床指南。
问题:基于多数据源融合的医疗知识图谱框架构建研究
具体到点的逻辑关系:通过收集与整合不同数据源,清洗预处理数据,识别关键实体与关系,再通过知识融合去重及优化存储查询机制,并定期更新维护,实现了多数据源的有效融合构建医疗知识图谱。