Pocket based Molecular Diffusion Model (PMDM) 模型 是 腾讯AI lab 2023年发表在预印本上的文章,第一作者为Huang Lei。文章链接:https://www.biorxiv.org/content/10.1101/2023.01.28.526011v1.full.pdf
当前,该文章已经正式发表在nature communications上。

PMDM 模型通过二元等变扩散网络,利用全局和局部分子动力学信息,将目标口袋的 3D 结构感知为条件信息,并结合分子和蛋白质之间的相互作用,学习分子概率。PMDM可以生成结构有效且构效合理的,契合口袋的3D分子。
一、PMDM 模型介绍
传统的3D分子生成的扩散模型,将口袋信息表征为3D点云。在向前过程中,往小分子中,逐步添加噪音。神经网络的目标是学习逆向地,在口袋条件下的去噪过程。但是,在这些3D分子生成的扩散模型中,无法处理分子边(原子键)的信息,因为使用点云表示分子和口袋原子的坐标和原子类型,没有表示原子和原子之间的化学键信息。
为了解决这个问题,PMDM制定一个二元扩散网络,构造了两种类型的虚拟边:局部边和全局边。原子间距离低于特定阈值的原子对通过共价局部边缘键合,因为当两个原子彼此足够接近时,化学键可以主导原子间力,而全局边缘连接到其余原子对以模拟范德华作用力。
此外,PMDM设计了服从分子几何系统的平移、旋转、反射和排列等变性的等变动态核。
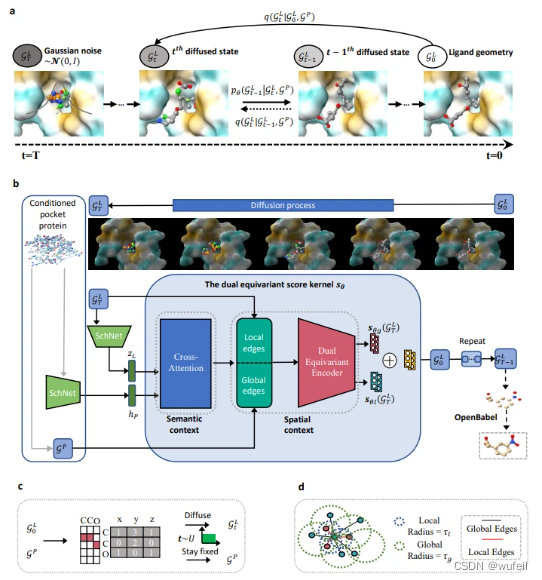
PMDM的模型结构如下图。将蛋白质点云数据输入不变编码器 SchNet 以获得语义表示。 然后通过交叉注意力层将语义信息与小分子融合。为小分子点云数据定义局部和全局边。 然后将具有两种边的小分子和口袋蛋白质经过二元变编码器,该编码器处理不同的边并保持蛋白质空间信息固定,实现去噪。经过重复T次去噪过程,生成新的分子点云(原子类型和原子坐标生成),然后使用obabel构建原子键,生成3D分子。
二、模型性能介绍
PMDM 训练使用的是CrossDock数据集。10W个分子-口袋对用于训练,100个分子-口袋对用于评估。PMDM 的评估指标包括:Vina score, vina score低于参考分子的比例,QED,SA,logP, Lipinski,以及采样时间。
CVAE, AR-SBDD,and DiffSBDD,CVAE 和 AR-SBDD模型作为参考。模型对比结果如下图:
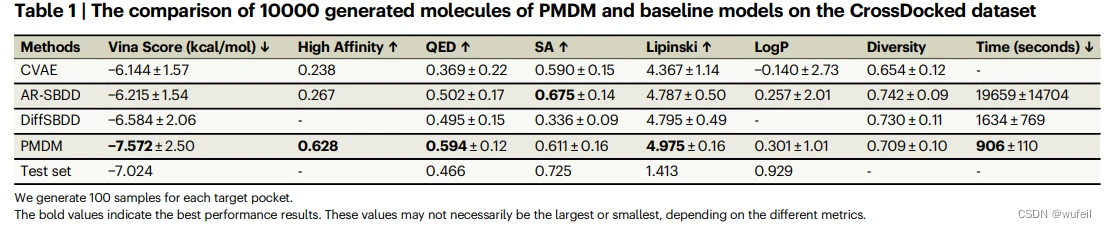
从表中可知,PMDM在生成分子的vina score, QED,Lipinski,采样时间上均明显优于对比模型。
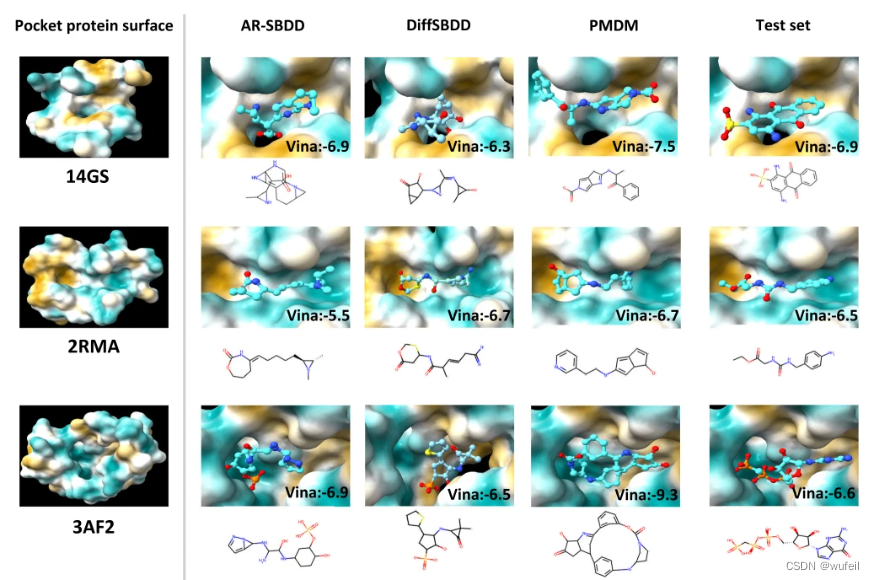
作者以GLUTATHIONE S-TRANSFERASE,Cyclophilin,Pantothenate 为例,进行了分子生成测试,如下图展示了生成的分子中打分最好的分子结构,以及在口袋中的pose。看起来PMDM生成分子要更加的合理。
随后,作者对比了PMDM,参考模型,训练集和测试集,在生成分子的环的数量上的分布,发现PMDM生成分子的环的分布上,更接近真实的训练集和测试集结果,如下图。

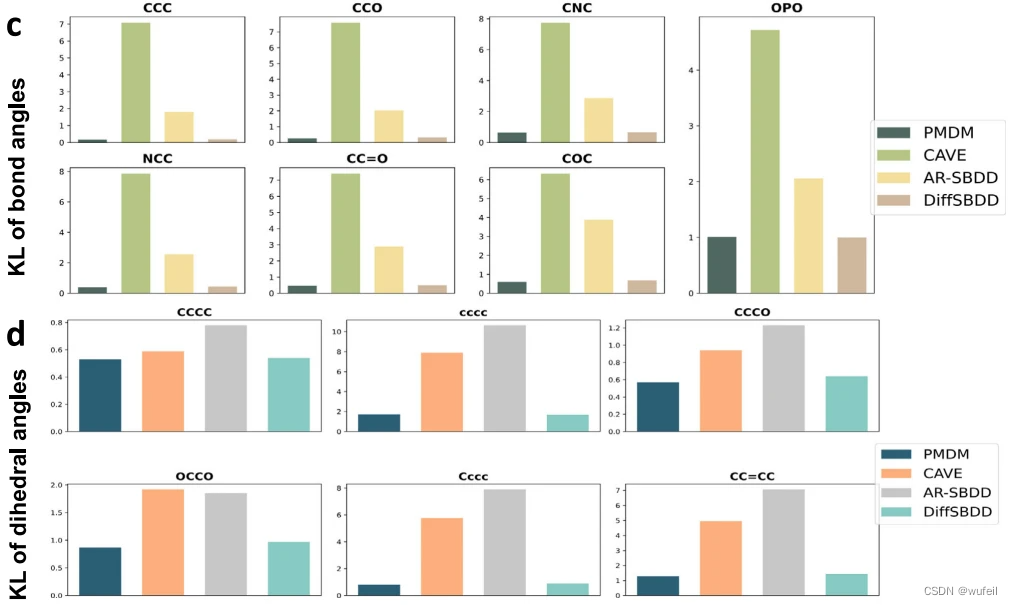
在生成小分子的构象方面,作者测量了与训练集的键角和键长的KL散度,如下图。PMDM的KL散度是最小的,说明PMDM生成的分子的键长与真实数据集的分布最相似,更符合真实化学分子的结构。
在lead优化方面,作者以 SARS-CoV-2 main protease为例。以下图B中的蓝色小片段作为seed(仅包含3个原子),然后进行分子生长,生成了4W个分子。图g中展示了从中挑选的两个高结合力,低MMPBSA的分子,及其结合模式。这两个分子与参考分子具有类似的骨架。

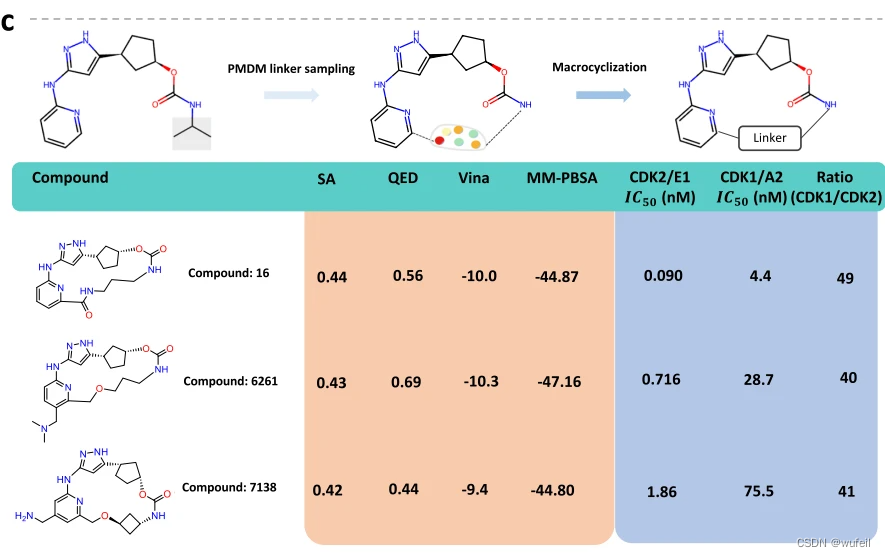
作者以CDK2作为另一个片段延伸的案例。目的是替换下图a中的吡啶环。使用PMDM生成了1W个分子,经过vina对接和MMPBS的分析,以及人工挑选,最终有四个分子被合成。四个分子均展现出更好的酶学活性。其中一个分子,同时展现出更好的选择性。
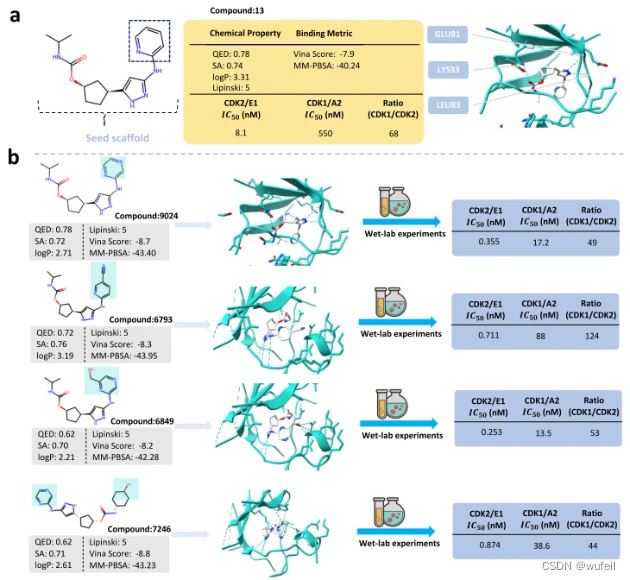
作者继续尝试使用PMDM为CDK2的分子,生成linker,成环。在参考分子的基础上,生成了5000个分子。同样,经过了vina对接,MMPBSA以及人工肉眼筛查,选择了3个大环分子,进行了湿实验。3个分子均展现出更好的酶活,和相似的选择性。如下图。
三、PMDM 模型评测
3.1 项目环境准备
复制项目代码
git clone https://github.com/Layne-Huang/PMDM.git安装环境,并激活环境
conda env create -f mol.yml
conda activate mol
安装biopython,torch-scatter, torch-sparse, torch-cluster,torch-geometric。注意:torch-geometric的版本,不能超过2.4.0,作者代码是适配2.4.0版本。
pip install biopython
pip install torch-scatter torch-sparse torch-cluster torch-spline-conv -f https://pytorch-geometric.com/whl/torch-2.1.1%2Bcu118.html
pip install torch-geometric==2.4.0torch-scatter, torch-sparse, torch-cluster,torch-geometric这几个包的安装有些困难,相互之间存在依赖关系。上述命令中,适用于cuda为11.8的版本。对于其他的版本,需要知道合适的cuda版本后,安装torch,再基于torch的版本和cuda版本,安装torch-scatter, torch-sparse, torch-cluster(这一步很重要,使用pip安装指定版本),然后再安装torch-geometric。大家可以参考:https://blog.csdn.net/qq_54478153/article/details/122683064
安装其他支持模块
conda install einops准备docking的环境,安装QuickVina 2:
wget https://github.com/QVina/qvina/raw/master/bin/qvina2.1
chmod +x qvina2.1在对接时,需要将分子和蛋白转化为PDBQT文件,需要新建一个Adt环境。在linker生成时会自动调用。模型评估也会自动调用。
conda env create -f evaluation/env_adt.yml下载作者训练好的checkpoint,放置在./目录。下载网址为:https://zenodo.org/records/10630921
3.2 口袋生成分子及评估
3.2.1 生成分子
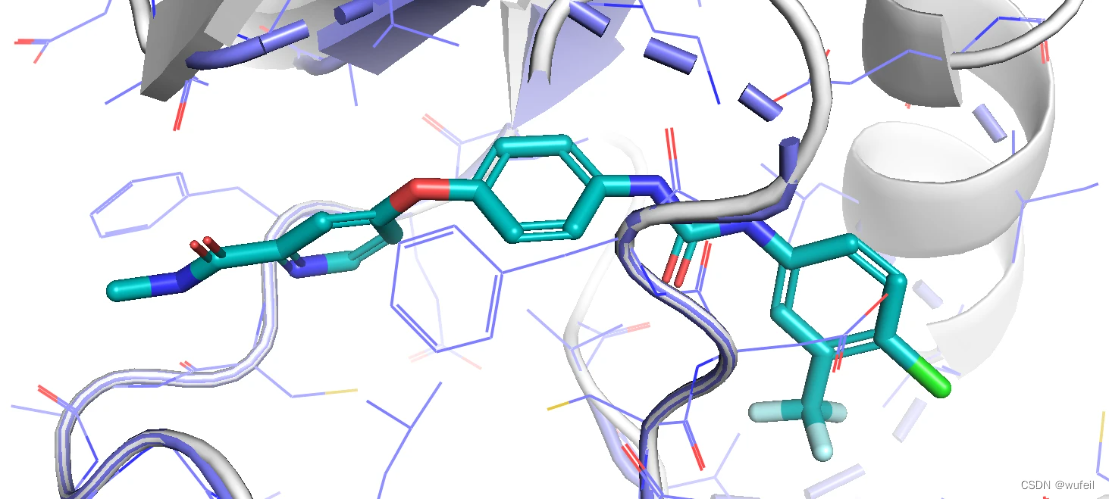
以我们老演员,3WZE为例。口袋及原参考分子见下图。将口袋pdb文件(小分子周围8埃)和小分子sdf文件,放置在新建的./3WZE文件夹内。
运行如下代码,为3WZE口袋生成分子:
python -u sample_for_pdb.py \--ckpt ./500.pt \--pdb_path 3WZE/pocket.pdb \--num_atom 32 \--num_samples 100 \--sampling_type generalized默认生成100个分子,存放在./3WZE文件夹下。
使用如下命令, 合并成一个sdf文件,以便查看。
for i in `ls ./3WZE/*.sdf | sort -t'_' -k 4,4 -n `; do cat $i >> ../all.sdf; done以下是生成分子在口袋中的位置。生成分子的位置比较OK,没有与口袋有严重的冲突。
PMDM-3WZE_Generated-case
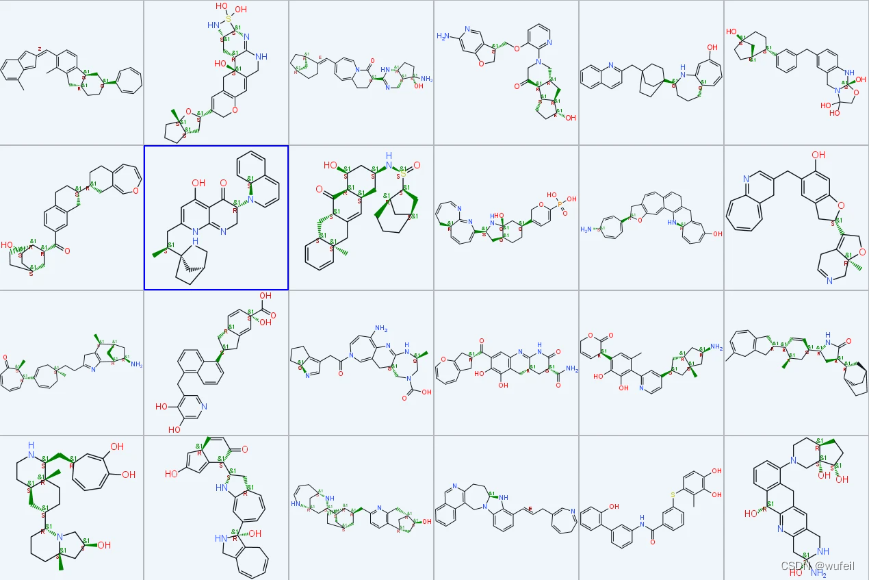
下图是生成分子的2D示例,可以看到生成的分子的手性很多,还有一些奇怪的单双键连接。
此外,生成的小分子构象也有奇怪的地方,例如,扭曲的不饱和环:
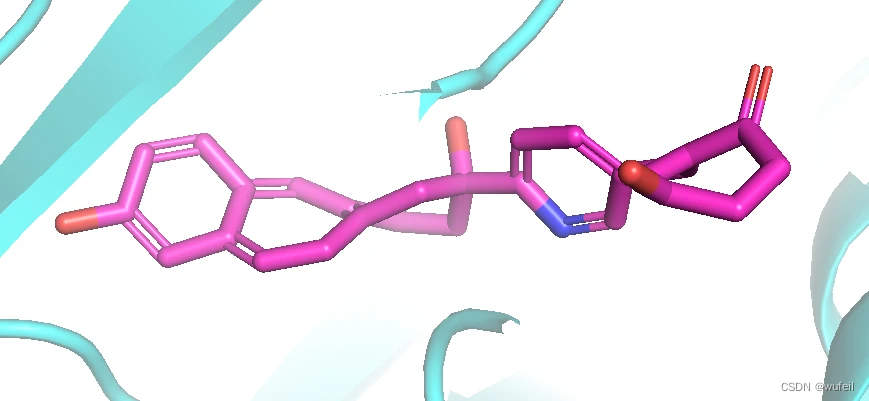
本想着使用作者的评估方法进行评估生成分子的情况,但是GitHub中的评估方式,在新版本的代码中,没有相应的代码(4月9日的版本,作者一直在根据读者的bugs反馈,不断更新、修改GitHub上的PMDM项目)。因此,就是用原位的形状相似性,药效团相似性,对接直接评估。
3.2.2 形状相似性
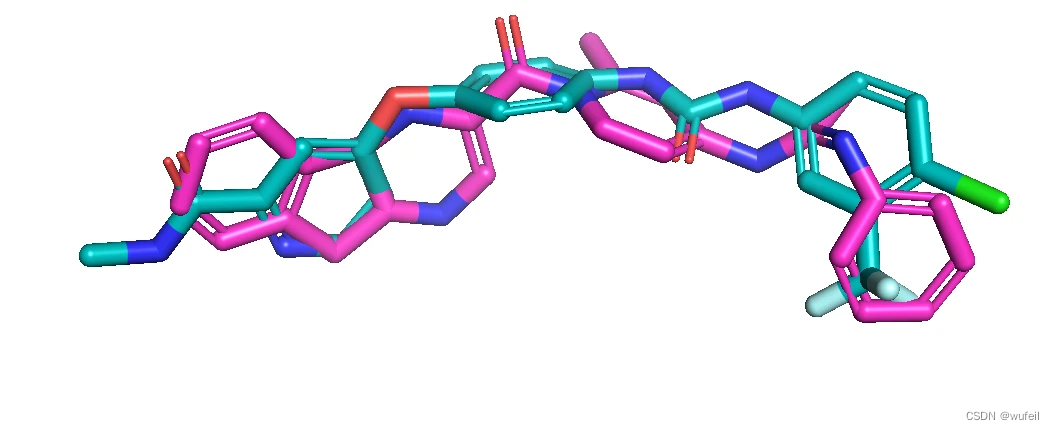
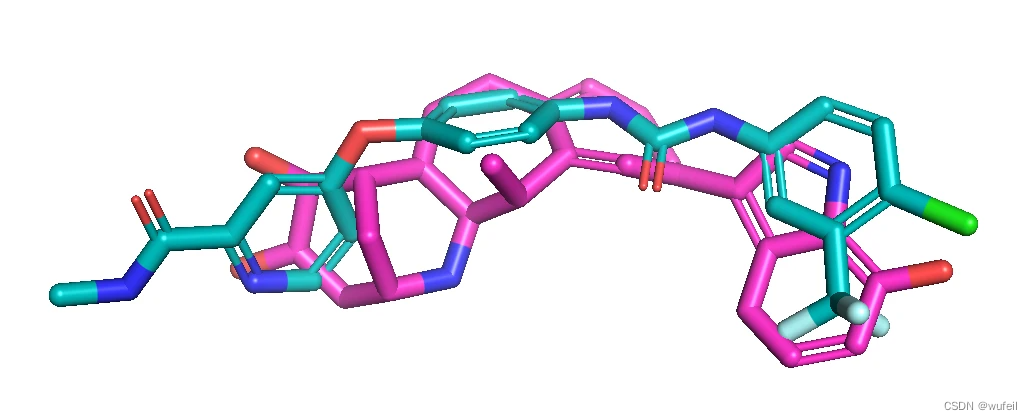
形状相似性最好的三个分子分别如下图(蓝绿色为参考分子,紫色为PMDM生成分子),对应的形状相似性打分为0.772,0.772,0.741。形状上,PMDM生成的分子基本与参考分子叠合,非常相似。这一点要优于其他之前测试过的分子生成模型。


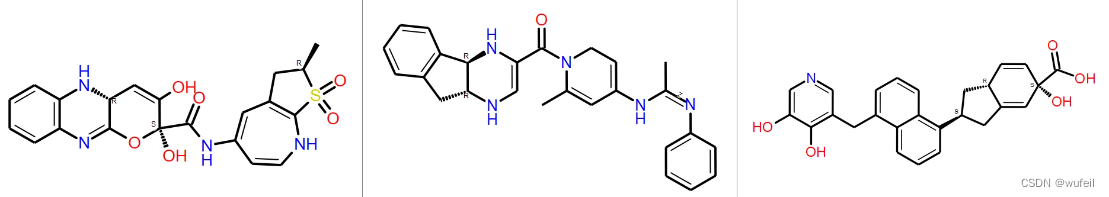
对应的2D结构:
3.2.3 药效团相似性
药效团相似性最好的三个分子,如下图,对应的药效团相似性分别为:0.536,0.506,0.492。药效团上,PMDM生成的分子与真实分子的motif差距很大,几乎不相似。
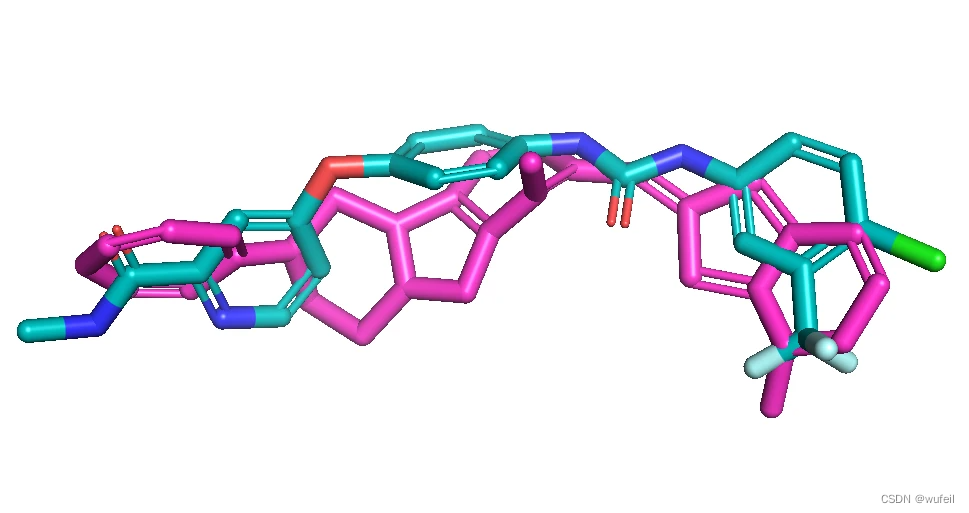
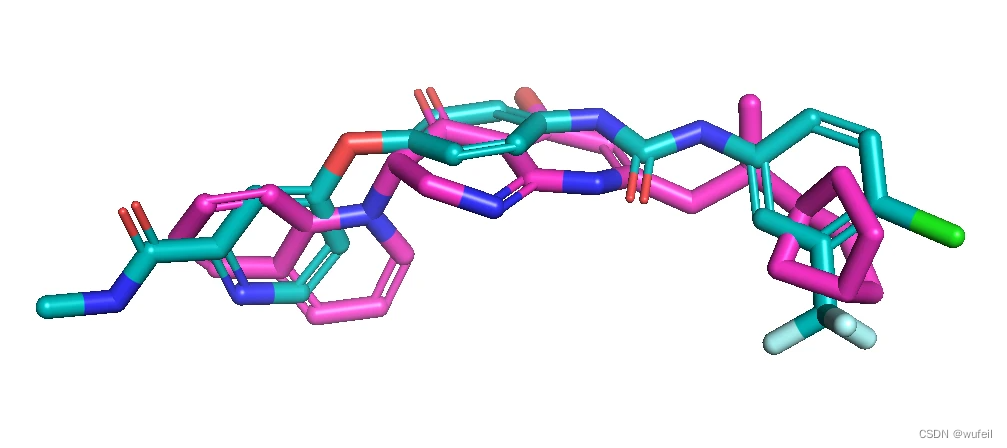
对应的2D结构为:
3.2.4 2D结构相似性
参考分子的2D结构为:
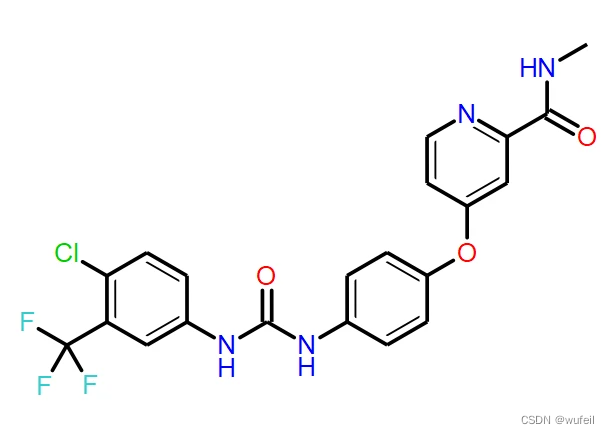
与参考分子2D结构最相似的3个分子分别见下图,从左至右,相似度分别为:0.062,0.047,0.047。基本与参考分子不相似,新颖性较高。
3.2.5 对接打分
将这些生成的分子,对接到3WZE的口袋上,打分最好的三个分子如下图。打分分别为:-11.3, -10.88, -10.41
3.3 片段延伸
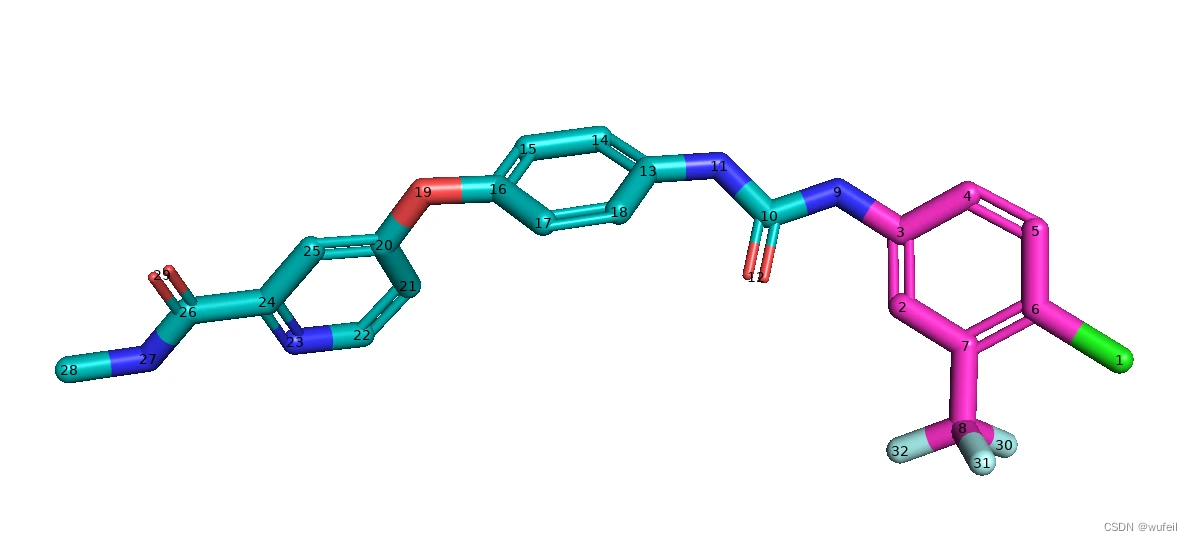
还是以3WZE为例,尝试让PMDM生成右侧的苯环以及CF3以及Cl取代,如下图紫色部分:
确认小分子中原子编号,运行如下代码:
from rdkit import Chem
from rdkit.Chem import Draw
mol = Chem.SDMolSupplier('3wze-mol.sdf')[0]
smiles = Chem.MolToSmiles(mol)
print(smiles)
mol.RemoveAllConformers()
for i, atom in enumerate(mol.GetAtoms()):atom.SetProp('molAtomMapNumber', str(i+1))
Draw.MolToImage(mol, size=(1000,1000))输出:
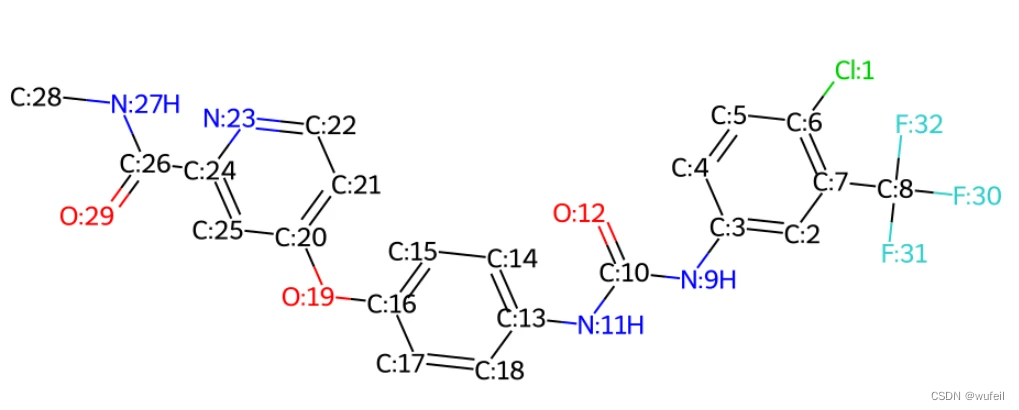
因此,keep_index 为:9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29
使用PMDM生成片段延伸分子命令:
python -u sample_frag.py \--ckpt 500.pt \--pdb_path 3WZE/pocket.pdb \--mol_file 3WZE/3wze-mol.sdf \--keep_index 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 \--num_atom 11 \--num_samples 100 \--batch_size 32 \--sampling_type generalized示例输出:
sample: 1000it [01:35, 10.44it/s]████████████████████████████████████████████████████████████████████████▋ | 2/3 [03:11<01:35, 95.86s/it]
generated smile: [C][N]C(=O)c1[c]c(Oc2[c][c]c([N][C]=O)[c][c]2)[c][c]n1.[F].[O]C(=O)[C]c1[c][c]c([O])[c][c]1
largest generated smile part: [C][N]C(=O)c1[c]c(Oc2[c][c]c([N][C]=O)[c][c]2)[c][c]n1
Generate SA score: 0.32
Generate QED score: 0.759074382854598
pock_35.sdf
[2024-04-08 02:30:21,340::test::INFO] Successfully generate molecule for pock, remining 35 samples generated
generated smile: [C][N]C(=O)c1[c]c(Oc2[c][c]c([N][C]=O)[c][c]2)[c][c]n1.[F].[O][C]1[C][C]([O])C([O])([O])[C][C]1[O]
largest generated smile part: [C][N]C(=O)c1[c]c(Oc2[c][c]c([N][C]=O)[c][c]2)[c][c]n1
Generate SA score: 0.32
Generate QED score: 0.759074382854598
pock_34.sdfgenerated smile显示,生成的分子并不是完全连接的,而是多个分子片段组成,例如下图:
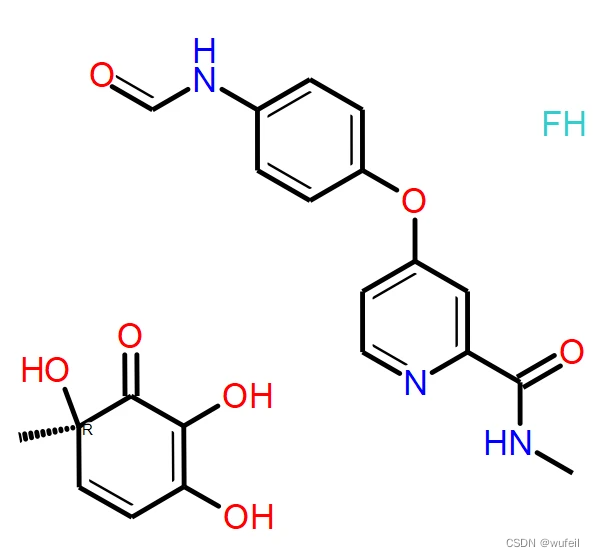
PMDM对生成的分子进行了处理,仅保留最大分子片段。生成的分子在3WZE/frag_gen路径。经查看,发现并没有生成任何新的分子,PMDM生成的所有sdf文件均为原来的seed frag。第一次尝试失败了。
进一步尝试,缩小PMDM生成片段的范围,缩小至CF3的区域,如下图:
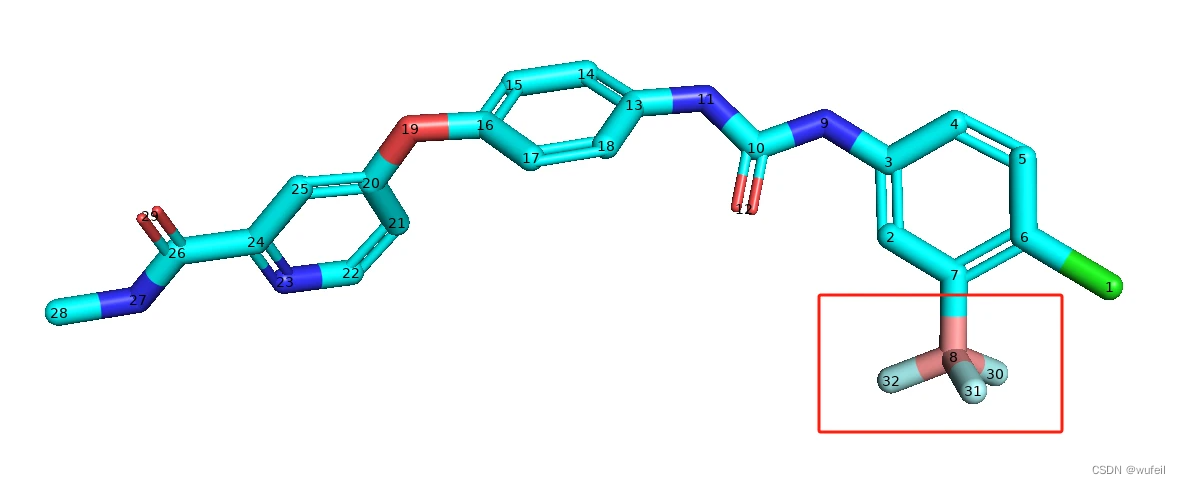
运行代码:
python -u sample_frag.py \--ckpt 500.pt \--pdb_path 3WZE/pocket.pdb \--mol_file 3WZE/3wze-mol.sdf \--keep_index 1 2 3 4 5 6 7 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 \--num_atom 4 \--num_samples 100 \--batch_size 32 \--sampling_type generalized结果并未产生新的分子。而且把Cl接在了CF3的C上。
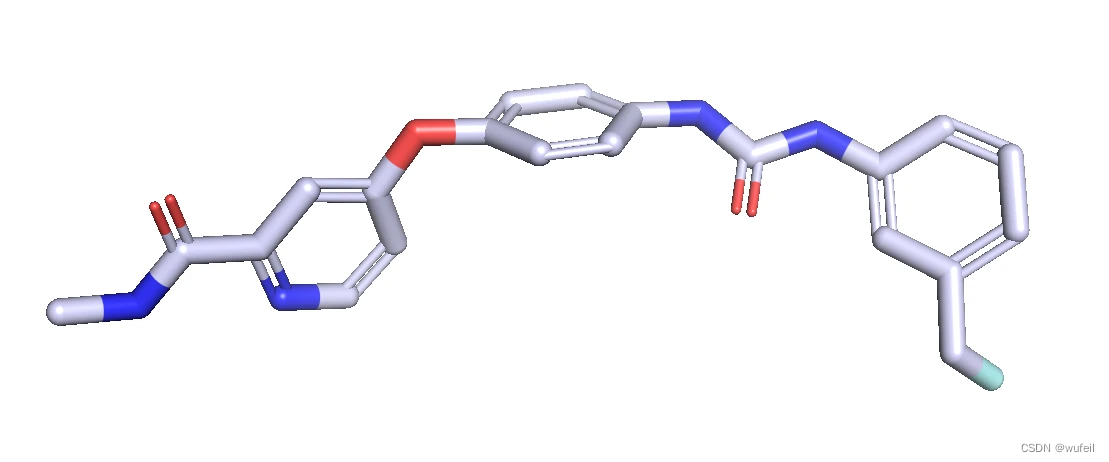
因此,在这个3WZE测试体系上,PMDM并不能进行分子延展任务。
此外,进行了保留的分子片段较小的测试,命令如下:
python -u sample_frag.py \--ckpt 500.pt \--pdb_path 3WZE/pocket.pdb \--mol_file 3WZE/3wze-mol.sdf \--keep_index 9 10 11 12 13 14 15 16 17 18 19 \--num_atom 31 \--num_samples 100 \--batch_size 32 \--sampling_type generalized输出示例:
[2024-04-10 02:20:58,201::test::INFO] Successfully generate molecule for pock, remining 6 samples generated
generated smile: C1CCC1.COc1ccc(NC=O)cc1
largest generated smile part: COc1ccc(NC=O)cc1
Generate SA score: 0.92
Generate QED score: 0.6603167590818222
pock_5.sdf
[2024-04-10 02:20:58,204::test::INFO] Successfully generate molecule for pock, remining 5 samples generated
generated smile: CCCO.COc1ccc(NC=O)cc1
largest generated smile part: COc1ccc(NC=O)cc1
Generate SA score: 0.92
Generate QED score: 0.6603167590818222
pock_4.sdf也不行,生成的分子是不完整,最后只保存了最大的分子片段。生成的分子,不知道为什么不在口袋内,虽然已经输入了seed片段,如下视频:
PMDM-3WZE_Frag
3.4 linker生成
计划是对以下下红色部分的区域进行linker生成。
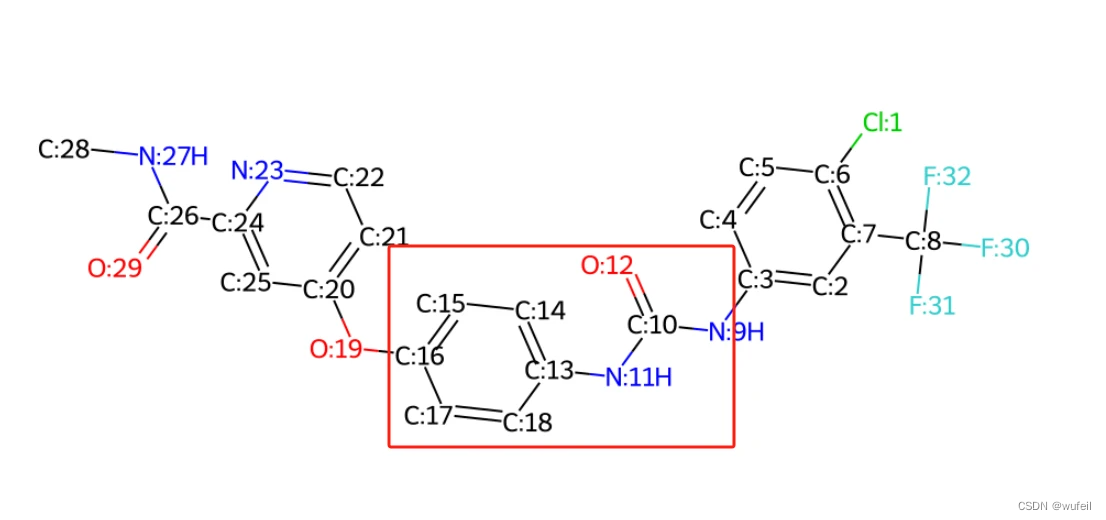
因此,设置的mask为: 9 10 11 12 13 14 15 16 17 18
linker生成命令:
python -u sample_linker.py \--ckpt 500.pt \--pdb_path 3WZE/pocket.pdb \--mol_file 3WZE/3wze-mol.sdf \--mask 9 10 11 12 13 14 15 16 17 18 \--num_atom 4 \--num_samples 100 \--sampling_type generalized \--batch_size 32运行示例输出:
[2024-04-10 01:43:28,082::test::INFO] Successfully generate molecule for pock, remining 52 samples generated
generated smile: [C][N]C(=O)c1[c]c(Oc2[c][c]c([C][C][N]c3[c][c]c(Cl)c(C(F)(F)F)[c]3)[c][c]2)[c][c]n1
largest generated smile part: [C][N]C(=O)c1[c]c(Oc2[c][c]c([C][C][N]c3[c][c]c(Cl)c(C(F)(F)F)[c]3)[c][c]2)[c][c]n1
Generate SA score: 0.32
Generate QED score: 0.5777521286060957
Generate logP: 3.242470000000001
Generate Lipinski: 5
[b'1 molecule converted\n', b'9 molecules converted\n']
Best affinity: -10.8
Generate vina score: -10.8
-10.8_pock_51.sdf
[2024-04-10 01:43:32,610::test::INFO] Successfully generate molecule for pock, remining 51 samples generated
generated smile: [C][N]C(=O)C1=NC(=O)[C]C(Oc2[c][c]c([C])[c][c]2)=[C]1.[N]c1[c][c]c(Cl)c(C(F)(F)F)[c]1
largest generated smile part: [C][N]C(=O)C1=NC(=O)[C]C(Oc2[c][c]c([C])[c][c]2)=[C]1
Generate SA score: 0.35
Generate QED score: 0.7278698579967423
Generate logP: -0.37148000000000014
Generate Lipinski: 5
[b'1 molecule converted\n', b'9 molecules converted\n']
Best affinity: -7.9
Generate vina score: -7.9
-7.9_pock_50.sdf当然,有一些生成的linker是不正确的。所以会出现Failed to kekulize aromatic bonds in OBMol等报错,这是正常的。
设置让PMDM生成100个新的Linker的分子,产生了200个sdf文件。小分子与口袋的位置见以下视频。
PMDM_3WZE_case_Linker
小分子几乎没有与口袋发生冲突,效果还是不错的。
但是有大量的小分子片段,是由于PMDM没有正确连接两端的片段,导致生成的分子断开,被保存成两个sdf文件,于是有200个。
同时,虽然指定了生成的linker的原子数量为4,但是PMDM会自动扩大为3~6的原子数量,在linker区域放不下原子时,会在其他区域生长原子,导致有其他侧链的出现,甚至导致原来的fragments发生结构重排。
PMDM linker生成的分子,进行原子数量<25过滤,剩下的分子有123个,2D结构见下图示例:
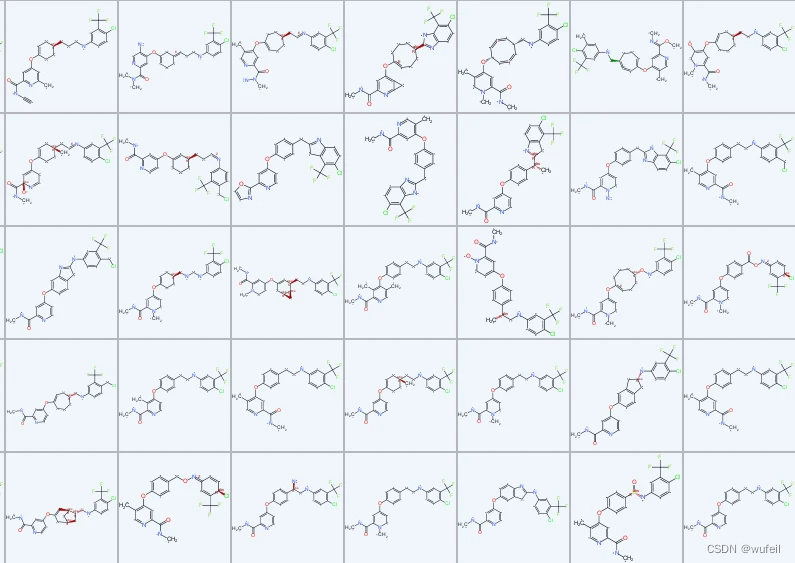
当设置生成linker的范围更小一些的时候,结果会好一些。以下为linker(mask)区域为9 10 11 12的结果。生成的分子如下视频:
PMDM_3WZE_case_Linker_2
linker区域设置小了,效果好一些。
注:虽然生成linker的新分子,但是这些分子均无法对接。
四、总结
PMDM是一种新的3D分子生成扩散模型,利用二元扩散网络,构造了两种类型的虚拟边,增强了模型关于原子间相互作用的识别能力,生成分子与寇弟啊亲和力更强,类药性更佳。
作者利用PMDM在实际的CDK2等体系上做了验证,并对生成的分子进行合成,活性测试,取得不错的效果。
在此次测试中:
以3WZE为例子,测试了PMDM的分子生成,片段生长,linker生成能力。
在分子生成中,PMDM成功生成了分子,并且和口袋并没有明显的clash。生成的分子与之前的模型相比,确实更合理一些。
在片段生长中,并没有成功的生成分子。
在linker生成中,大量的生成的linker无法成功的连接两侧的分子片段。
部分成功连接两侧片段的分子,也会导致两侧片段的拓扑结构重排。linker生成的分子需要认真的挑选才可以用。
在测评中,PMDM的结果比较差的原因可能是体系的原因,也可能是作者提供的额、checkpoint不是训练收敛以后的。本想着重新训练模型的,但是当时作者没有提供评估部分的代码,所以就算了。
注:PMDM的代码还不是非常完善,但是,作者非常热心,也在不断地根据大家的反馈修正代码,指导PMDM的使用。