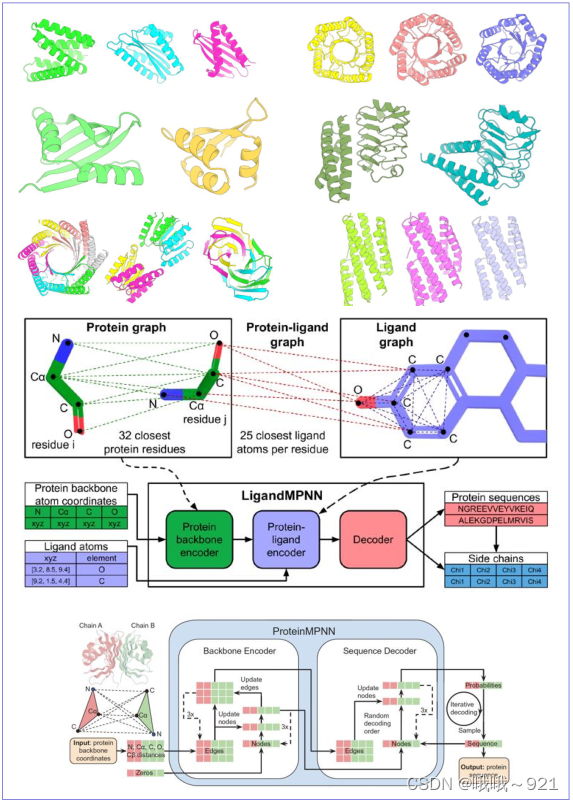
在生物信息学、合成生物学、药物研发、生物材料制备等领域,深度学习技术的革新正在重塑蛋白质设计工具的边界,使其从单纯的结构预测跃升至具有创造性的设计层面。RFdiffusion作为美国华盛顿大学David Baker教授团队的最新科研成果,凭借其卓越性能与开创性功能,标志着蛋白质设计步入全新阶段。
RoseTTAFold,作为David Baker教授团队早期开发的蛋白质结构预测工具,在学术界与工业界广受认可。然而,随着时间推移,David Baker教授团队敏锐洞察到,仅局限于预测已知结构的蛋白质并不能满足生物医药和生物工程领域对创新设计的需求。这促使David Baker教授团队继续深入研究突破。在此背景下,RFdiffusion应运而生,继承了RoseTTAFold的优良基因,但在功能上实现了质的飞跃,不仅能够预测蛋白质结构,更能够从无到有地进行创新设计。这种“从零开始”的设计理念赋予科学家们前所未有的设计自由度,这一突破不仅标志着蛋白质设计技术的重大进步,也意味着科研人员可以更深入地探究生命现象的本质,甚至创造出全新的生物功能和应用。
2023年7月11日,David Baker团队在国际顶级期刊《Nature》上发表了题为《De novo design of protein structure and function with RFdiffusion》的论文,全面揭示了RFdiffusion在蛋白质设计领域的核心竞争力,这篇论文的发表,无疑将蛋白质设计工具推向了一个全新的高度,昭示着未来蛋白质设计将更加注重创新性和实用性,以适应并推动生物医学、材料科学、生物信息学、合成生物学等多学科领域的前沿研究。
本次会议是在深度学习驱动蛋白质设计这一新兴领域快速发展的背景下举办,旨在系统介绍和深入探讨RFdiffusion与ProteinMPNN等前沿深度学习技术的原理、应用和最新研究成果,促进相关领域的科研人员、工程师及产业界人士对该领域的理解与技术掌握,推动深度学习技术在蛋白质设计领域的创新应用与实际转化。
适合生物信息学、结构生物学、合成生物学、药物研发、生物材料等相关领域的科研人员;生物医药、生物科技企业研发、技术部门负责人、对深度学习驱动蛋白质设计感兴趣的跨界人士
深度学习驱动的蛋白质设计技术与前沿实践-从基础到尖端应用
RFdiffusion 创新结构生成技术
一、背景介绍
1、从蛋白质预测到蛋白质设计
2、结构生成过程中的物理能量函数与约束
3、基于Deep learning的预测模型和生成模型
4、结构验证与性能评估
二、RFdiffusion基于指定骨架的蛋白质结构设计
核心知识点:利用用户提供的特定结构框架进行蛋白质结构设计
应用案例:
1、无约束单体设计(contigmap):全新骨架的蛋白质结构创新设计,通过RFdiffusion实现从头生成新颖、非同源蛋白质结构
2、特定骨架引导设计 (scaffoldguided):利用已有结构骨架指导蛋白质结构创新与改造
RFdiffusion 高级应用及领域热点
三、RFdiffusion含活性位点的蛋白质结构设计
核心知识点:使用RFdiffusion构建具有特定生物活性的蛋白质结构
应用案例:
1、Motifscaffold: inference.input_pdb & contigmap.inpaint_seq:如何整合已知活性位点信息,嫁接到设计的新蛋白质结构上
实例分析:酶活位点嫁接,对称位点嫁接的设计策略与实施
四、蛋白质-蛋白质相互作用界面(PPI):Binder设计
核心知识点:RFdiffusion设计能有效介导蛋白间相互作用的Binder区域
应用案例:
1、ppi.hotspots:识别和利用PPI热点信息来定制Binder结构
2、设计实例:针对指定骨架和/或特定motif的Binder结构创建
五、RFdiffusion中的对称性设计
核心知识点:阐述如何在蛋白质结构设计中考虑并实现对称性特征
应用案例:
1、Inference.symmetry: 对称性建模与控制
2、potentials:优化对称单元间的能量分布与稳定性
六、多样性
核心知识点:探究RFdiffusion在处理结构多样性上的机制和优势
应用案例:diffuser.partial_T:局部扩散与全局优化结合,提高结构多样性和稳健性、RFdiffusion中的noise
ProteinMPNNLigandMPNN 序列设计基础与高级应用
七、ProteinMPNN序列设计入门
核心知识点:介绍ProteinMPNN的核心架构与训练过程,掌握ProteinMPNN和LigandMPNN在不同场景下的蛋白质结构序列设计流程
应用案例:
1、Monomer单体序列设计:用ProteinMPNN针对单体蛋白质进行序列设计
2、Complex复合物zhong 指定链设计:利用ProteinMPNN在复合物环境下设计特定链的序列,涉及链间相互作用的考虑与优化。
Models, Helper scripts, number of sequences:深入了解模型使用方法、辅助脚本的功能以及决定生成序列数量的因素
ProteinMPNN和LigandMPNN 序列设计基础与高级应用
八、指定设计位点与氨基酸偏好性设置
核心知识点:在蛋白质设计中如何运用深度学习工具来精准控制特定氨基酸残基的位置与特性
应用案例:
1、Fix position与Redesign position:保留某些部位不变(Fix position)和重新设计其他部位(Redesign position)的具体操作。
Bias AA与Omit AA:在设计过程中如何设置氨基酸偏好性,包括优先选择某些氨基酸(Bias AA)和排除特定氨基酸(Omit AA),以满足特定功能需求或生物物理化学特性。
RFdiffusion与ProteinMPNN 高级应用案例分析
九、MPNN进阶应用
核心知识点:MPNN对称性处理与同聚多体设计、MPNN设计多样性与温度参数调控
应用案例:
1、MPNN处理对称性Symmetry:讲解MPNN如何识别和处理蛋白质的对称性特征,特别是对于Homooligomers(同聚多体)的设计。
2、Tied position(绑定位置):探讨MPNN在处理需要保持多个位置协同变化以维持特定对称模式的情况下的具体方法。
MPNN设计多样性:探究MPNN如何通过温度参数调整来促进设计序列的多样性,以及多样性对最终蛋白质功能和稳定性的影响。
RFdiffusion与ProteinMPNN 高级应用案例分析
十、Colabfold 结构预测与深度学习应用
1、MSA、pLDDT和pAE:介绍多序列比对(Multiple Sequence Alignment)、预测精度得分(predicted Local Distance Difference Test, pLDDT)和原子接触误差(predicted Atomic Error, pAE)在蛋白质结构预测中的重要作用。
2、逆转网络与幻想蛋白设计
探讨如何通过深度学习技术逆向设计已知结构或创造新型蛋白质结构的可能性。
互动讨论
1、分享与解析近期领域内热点研究成果中RFdiffusion + ProteinMPNN的实际应用案例
2、讨论并解决实际操作中可能遇到的问题与挑战,进一步提升学员对深度学习在蛋白质结构设计领域中的理解和应用能力
最新顶刊文献中对应的该课程内深度学习蛋白质结构设计技术
RoseTTAFold Diffusion
De novo design of protein structure and function with RFdiffusion. Nature. 2023, 620(7976):1089-1100
De novo design of high-affinity binders of bioactive helical peptides. Nature. 2024, 626(7998):435-442 (多肽Binder)
Computational design of sequence-specific DNA-binding proteins. bioRxiv. 2023.09.20.558720 (核酸Binder)
Atomically accurate de novo design of single-domain antibodies. bioRxiv. 2024.03.14.585103v1
ProteinMPNN
Robust deep learning–based protein sequence design using ProteinMPNN. Science. 2022, 378(6615):49-56.
Improving Protein Expression, Stability, and Function with ProteinMPNN. J Am Chem Soc. 2024, 146(3):2054-2061.
LigandMPNN
Atomic context-conditioned protein sequence design using LigandMPNN. bioRxiv. 2023.12.22.573103v1
RoseTTAFold and RFDesign
Accurate prediction of protein structures and interactions using a three-track neural network. Science. 2021, 373 (6557) : 871-876.
Efficient and accurate prediction of protein structure using RoseTTAFold2. bioRxiv. 2023.05.24.542179v1
Accurate prediction of protein–nucleic acid complexes using RoseTTAFoldNA. Nat Methods. 2024, 21(1):117-121.
Generalized biomolecular modeling and design with RoseTTAFold All-Atom. Science. 2024, eadl2528.
Scaffolding protein functional sites using deep learning. Science. 2022, 377(6604):387-394.
trRosetta and trDesign
Improved protein structure prediction using predicted interresidue orientations. Proc Natl Acad Sci U S A. 2020, 117 (3) : 1496-1503.
Protein sequence design by conformational landscape optimization. Proc Natl Acad Sci U S A. 2021, 118(11):e2017228118.
De novo design of small beta barrel proteins. Proc Natl Acad Sci U S A. 2023, 120(11):e2207974120.
Colabfold, Alphafold and ESMfold
ColabFold making protein folding accessible to all. Nat Methods. 2022, 19(6):679-682.
Improved protein structure prediction using potentials from deep learning. Nature. 2020, 577(7792):706-710.
Highly accurate protein structure prediction with AlphaFold. Nature. 2021, 596(7873):583-589.
Protein complex prediction with AlphaFold-Multimer. bioRxiv. 2021.10.04.463034v2.
Evolutionary-scale prediction of atomic-level protein structure with a language model. Science. 2023,379(6637):1123-1130.
幻想蛋白
部分案例图示:(见下页)

![Ceph [OSDI‘06]论文阅读笔记](https://img-blog.csdnimg.cn/direct/b15384f67a1243e29d76e63ac627afff.png#pic_center)