DrugBan:一种可解释的双线性注意力网络进行药物-靶点结合预测。
提示:写完文章后,目录可以自动生成,如何生成可参考右边的帮助文档
文章目录
- DrugBan:一种可解释的双线性注意力网络进行药物-靶点结合预测。
- 前言
- 一、模型框架
- 1. 编码器
- 2. 双线性注意力网络(BAN)
- 3. 对抗性的领域适应网络(CDAN)
- 二、结果
- 1. 模型性能
- 2. 消融实验
- 3. 模型解释性
- 总结
前言
预测药物-靶点相互作用(DTI)时药物发现中的关键环节,近年来一些深度学习方法在该环节中显示了广泛的应用前景,但目前仍然存在两个关键问题:
1. 如何明确地建模和学习药物和靶标之间的局部相互作用,以便更好地预测。
2. 如何提升药物-靶标对的预测的泛化能力。
针对这两个问题,作者提出了一个深度双线性关注网络(drug- BAN)框架以学习药物和靶标之间的局部相互作用,并使用领域自适应模块应用到训练集分布以外地数据中(即未知的药物-靶标结构对)。
一、模型框架
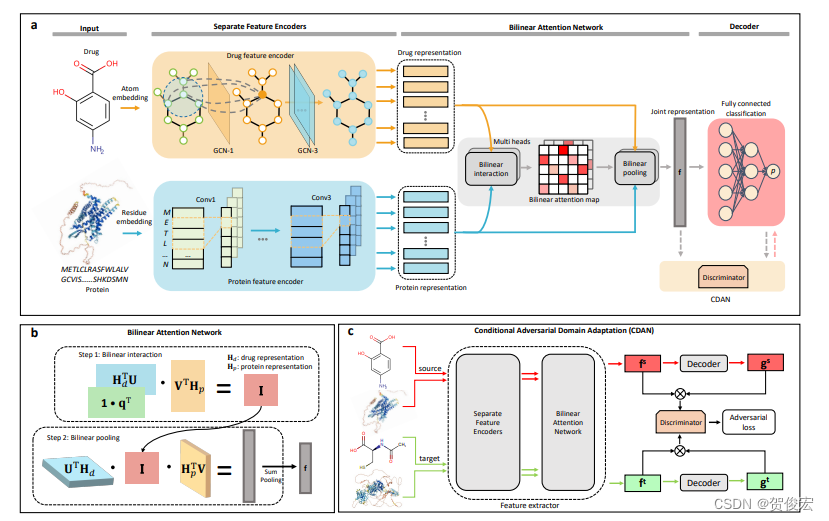
模型由三部分组成:
1. 编码器
基于基于GCN编码分子smiles,CNN编码蛋白序列。
2. 双线性注意力网络(BAN)
为了更好的学习蛋白和配体之间的局部相互作用特征,没有将分子和蛋白特征直接拼合以表示分子-蛋白对。而是加入了一个attenion去生成一个新的集合特征I,之后再进行一层attention的套娃。与直接采用单层attention相比,双线性注意力网络能够更好的去学习两个子特征之间的联系,本质上是一种多模态模型。
3. 对抗性的领域适应网络(CDAN)
为了增强模型的泛化能力,作者在BAN之后接入了一个CDAN。该模块属于迁移学习的一种方式,其核心思维和生成对抗网络很像。具体表现为,当我们已经在source数据集上训练完成了BAN之后,如何去提升BAN模型的泛化能力的让其能够将soutce数据集中的知识应用数据分布有所不同的target数据集上。首先,两个数据集都会通过BAN网络得到特征f,之后经过分类器去进行鉴别。 BAN和这个分类器之间进行了一场”猫捉老鼠“的竞赛,分类器目标是识别出target和source,而BAN网络则是为了蒙混过关。在这个过程中,BAN也就逐渐将source上所学到的知识传到了target上以用于”伪装“。 此外,CADA 还引入了一个条件变量,也就是上图中的g,表示不同的任务场景。在本论文中即蛋白-配体结构对的类别,让BAN能够根据不同的类别以调整权重,更好地适应目标域的任务。
二、结果
1. 模型性能
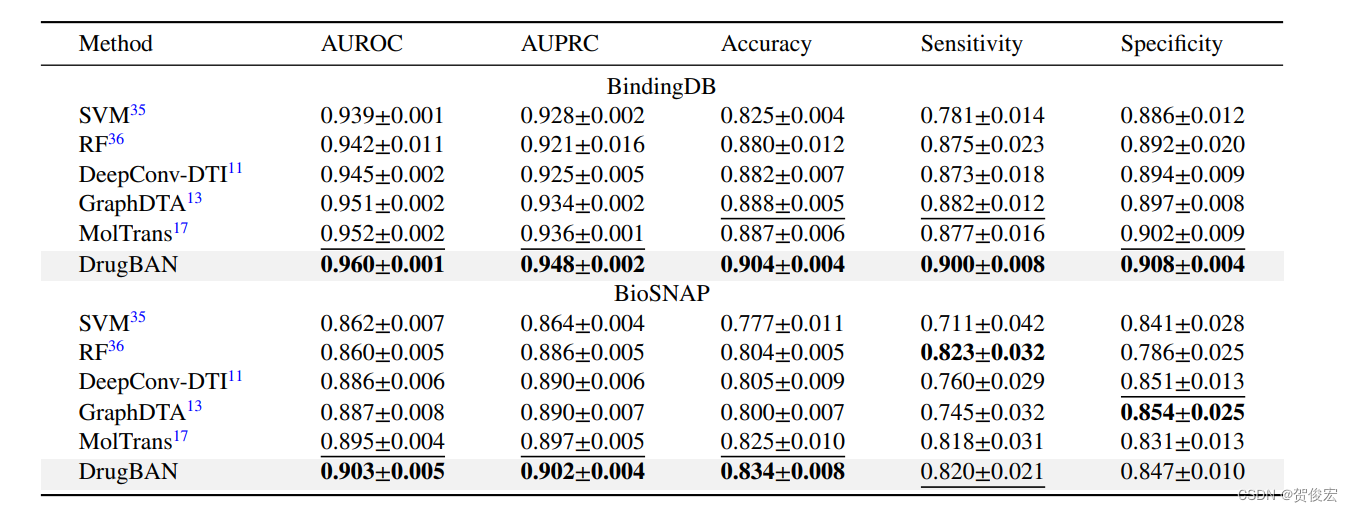
作者一共在两个数据集上进行了相关的性能测试,BindingDB和BioSNAP。并分了两个任务场景,其一是随机批分或据集,第二则是先将数据进行相似性聚类后再批分数据集。前者的性能如下:
后者作者采用了ECFP4指纹和氨基酸序列进行了聚类,并随机抽取了百分之60的类作为sorce训练集,剩下的数据中的百分之80做验证集(target数据集),百分之20做最终的测试集合, 模型表现如下:
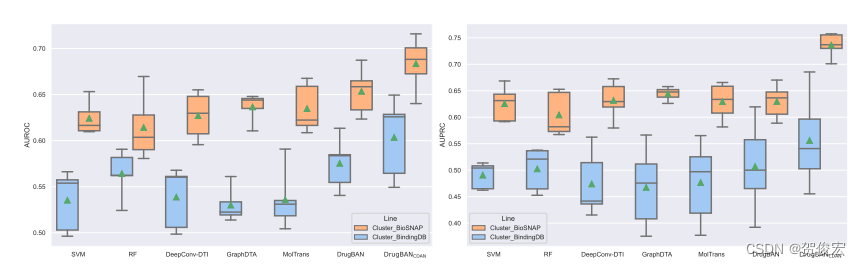
可以看到drug-ban呈现了SOTA性能。
2. 消融实验
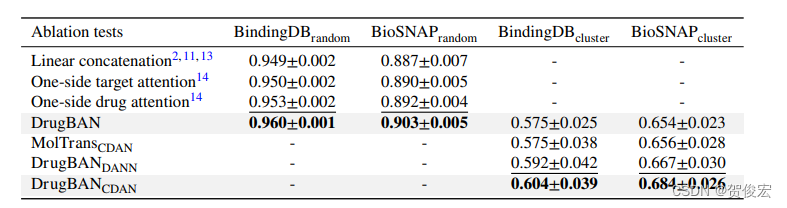
CDAN的引入能够显著替身模型的泛化能力。
3. 模型解释性
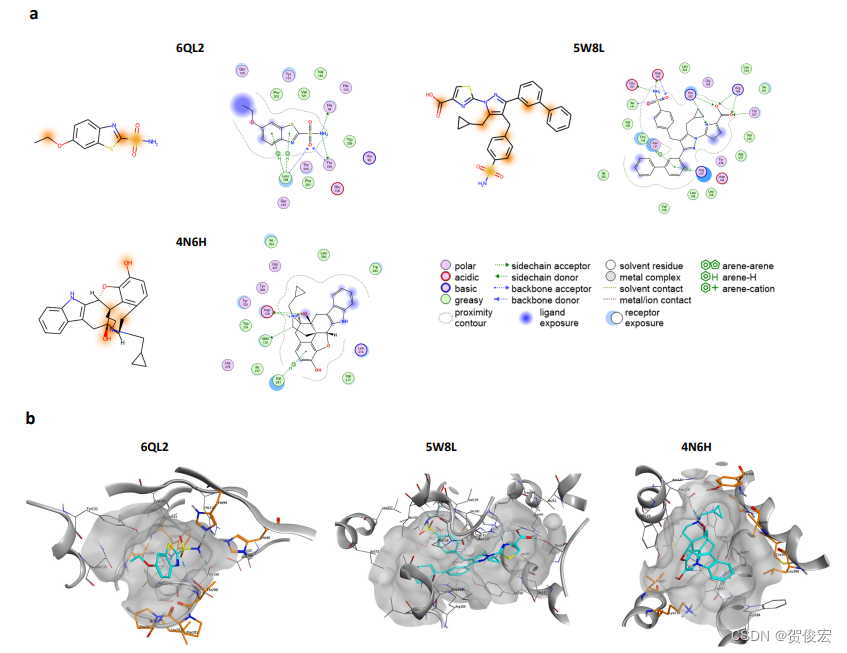
根据模型给出的attention分数丢小分子进行着色,可以发现准确了发现了关键的原子位点。这些原子是配体与蛋白复合物稳定的关键。(但就展示了三个,这个解释性就见仁见智了)
总结
作者提出了 DrugBAN,一个用于 DTI 预测的端到端双线性注意深度学习框架。该模型具备如下三点优势:
- 作者将 CDAN整合到建模过程中,增强了模型的泛化能力。
- 通过将注意力权重映射到蛋白质子序列和药物化合物原子上,作者的模型可以为解释相互作用的性质提供生物学见解。
个人认为阻碍其性能进一步增长的原因:
- 蛋白和分子的表征仅仅包含结构,甚至于蛋白都只有序列信息。特征信息有待扩充:如三维坐标,物理化学性质。
- 看上去似乎是构建了蛋白和配体之间的相互作用,但实际上也就是两个图之间的特征相关性。相互作用其实也是可以作为一种输入表征的。
- 蛋白和配体的结合过程是一个动态拟合过程,依赖固定的2维分子图做预测可能并不够充分。