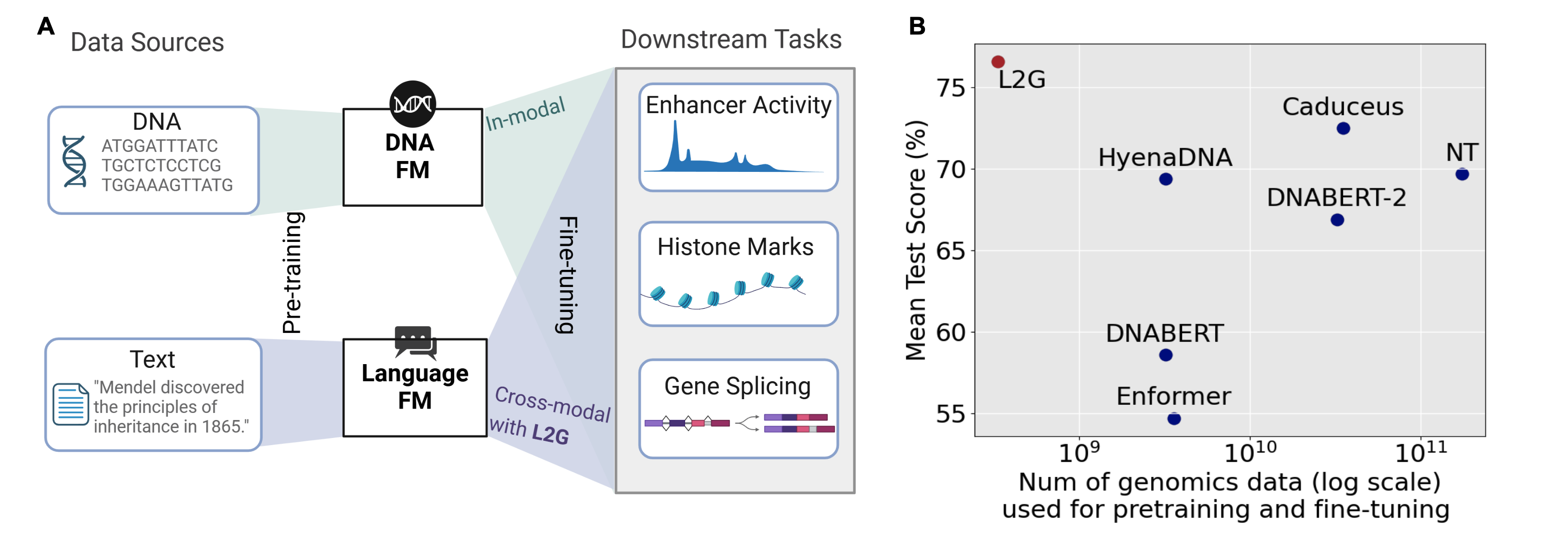
预训练语言模型(如BERT、GPT等)在自然语言处理(Natural Language Processing, NLP)领域取得了显著进展,其在多种语言任务中的表现令人瞩目。这一成功激发了基因组学领域的研究者们尝试开发类似的基础模型(Foundation Models, FMs),以解决复杂的基因组学任务。然而,构建高质量的基因组学基础模型通常需要消耗大量计算资源和高质量的训练数据。
针对这一挑战,来自卡内基梅隆大学的研究团队提出了一种名为L2G(Language-to-Genome)的新型方法,通过跨模态迁移学习将现有的自然语言模型(Large Language Models, LLMs)重新定向用于解决基因组学任务。这一方法避免了从头开始预训练基因组学模型的高成本,同时在多个基准测试中表现优异,展示了跨领域模型迁移的潜力。
背景与研究挑战
基因组学模型的开发通常依赖于对大规模DNA序列数据的无监督预训练,以提取与基因组功能相关的复杂特征。这些模型在预测基因组元素、染色质状态以及基因调控功能等方面显示出了巨大潜力。然而,其高昂的训练成本和对计算资源的依赖成为了研究中的主要障碍。
例如,训练Nucleotide Transformer需要处理约1740亿个token,并在128个NVIDIA A100 GPU上连续训练28天,而较小的模型如DNABERT-2也需要在8个GTX 2080 Ti GPU上训练两周。这种资源密集型的开发过程极大地限制了基因组学基础模型的广泛应用。
与此同时,预训练语言模型的成功为跨模态迁移学习提供了启发。一些研究表明,预训练语言模型在蛋白质属性预测、偏微分方程求解等任务中也表现出色,这表明这些模型具备一定的通用推理能力。L2G正是基于这一观察,通过设计一套高效的迁移和适配机制,将自然语言模型应用于基因组学任务。
L2G框架的核心思想
L2G的核心在于利用跨模态迁移学习,将自然语言模型的强大推理能力迁移到基因组学领域,而无需在DNA序列数据上进行大规模预训练。
模型架构设计
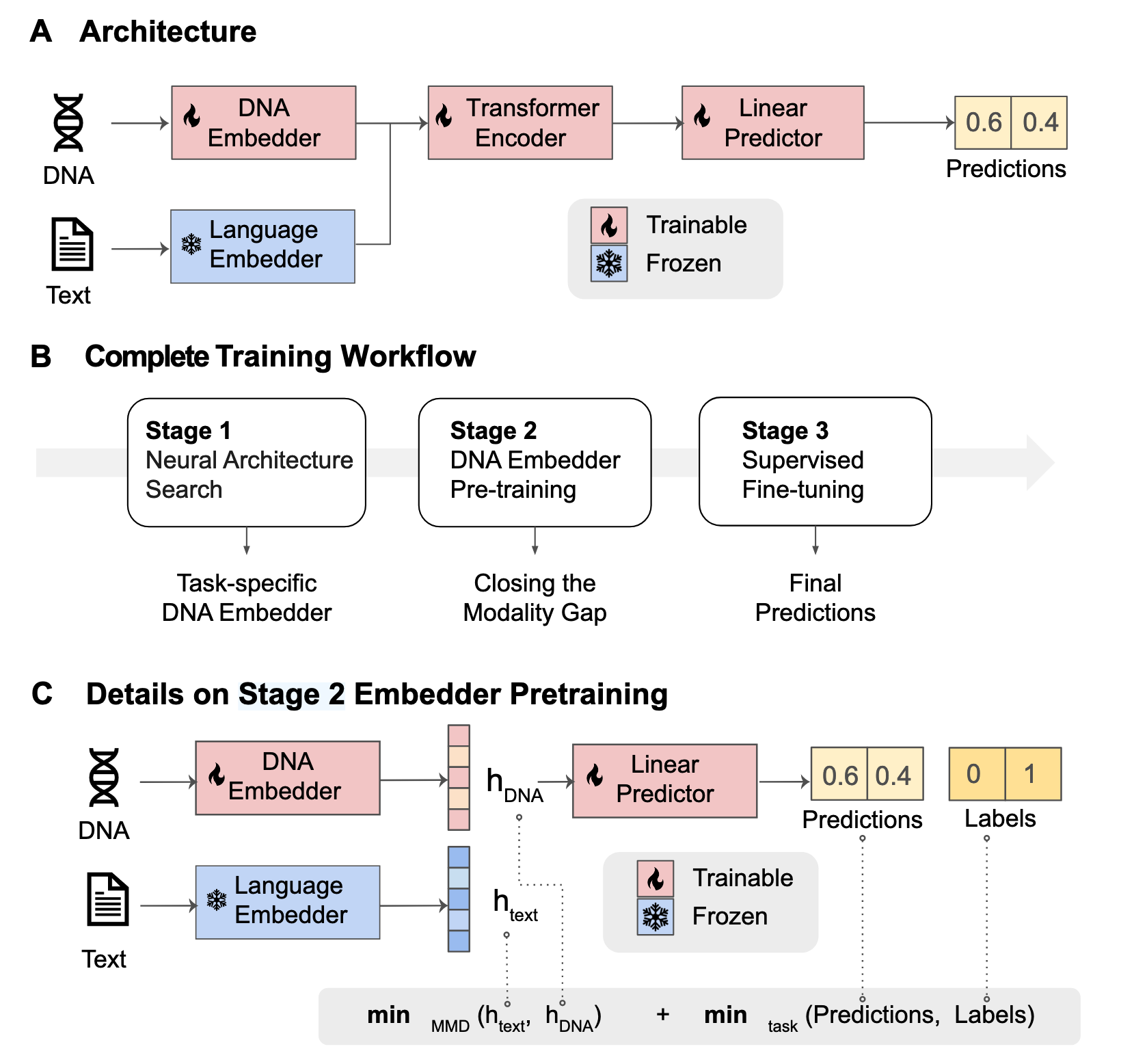
L2G的架构由三部分组成:
- CNN嵌入器:负责将输入的DNA序列数据映射到嵌入空间,以适配自然语言模型的输入格式。
- Transformer编码器:直接复用预训练语言模型的编码器部分,用于从输入嵌入中提取特征。
- 线性预测器:将编码器的输出映射到目标任务的标签空间。
这种模块化设计使得L2G能够充分利用现有语言模型的能力,同时对基因组学任务的需求进行定制化调整。
三阶段训练流程
为了实现高效的跨模态迁移,L2G采用了以下三阶段训练流程:
-
神经架构搜索(NAS)
通过自动化搜索优化CNN嵌入器的架构,选择最适合任务需求的卷积神经网络(如ResNet或UNet),并使用DASH算法调整卷积层的核大小和扩张率。 -
嵌入器预训练
在这一阶段,通过最小化DNA序列嵌入与自然语言嵌入之间的分布差异,同时优化下游任务性能,显著减少两种模态之间的差距。研究使用最大均值差异(Maximum Mean Discrepancy, MMD)作为分布距离度量,并结合特定任务的损失函数进行优化。 -
微调
在目标任务数据上对整个模型进行微调,包括嵌入器、Transformer编码器和线性预测器,以进一步提升任务性能。
实验结果与性能评估
研究团队在多个基因组学基准测试和实际任务中验证了L2G的有效性。结果表明,L2G不仅在计算和数据效率上具有显著优势,还在性能上超越了许多从头训练的基因组学基础模型。
-
Nucleotide Transformer基准测试
在18个任务中,L2G在10个任务中取得最佳成绩,并在其他6个任务中排名第二,尤其是在组蛋白标记和增强子预测任务中表现突出。 -
Genomic Benchmarks数据集
在8个分类任务中,L2G在5个任务中超过了所有其他模型的性能,在其余3个任务中排名第二。 -
增强子活性预测任务
在使用DeepSTARR数据集进行的增强子活性预测中,L2G成功预测了果蝇S2细胞中的发育和管家增强子活性,表现优于专家设计的模型。此外,通过DeepLIFTShap和TF-MoDISco-lite等解释性算法,L2G还识别出了与增强子活性相关的转录因子基序(如AP-1、GATA和SREBP),并揭示了一些独特的基因调控机制。
这些实验结果表明,L2G在不依赖于大规模基因组学预训练的情况下,能够在多种基因组学任务中达到甚至超越领域特定模型的性能。
优势与局限性
优势
- 高效性:L2G显著降低了计算和数据资源需求,所有实验均可在单个A6000 GPU上在数小时内完成。
- 性能优越:在多个基因组学任务中,L2G的表现优于传统的基因组学基础模型,尤其是在增强子活性预测等复杂任务中。
- 跨模态迁移能力:通过跨模态迁移,L2G为自然语言模型在生物学领域的应用提供了新的可能性。
局限性
- 任务覆盖范围有限:当前的评估主要集中在调控元素预测等任务,尚未覆盖基因组学中更复杂的长距离依赖任务。
- 依赖微调:L2G目前依赖于对目标数据的微调,未来可以探索结合无监督DNA数据的继续预训练方法,以进一步提升性能。
结论与展望
L2G通过跨模态迁移学习,将预训练语言模型应用于基因组学任务,展示了高效性和卓越的性能。这一框架不仅减少了计算和数据的需求,还为基因组学研究开辟了新的可能性,引发了对传统预训练方法的反思。未来的研究可以进一步扩展L2G的应用范围,探索其在基因表达预测、染色质三维结构建模等复杂任务中的表现。此外,结合无监督文本和基因组数据的继续预训练方法,也有望进一步提升模型的性能和泛化能力。
参考
- 参考文献:Cheng, W., Shen, J., Khodak, M., Ma, J., & Talwalkar, A. (2024). L2G: Repurposing Language Models for Genomics Tasks. bioRxiv. doi: https://doi.org/10.1101/2024.12.09.627422
- 代码:https://github.com/wenduocheng/L2G
- 数据集:研究中使用了多个公共数据集,包括Genomic Benchmark、Nucleotide Transformer benchmarks和DeepSTARR数据集,这些数据集均可在相应的网站上获取。