API
API 这个词在大多数人看来可能和 CNS 差不多,前者天天听说就是用不上,后者天天读就是发不了。
不过,通过今天的一个简短介绍,今后 API 这个东西你就用上了,因为在文章最后我将会展示一个最最基础且高频的 API 使用示例。
所谓 API(Application Programming Interface) 就是应用程序接口。这个应用程序可以类比于手机和电脑,这个接口就类似于数据线,如果我们想用数据线把东西在两个设备之前传输,就需要两者可以接受彼此的传输协议。如我们在电子商务中,经常就会用到的电商API即商品详情关键字搜索等封装了商品详情介绍,主图,商品价格,商品SKU数据等API数据接口,以获取商品信息。
对一个应用程序来说,如果你想快速的和它交换数据,也需要采用应用程序可以识别的规则。对于一些成熟的应用程序和网站来说,通常都会提供自己的一套API供开发者友好的使用,一方面可以大大扩展应用程序的生态环境,另一方面也可以避免暴力的爬虫。
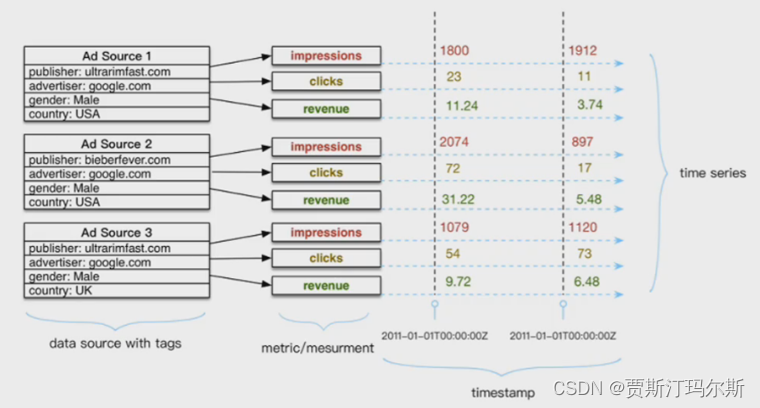
我们通过下图的「印象笔记 API」为例,如果你使用过印象笔记(Evernote)的话,会发现大量第三方应用都可以支持绑定印象笔记账户进行各种创建笔记推送笔记等操作。这里不是印象笔记去适配第三方应用,而是它提供了一个完善的 API,可以供其他开发者来使用。
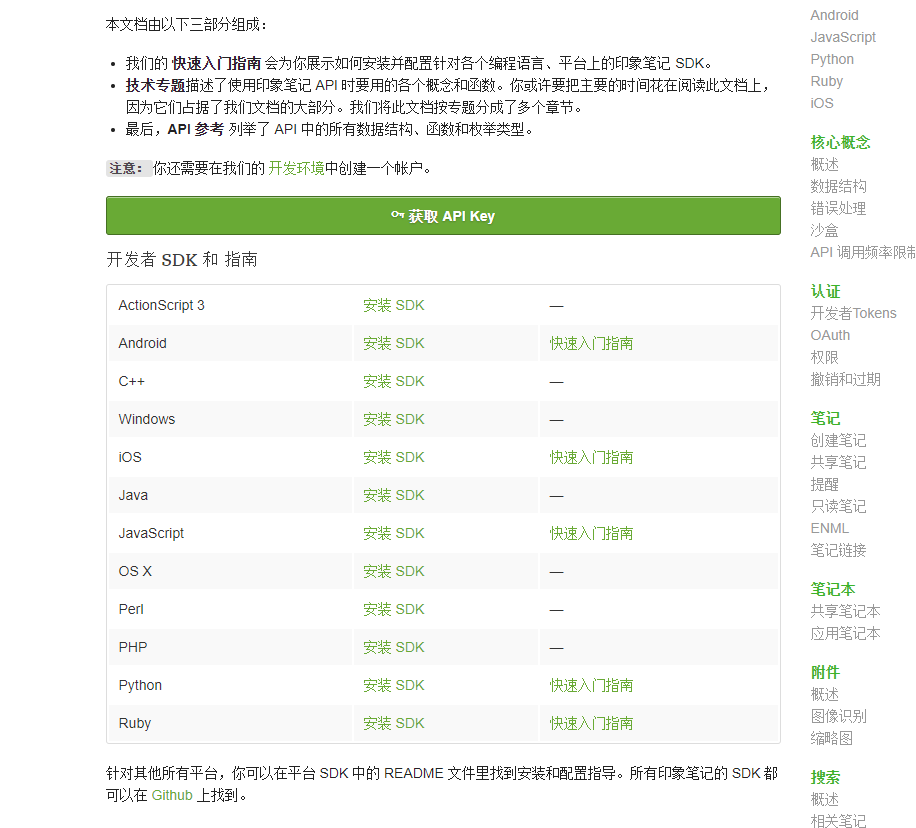
REST API
API 之后还有一个概念是 REST API。 REST(Representational State Transfer) 的中文翻译一般是「表现层状态转化」(这也太抽象了吧),这个架构是 Roy Thomas Fielding 在2000年的毕业论文里提到的,他同时还是HTTP协议(1.0版和1.1版)的主要设计者、Apache服务器软件的作者之一、Apache基金会的第一任主席。
关于究竟什么是「表现层状态转化」这里就跳过了,因为不跳过你也不会看(我也解释不清楚)。只需要了解,符合 REST 设计风格的 Web API 称为 RESTful API,有如下几个要点:
-
资源地址:URI,比如:
http://example.com/resources。 -
传输的资源:Web服务接受与返回的互联网媒体类型,比如JSON,XML,YAML等。
-
对资源的操作:Web服务在该资源上所支持的一系列请求方法,比如:POST,GET,PUT 或 DELETE。
常用数据库 API
NCBI
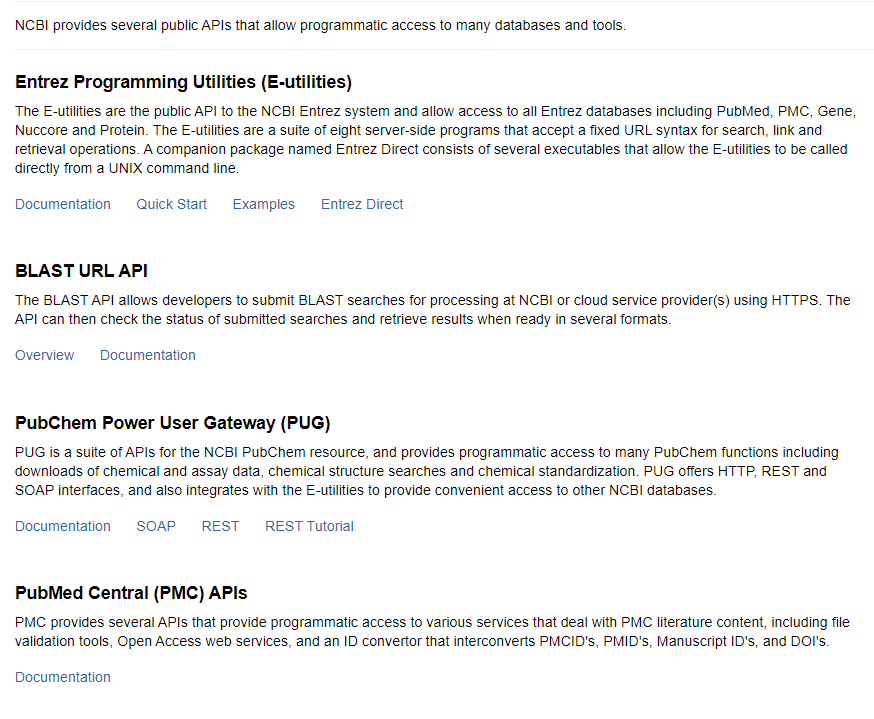
NCBI 是啥就不介绍了,如果太多的功能没用过 PubMed 应该怎么也用过。在 NCBI 的开发文档中,有一个部分专门是讲 API 的。
NCBI 提供的 API 如下图所示,如果需要可以去看看。
EMBL-EBI
EMBL-EBI 来自于欧洲,里面有很多很多数据库都是我们日常会使用的,只是你可以还不知道它们和 EBI 有关系的,例如存放了大量基因组及相关数据的 Ensembl ,包括大量蛋白序列和功能信息的数据库 UniPort,当然还有还有不逊色与 pubmed 的文献数据库 Europe PMC。
从个人的使用体验来说,一般能用EBI的时候我就尽量会绕过NCBI,因为整个一系列网站用起来都要更舒服些,文档查起来更顺手些,对一些有进阶需求的开发者会更友好些(如果你有二次开发的需求,经过对比不难理解我的感受)。
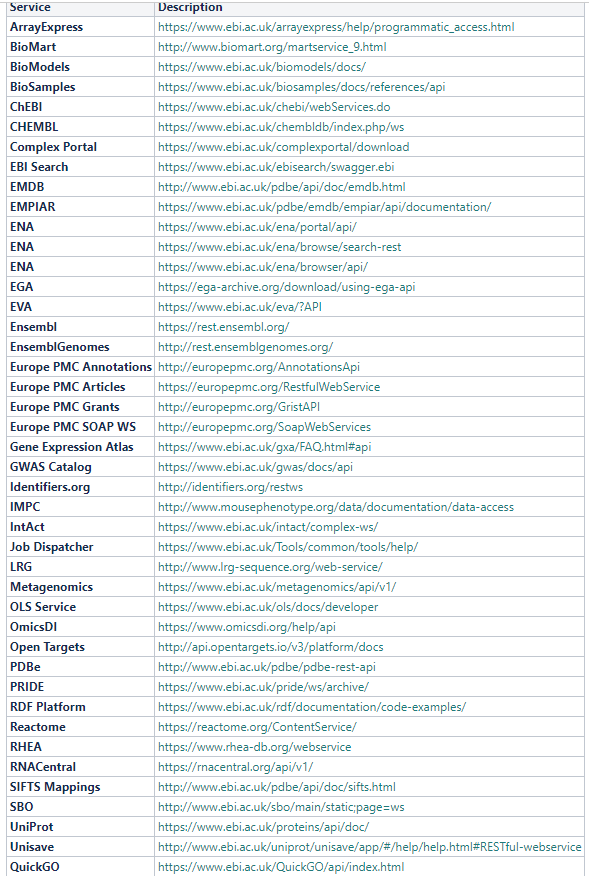
目前 EBI 比较知名的几个数据库都有很不错的 RESTful API 支持。
其中 Ensembl 支持 21 个 POST 和 98 个 GET 操作,可以在官方说明中查看;Uniport 数据库 和 Europe PMC 也有大量的操作支持。通过这些 API 你就可以接触到数据库中有的所有信息和 33 million 的文献。另外,EBI 还有一个 QuickGO 的网站也支持 RESTful API 。
他们的 API 完善到直接在 NAR 发了一篇文章。

可用的数据库和工具如下
使用 API
这里以 Ensembl 的一个基础 API 为例对使用方法进行简单的演示。如果我们在 Ensembl 的网站上查看一个基因,会是如下页面。在左侧我圈出来的是和这个基因相关的所有信息,其中99%的信息都可以通过 API 获取到。
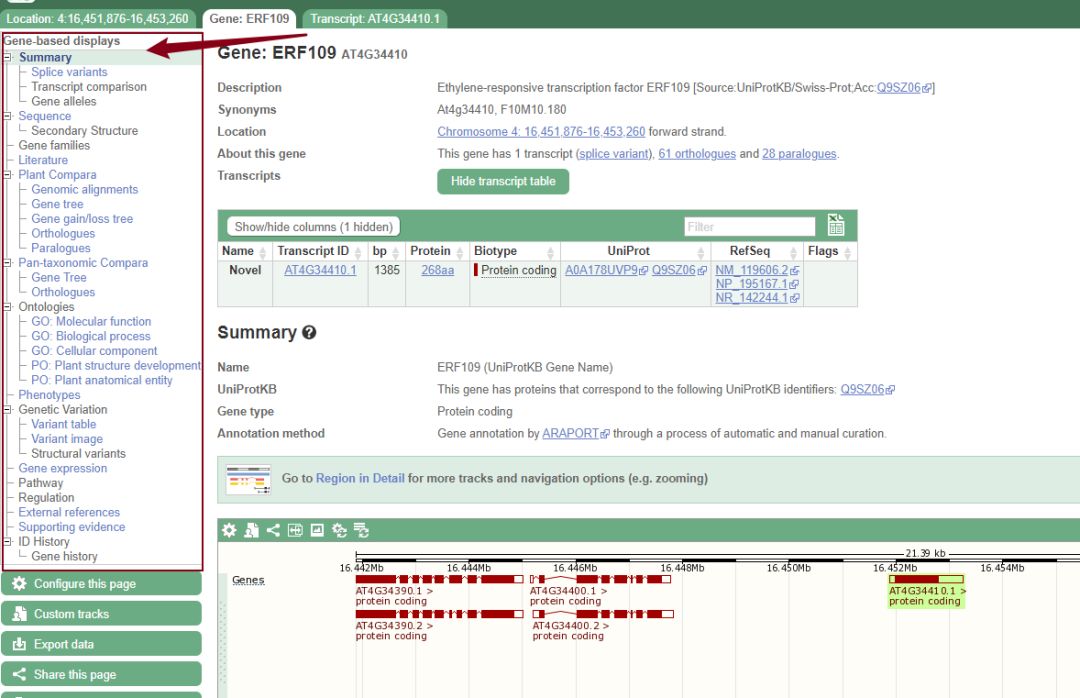
查看单基因信息
这里以最基础的单基因信息查询作为示例。Ensembl 的 RUSTful API 支持使用各种语言实现,既可以在 Unix 操作系统中使用 curl 和 wget 命令,也可以使用 python java perl 和 R 语言来操作。
如果要是用 wget 来查询一个基因的话,查询规则示例是http://rest.ensembl.org/lookup/id/AT4G34410?expand=1'。其实这就是一个简单的我们都能理解的「网址」,其中 id 后面是我们要查询的基因id,问号后面可以添加任意支持的参数。另外,还需要 header 信息'Content-type:application/json' 来指定获取的资源类型。
运行命令如下:
wget -q --header='Content-type:application/json' \
'http://rest.ensembl.org/lookup/id/AT4G34410?expand=1' -O -
得到的内容会是一行 json 内容,这个信息大家看到都是崩溃的我就不直接放上来了。我们可以使用一些命令和操作稍微进行美化。
wget -q --header='Content-type:application/json' \
'http://rest.ensembl.org/lookup/id/AT4G34410?expand=1' -O - | jq '.' -
通过 jq 这个命令,可以让输出变成标准的 json 格式。输出截图如下:
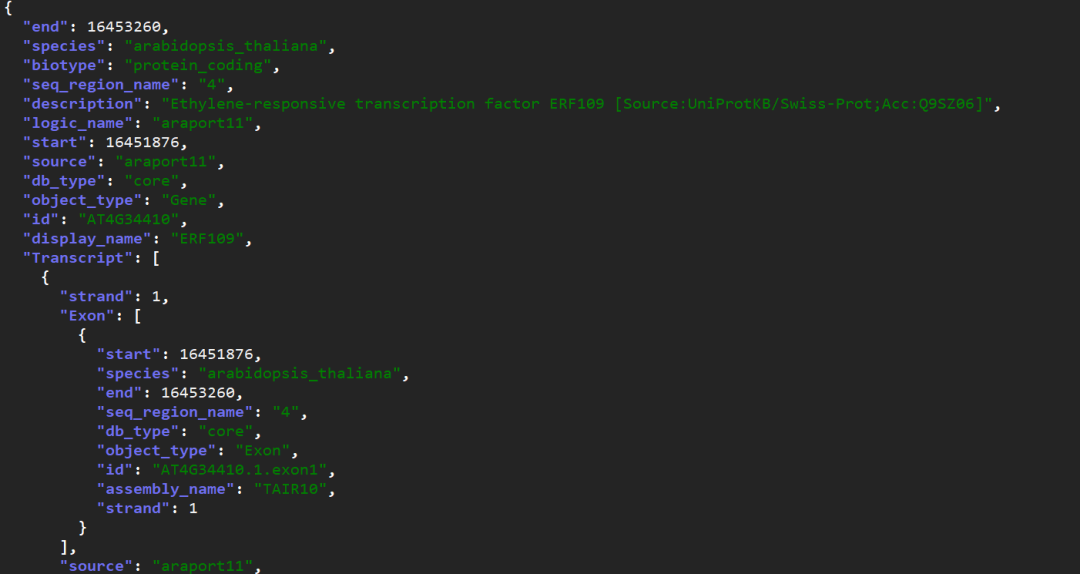
为了更方面的处理 json 内容和进行后一步的分析,我们可以移步到 R 中,看看如何使用。
要在 R 使用 RUSTful API 并进行后续的 json 文件处理,首先需要加载两个包,httr 用来调取 GET 和 POST 等命令,jsonlite 用来处理 json 格式的文件。
httr 会把 GET 的结果保存为一个 response 类型的对象,其中包括了 url,状态码以及header等各种各样的信息,jsonlite 可以帮助我们根据需求提取 json 里的内容并输出为 list 对象。
简单的运行命令如下:
# 加载 R 包
library(httr)
library(jsonlite)
# 指定 server
server <- "http://rest.ensembl.org"
# 指定查询内容,为了方便展示这里 expand=0
ext <- "/lookup/id/ENSG00000157764?expand=0"
# 使用 httr 包的 GET 进行查询
r <- GET(paste(server, ext, sep = ""), content_type("application/json"))
# 这里的 r 是一个response类型的对象。
# 将http错误转换为R错误方便debug
stop_for_status(r)
到这里其实查询的步骤已经完成,对象 r 的结构如下:

接下来就是首先把这个对象中的内容转换为json然后在转换为table即可,命令非常简单。
list <- fromJSON(toJSON(content(r)))
tb <- do.call(rbind,list)
得到的table内容如下
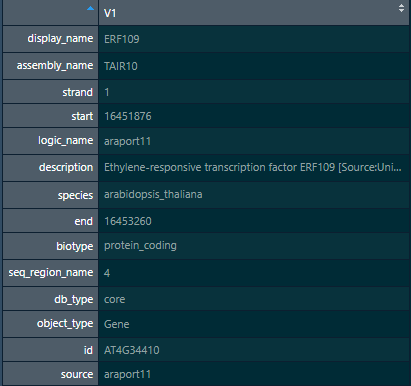
如果需要稍微优雅一些,可以改写为一个函数,如果需要一次查找多个基因,可以使用 POST 方法。
你可能会好奇使用 API 的优势在哪里。
如果只是查找一个基因,API 的优势并不明显,如果只是偶尔查找几个基因,API 的优势也不明显。
那怎么使用就有优势了呢,类比于「印象笔记」以及和它相关的使用了「印象笔记 API」的第三方应用,不知道会不会给你一些启发。
话不能说的太透,点到为止,更多的应用场景我们以后有机会再聊。