大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
哺乳动物的早期胚胎发育会经历重要的表观遗传重编程过程,这一过程需要重置从亲本基因组继承的表观遗传信息,以启动胚胎基因表达程序,而全基因组去甲基化对表观遗传重编程至关重要。哺乳动物基因组在CpG位点上有较高水平的甲基化,主要以5-甲基胞嘧啶(5mC)的形式存在。早期胚胎发育的一个显著特征是DNA甲基化广泛丢失,尤其在第一次细胞分裂期间。这一过程在哺乳动物中保守,但在去甲基化动态和程度上存在差异。全基因组去甲基化过程对早期胚胎的发育能力至关重要,具有高甲基化基因组的小鼠胚胎表现出异常。哺乳动物中,与全基因组DNA甲基化减少相关的生物学过程相对异常。因此早期胚胎具有特异性调控机制,但这些机制目前尚部分未知。DNA甲基转移酶1(DNMT1)在这个阶段主要被排除在细胞核外,但其核内活性是否受其他机制调节尚不明确。
近日,中国科学院生物物理研究所朱冰和张珠强团队共同研究发现,母源蛋白Pramel15通过泛素-蛋白酶体通路系统靶向DNA甲基转移酶1(DNMT1)进行降解,调节其在早期胚胎细胞核中的DNA甲基化水平,影响DNA去甲基化过程。这一发现首次揭示早期胚胎细胞核中存在着依赖于蛋白酶体的DNMT1调控机制,帮助受精卵(合子)DNA去甲基化。相关研究成果以“Pramel15 facilitates zygotic nuclear DNMT1 degradation and DNA demethylation“为题发表在Nature子刊《Nature Communications》。

标题:Pramel15 facilitates zygotic nuclear DNMT1 degradation and DNA demethylation(Pramel15促进受精卵细胞核的DNMT1降解和DNA去甲基化)
发表期刊:Nature Communications
影响因子:IF 14.7/1区
技术平台:单细胞/微量WGBS、RNA-seq等(易基因金牌技术)
本研究首先假设早期胚胎中存在调控整体 DNA 去甲基化过程的未知调节机制,并通过寻找 DNA 甲基化的新调节因子来研究这一假设,其表达对早期胚胎具有特异性。为此,研究首先鉴定出一个母源蛋白 Pramel15,Pramel15通过Cullin-RING E3连接酶靶向DNMT1进行降解,可调节胚胎核内 DNMT1 的蛋白水平。而Pramel15 缺失会提高合子原核中的DNMT1水平,损害合子DNA去甲基化,并导致小鼠早期胚胎在全基因组中表现出 DNA 甲基化水平的随机增加。本研究揭示Pramel15可以在受精卵DNA复制过程中调控细胞核中的DNMT1水平,从而确保早期胚胎中有效的DNA甲基化重编程。这些发现将有助于进一步理解调控哺乳动物 DNA 甲基化重编程的复杂机制。
研究方法
- 基因表达分析:通过转录组和翻译组分析,研究Pramel15在早期胚胎发育中的表达模式。
- 蛋白互作分析:利用免疫共沉淀(Co-IP)和质谱分析,确定Pramel15与DNMT1互作。
- 细胞和分子生物学技术:包括CRISPR/Cas9基因编辑、免疫荧光、西方印迹(Western blot)等,用于研究Pramel15和DNMT1的功能。
- 表观遗传学分析:利用单细胞全基因组亚硫酸盐测序(scWGBS)技术分析DNA甲基化水平。
- 遗传学模型:生成Pramel15基因敲除小鼠模型,研究其在胚胎发育中的作用。
结果图形
(1)筛选鉴定促进DNA去甲基化因子Pramel15,Pramel15过表达导致整体DNA去甲基化
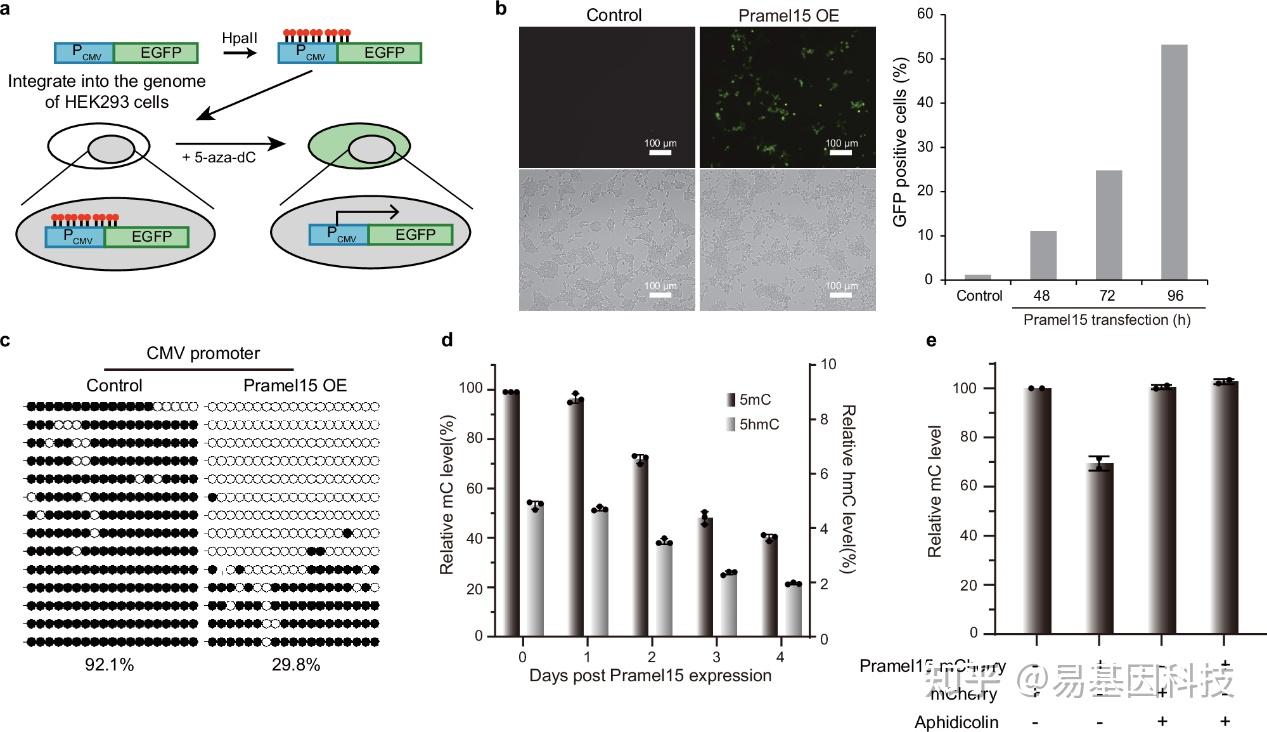
图1:Pramel15过表达导致整体DNA被动去甲基化。
a. B2-17细胞系(对DNA去甲基化敏感)方案。CMV启动子驱动的GFP片段通过HapII体外甲基化,然后整合到HEK293细胞中。选择低GFP背景和对5-氮杂-2'-脱氧胞苷高敏感的克隆作为B2-17。
b. 左侧荧光图像显示B2-17转染Pramel15表达质粒后GFP激活的细胞。右侧条形图显示使用流式细胞排序(FACS)的B2-17转染Pramel15表达质粒后GFP阳性细胞比例。进行三次生物学重复。
c. BS-seq显示B2-17细胞转染Pramel15表达质粒后CMV启动子的DNA甲基化状态。96小时后提取基因组DNA,然后进行CMV启动子的亚硫酸盐测序。
d. 条形图显示B2-17基因组中5-甲基胞嘧啶(mC)和5-羟甲基胞嘧啶(hmC)的相对水平。细胞在Pramel15表达后的1-4天收获,提取基因组DNA进行核苷质量谱分析。参照第0天细胞的5mC水平进行归一化。
e. B2-17基因组DNA在转染表达Pramel15-mCherry或mCherry的质粒后提取进行核苷质量谱分析。
(2)Pramel15与DNMT1 RFTS结构域互作并调节DNMT1蛋白稳定性
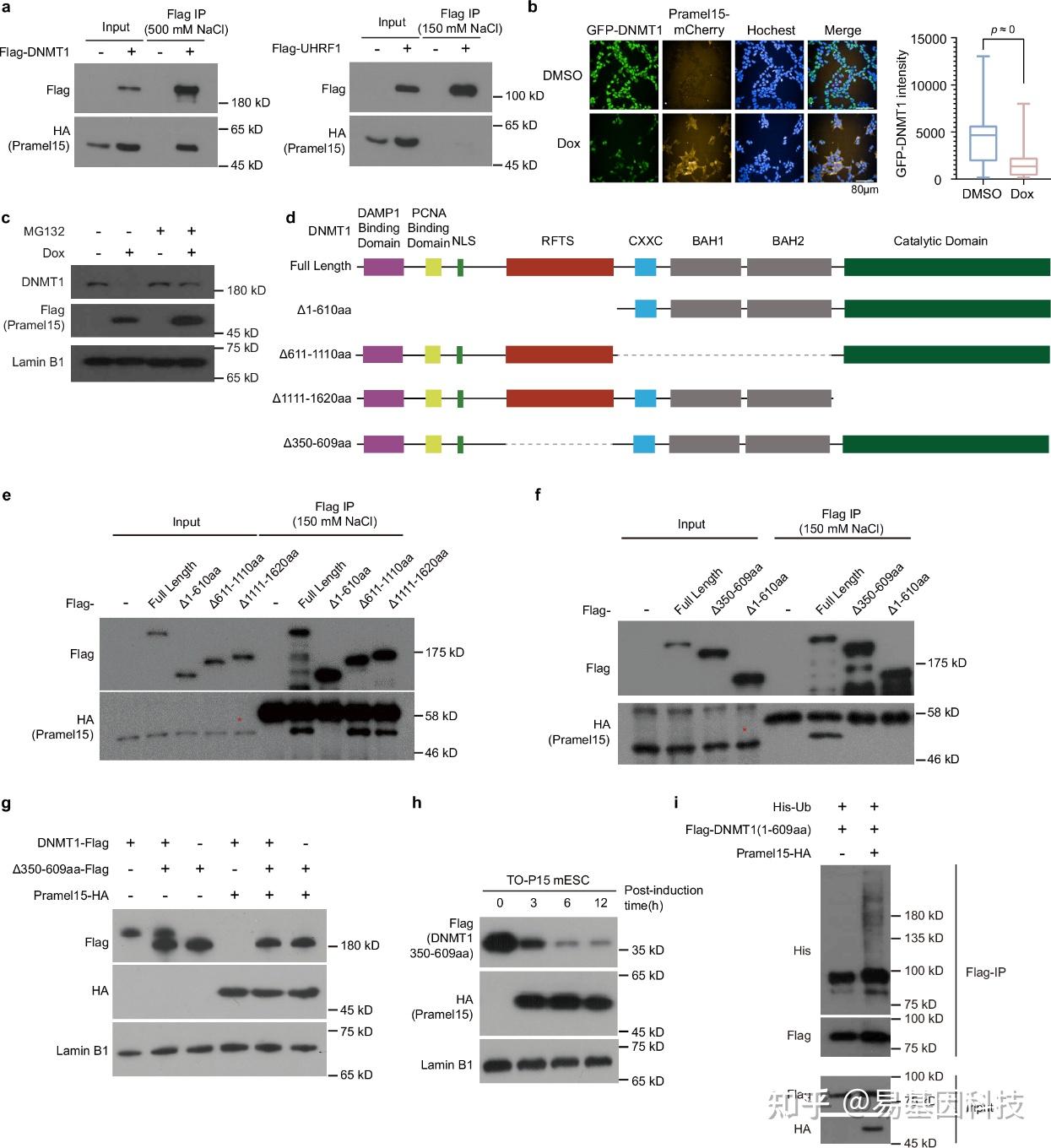
图2:Pramel15与DNMT1互作,并通过泛素-蛋白酶体通路介导DNMT1降解。
a. 293FT细胞转染Pramel15-HA和DNMT1-Flag(上)或UHRF1-Flag(下)表达质粒后的Flag免疫沉淀。
b. 高含量图像显示有无Pramel15-mCherry诱导的DNMT1-GFP水平,通过多西环素(Dox)处理48小时(左),箱线图显示DMSO组(n=3973)和Dox组(n=3109)的统计结果(右)。
c. 在表达Dox诱导的Pramel15-Flag的T-REx293细胞中,DNMT1蛋白水平的Western Blot分析,
d. DNMT1及其截短体的示意图。
e-f. 293FT细胞转染DNMT1-Flag或其截短体和Pramel15-HA表达质粒后的Flag免疫沉淀。
g. 293FT细胞中有无Pramel15-HA的Flag-DNMT1和Flag-DNMT1(Δ350-609aa)蛋白水平Western Blot分析。
h. 在表达Flag-DNMT1(350-609aa)和Dox诱导的Pramel15-HA的mES细胞中,Flag-DNMT1(350-609aa)蛋白水平的Western Blot分析。
i. 293FT细胞转染表达His-ubiquitin、Flag-DNMT1(1-609aa)和Pramel15-HA或空质粒,并用20μM MG132处理后的Flag免疫沉淀。所有通过Western Blot分析的免疫沉淀和定量实验至少重复三次。
(3)Cullin-RING连接酶参与Pramel15介导的DNMT1降解
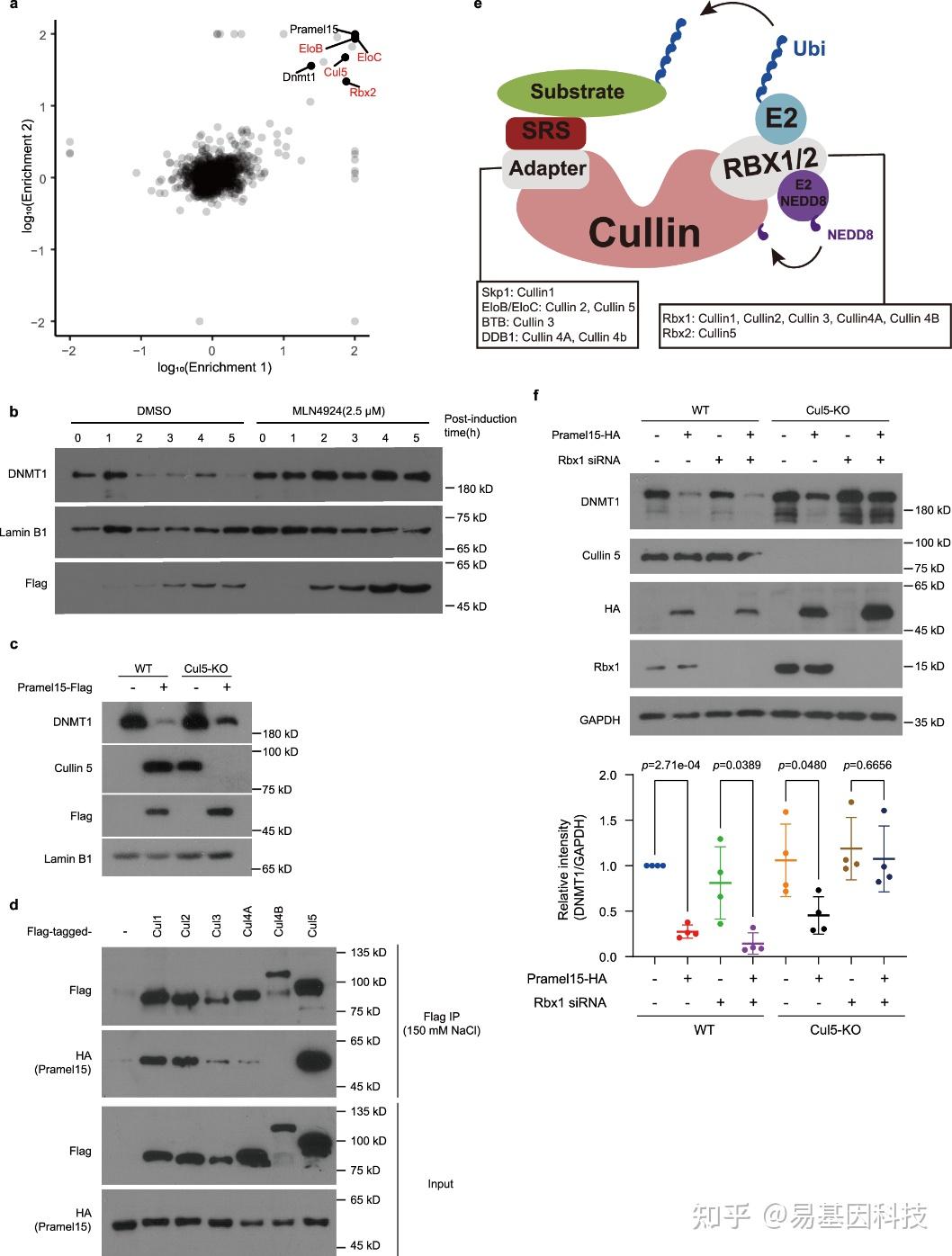
图3:Cullin-RING E3泛素连接酶参与Pramel15介导的DNMT1降解。
a. 在两个独立实验中, mES细胞中Pramel15-Flag共免疫沉淀的蛋白质无标签质谱分析。Pramel15、DNMT1和CRL5复合体的亚基(红色高亮)是主要的富集标靶。
b. 表达Dox诱导的Pramel15-Flag的mES细胞中,有或没有 2.5 μM MLN4924处理的DNMT1蛋白水平的Western Blot分析。
c. 表达Dox诱导的Pramel15-Flag的mES细胞和Cul5-KO mES细胞中,DNMT1蛋白水平的Western Blot分析。
d. 293FT细胞转染表达Pramel15-HA和不同Flag标记的Cullin蛋白质粒后的Flag免疫沉淀。
e. Cullin-RING E3泛素连接酶复合体的模型。不同的Cullin复合体包含不同接头,Cul5与Rbx2结合,而其他Cullin蛋白与Rbx1结合。
f. 表达Dox诱导的Pramel15-Flag的mES细胞和Cul5-KO mES细胞中,有或没有Rbx1 siRNA转染的DNMT1蛋白水平的Western Blot分析(左)。四个生物学重复的统计数据(右)。
(4)Pramel15介导DNMT1降解的主要motifs
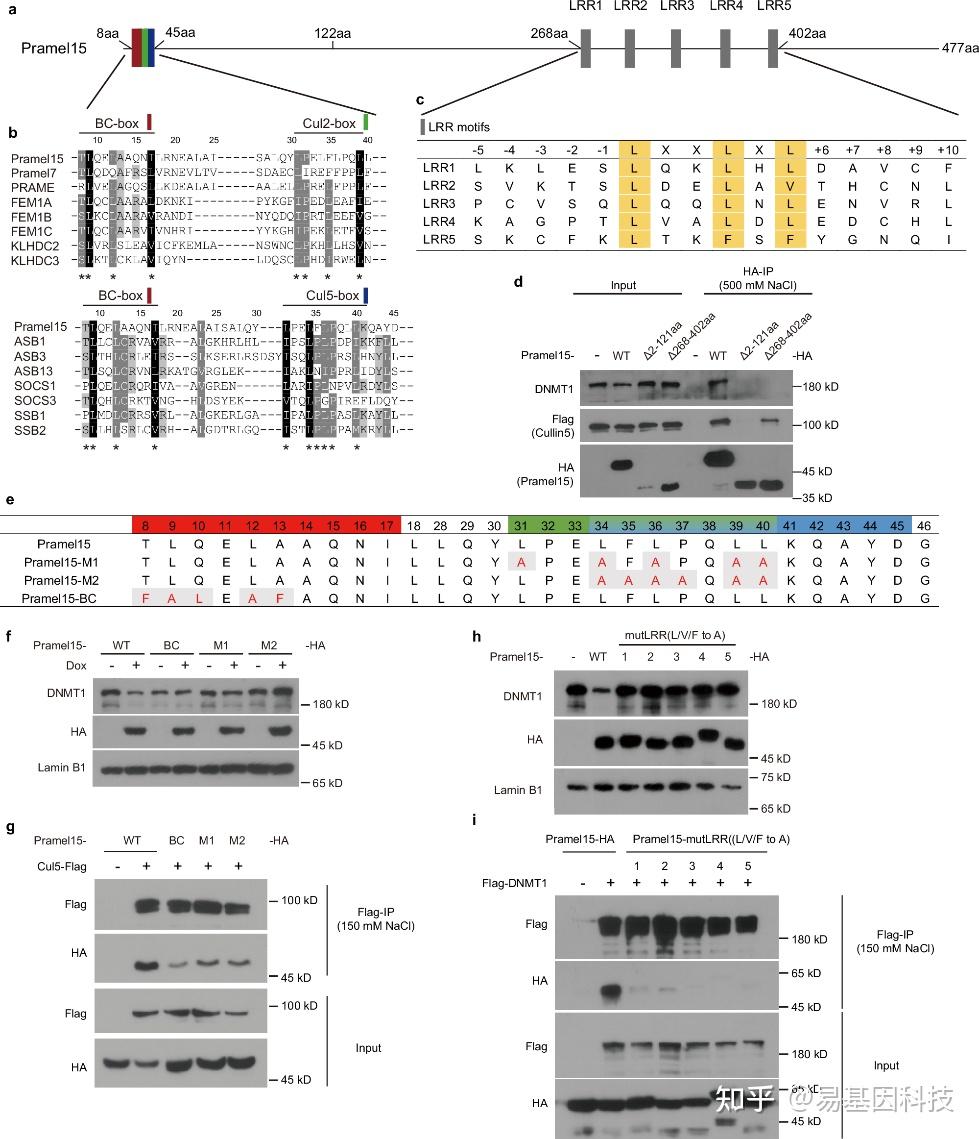
图4:Pramel15的域结构及其与Cul5和DNMT1的互作。
a. 示意图展示Pramel15的域结构,包含预测的BC-box、Cul2-box、Cul5-box和LRR(亮氨酸富集重复序列)motif(基序)。
b. Pramel15与含有BC和Cul2 box或BC和Cul5 box motif的蛋白质的多序列比对,突出显示保守的残基。黑色表示相同的氨基酸,非常相似-深灰色,相似-浅灰色。星号标记保守的Pramel15残基。
c. 预测的Pramel15 LRR motif序列,LRR序列模式(LXXLXL)中保守的亮氨酸用黄色突出显示。
d. 293FT细胞转染表达Pramel15-HA和Flag标记的Cul5的质粒后的HA免疫沉淀。Western blot显示DNMT1和Cul5与Pramel15、Pramel15(Δ2-121aa)和Pramel15(Δ268-402aa)的关联。
e. Pramel15在BC-box、Cul2-box和Cul5-box的点突变体表格。
f. Western blot显示在293FT细胞中表达Dox诱导HA标记的Pramel15突变体(如e所示)的DNMT1蛋白水平。
g. 293FT细胞转染表达HA标记的Pramel15或N端突变体和Flag标记的Cul5的质粒后的Flag免疫沉淀。Western blot显示Cul5与Pramel15及其突变体的关联。
h. 在293FT细胞中表达Dox诱导的HA标记的LRR motif突变体(根据图4c,L/V/F替换为A)的Pramel15的DNMT1蛋白水平的Western blot。
i. 293FT细胞转染表达Flag标记的DNMT1和HA标记的Pramel15或LRRs突变体(根据c,L/V/F替换为A)的质粒,并用2.5μM MLN4924处理以防止DNMT1降解后的Flag免疫沉淀。Western blotting显示DNMT1与Pramel15及其LRRs突变体的关联。
(5)Pramel15基因敲除小鼠没有明显的发育和生育缺陷,Pramel15调节早期胚胎细胞核中DNMT1蛋白水平
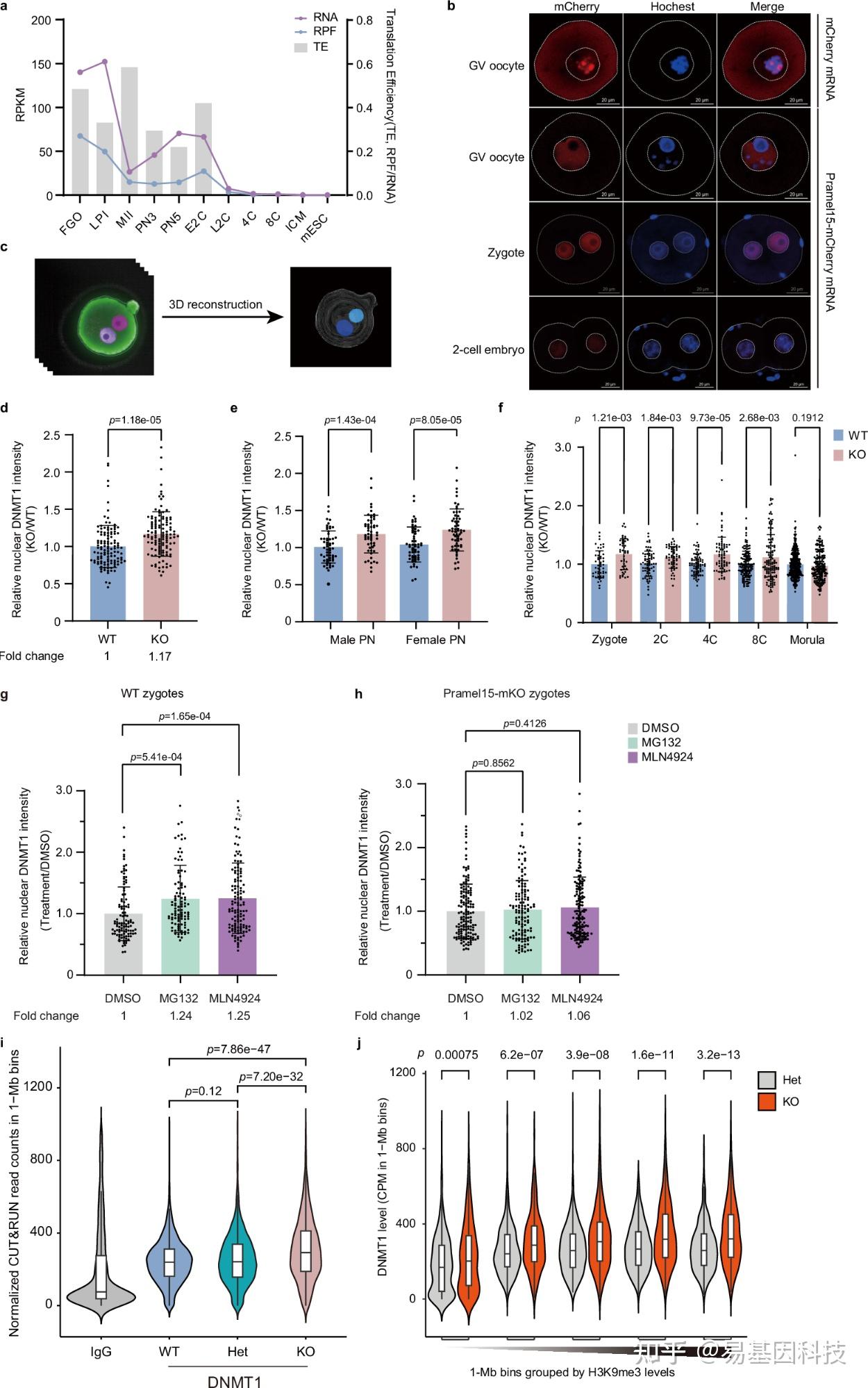
图5:母源性Pramel15缺失增加了胚胎核中的DNMT1水平。
a. Pramel15 mRNA和核糖体保护片段(RPF)在早期发育过程中的动态变化线图,以及显示翻译效率(RPF/mRNA)的条形图。源数据:GSE165782。
b. 通过显微注射Pramel15-mCherry mRNA在GV期卵母细胞、合子(受精卵)和2C胚胎中Pramel15的定位代表性荧光图像。三次重复。
c. 高含量成像分析系统获取的图像的3D重建示意图。
d. 野生型(WT,n=118)和Pramel15基因敲除(KO,n=118)卵母细胞的受精卵核中相对DNMT1强度条形图。
e. 雄性(WT,n=58;KO,n=56)和雌性(WT,n=57;KO,n=60)原核中相对DNMT1强度条形图,原核根据核大小来定义。
f. 受精卵(合子)(WT,n=49;KO,n=45)、2细胞胚胎(2C)(WT,n=67;KO,n=57)、4细胞胚胎(4C)(WT,n=73;KO,n=71)、8细胞胚胎(8C)(WT,n=139;KO,n=124)和囊胚(WT,n=337;KO,n=185)核中相对DNMT1强度条形图。DNMT1强度在每个阶段都参照WT组进行归一化。
g-h. 用DMSO(WT,n=107;KO,n=145)、MG132(WT,n=100;KO,n=127)和MLN4924(WT,n=126;KO,n=143)处理的受精卵(合子)核中相对DNMT1强度条形图。合子在受精后2小时转移到含有DMSO、10μM MG132和5μM MLN4924的HTF中,进一步培养额外的9小时。
i. 基因组被划分为1Mb bins。归一化DNMT1 CUT&RUN计数代表DNMT1水平。
j. 每个相应基因组bins中DNMT1水平由归一化计数表示。基因组 bins根据MII期卵母细胞中H3K9me3 ChIP-seq reads排序。H3K9me3 ChIP-seq数据来源:GSM2588560。
(6)Pramel15 缺失胚胎中 DNA 甲基化水平增加
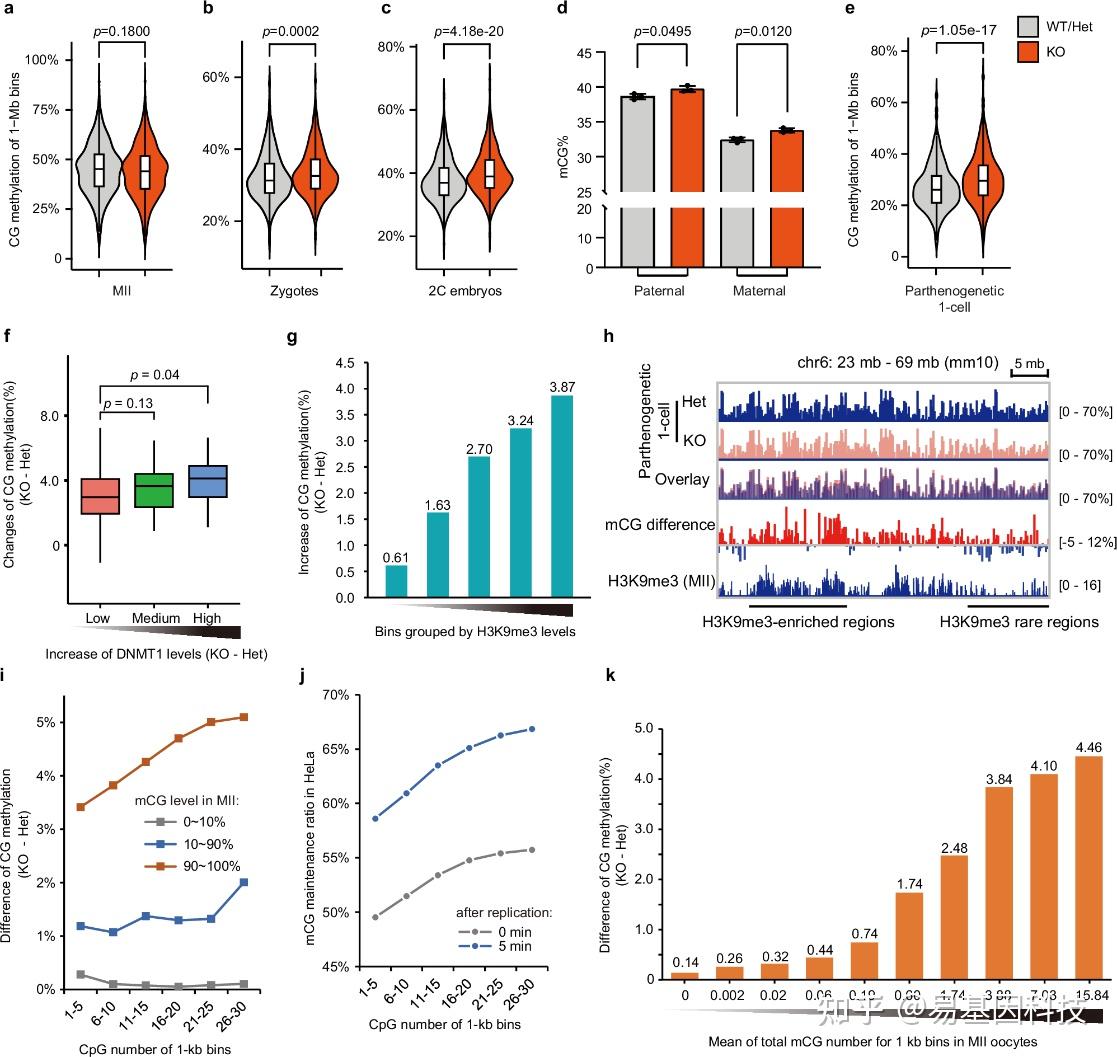
图6:母源性Pramel15缺失导致DNA甲基化水平增加。
a-c. WGBS揭示野生型(WT)或Pramel15杂合(Het)和Pramel15基因敲除(KO)卵母细胞的MII期卵母细胞、受精卵(合子)和2细胞胚胎的全基因组DNA甲基化水平。
d.受精卵中父本和母本基因组的全基因组DNA甲基化水平,分别通过DBA/2J和C57BL/J的单核苷酸多态性(SNPs)进行区分。
e. Pramel15 Het和KO卵母细胞孤雌生殖(parthenogenesis)1细胞胚胎的全基因组DNA甲基化水平。
f. Pramel15 Het和KO卵母细胞孤雌生殖1细胞胚胎之间1Mb bins的平均CpG甲基化差异箱线图。基因组bins根据DNMT1 CUT&RUN信号增加排序。
g. Pramel15 Het和KO卵母细胞孤雌生殖1细胞胚胎之间1Mb bins的平均CpG甲基化差异条形图。基因组bin根据MII期卵母细胞中H3K9me3 ChIP-seq reads排序。
h. Pramel15 Het和KO卵母细胞孤雌生殖1细胞胚胎中CpG甲基化和H3K9me3密度差异基因组浏览器视图。H3K9me3 ChIP-seq数据来源:GSM2588560。
i. MII期卵母细胞中1kb bins的CpG甲基化变化和CpG数量相关性分析。
j. DNA复制过程中1kb bins的DNA甲基化维持效率,展示了复制耦合(0分钟)和复制后5分钟的情况。数据来源:GSE131098。
k. Pramel15 Het和KO卵母细胞孤雌生殖1细胞胚胎中1kb bins的平均CpG甲基化差异(mCG%KO− mCG%het),按MII期卵母细胞中1kb bins总甲基化CpG数量平均值排序。
易小结
本研究首先揭示了Pramel15通过Cullin-RING E3泛素连接酶复合体促进DNMT1降解。同时Pramel1的缺失导致受精卵细胞核中DNMT1水平升高,影响受精卵DNA去甲基化,并在早期胚胎中引起DNA甲基化水平的随机增加。此外,Pramel15基因敲除小鼠表现显著的DNA甲基化增加,但并未观察到明显的发育或生育缺陷。
研究结果表明Pramel15通过调节DNMT1在核内的丰度,精细调控整体去甲基化过程,这对于早期胚胎的表观遗传重编程至关重要。
研究亮点
- 新的表观遗传调控机制:揭示了Pramel15在DNA去甲基化中的新作用,为理解哺乳动物DNA甲基化重编程提供了新的视角。
- 泛素-蛋白酶体系统新功能:发现Pramel15通过泛素-蛋白酶体系统调节DNMT1的稳定性,为DNA甲基化调控提示新机制。
- 胚胎发育新认识:研究结果为理解早期胚胎发育中的DNA甲基化动态变化提供了重要信息,可能对生殖生物学和再生医学有重要意义。
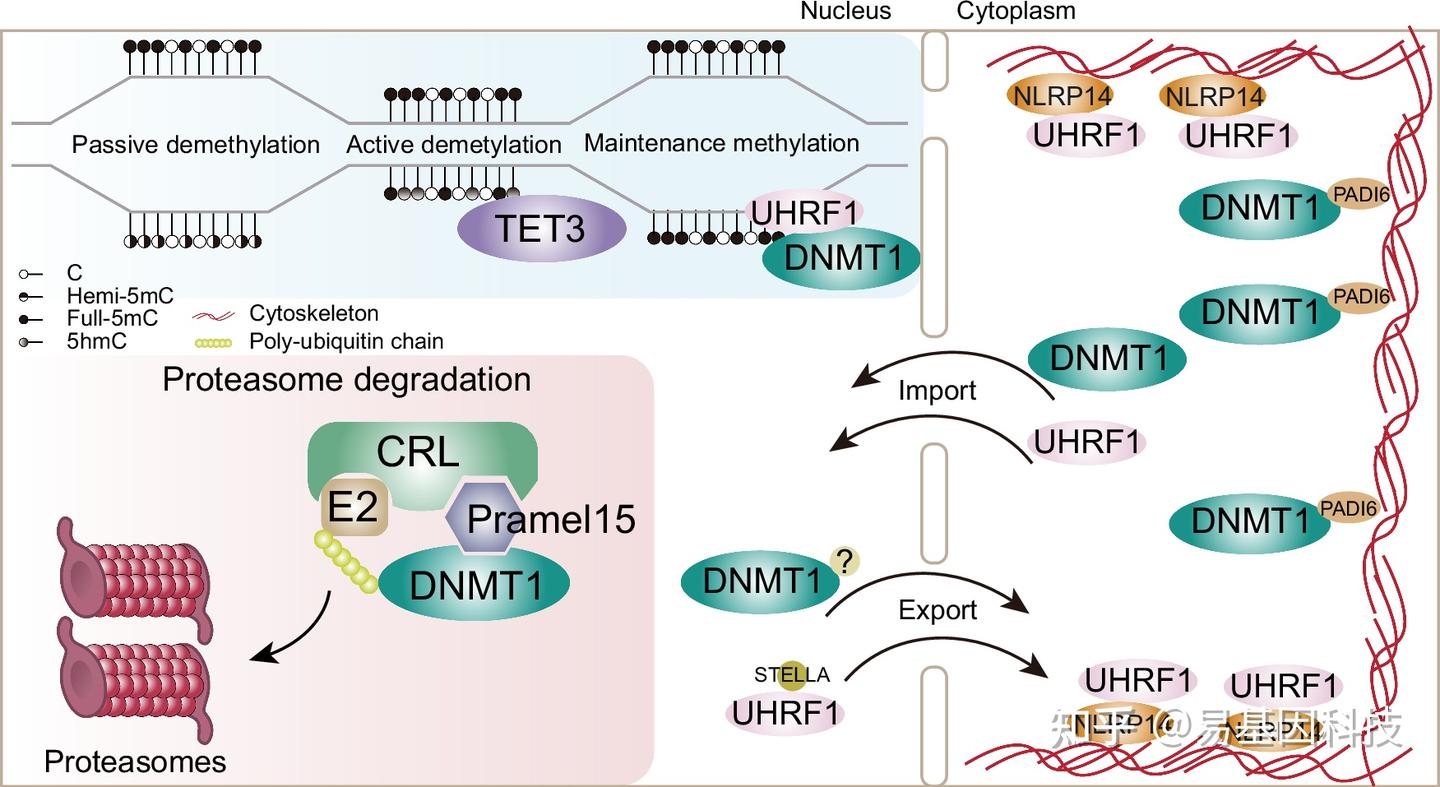
图7:受精卵DNA甲基化重编程参与机制模型。
为保护受精卵中DNA甲基化重编程,DNMT1和UHRF1受多层调控。NLRP14最近被鉴定为调控UHRF1细胞质滞留因子,而DNMT1通过PADI6在细胞质中受限。同时,DNMT1从细胞核中积极出核。但涉及DNMT1细胞核出核和细胞质滞留因子仍然未知。在细胞核内,TET3通过主动去甲基化某些区域参与DNA重编程。另一方面,在重编程过程中,某些区域的DNA甲基化得以保留,例如印记区域,这依赖于细胞核中的DNMT1/UHRF1。为了防止过量的DNMT1损害DNA去甲基化,Pramel15通过招募Cullin-RING E3泛素连接酶,靶向DNMT1通过泛素-蛋白酶体系统(UPS)进行降解。
目前,易基因科技有限公司在DNA甲基化修饰等表观遗传检测领域,积累了一系列国际先进技术,可对高通量单细胞甲基化组学研究,微量DNA甲基化测序,ctDNA、FFPE等严重降解痕量DNA甲基化检测等,提供多种有效解决方案。低成本、高通量的单细胞甲基化组学测序技术建立,将为研究者对不同时间和空间的单细胞表观修饰研究奠定基础。
1)微量细胞或单细胞全基因组甲基化测序(Micro DNA-WGBS)DNA起始量:
单细胞/100-1000个细胞
1ng基因组DNA
90%以上基因组CG覆盖
2)微量细胞或DNA简化基因组甲基化测序(Micro DNA-RRBS)DNA起始量:
1ng基因组DNA;
10-20M有效CG位点覆盖;
10-20G测序数据量;
3)微量cfDNA简化基因组甲基化测序(cfDNA-RBS)
100ul血浆或1ng cfDNA
10M有效CG位点覆盖
15-20G测序数据量

易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G、ac4C)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
参考文献:
Tan J, Li Y, Li X, Zhu X, Liu L, Huang H, Wei J, Wang H, Tian Y, Wang Z, Zhang Z, Zhu B. Pramel15 facilitates zygotic nuclear DNMT1 degradation and DNA demethylation. Nat Commun. 2024 Aug 25;15(1):7310. pii: 10.1038/s41467-024-51614-0. doi: 10.1038/s41467-024-51614-0. PubMed PMID: 39181896.
相关阅读:
1. Nature | 易基因DNA甲基化测序助力人多能干细胞向胚胎全能8细胞的人工诱导
2. Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
3. 项目文章 | WGBS+RNA-seq揭示松材线虫JIII阶段形成过程中的DNA甲基化差异
4. 项目文章|WGBS揭示Vpr蛋白在HIV-1感染中对CD4+ T细胞DNA甲基化变化的作用
5.项目文章 | WGBS+RNA-seq揭示黄瓜作物的“源-库”关系受DNA甲基化调控
6. 项目文章 | WGBS等揭示SOX30甲基化在非梗阻性无精症中的表观遗传调控机制
7. 项目文章|WGBS+RNA-seq揭示PM2.5引起男性生殖障碍的DNA甲基化调控机制
8.项目文章:oxBS揭示复发性膀胱癌的DNA甲基化和羟甲基化变化并鉴定预测PD-L1表达标记物
9.项目文章|PNAS:中国农大田见晖教授团队揭示DNA甲基化保护早期胚胎线粒体基因组稳定性
10.年终盘点 | 易基因2023年度DNA甲基化研究项目文章精选
11. DNA甲基化修饰整体研究方案
12. DNA与蛋白质互作及染色质开放性研究方案
13. RNA甲基化修饰整体研究方案
14. 游离细胞DNA(cfDNA)检测整体研究方案
15. 技术推介 | 全基因组重亚硫酸盐甲基化测序(WGBS)