这篇论文介绍了一种名为PointSite的创新方法,用于准确识别蛋白质结构中的配体结合位点(LBS),相较于先前的技术具有更高的准确性。PointSite采用了蛋白质为中心的方法,而不是仅仅依赖于蛋白质结构外的伪表面点,它利用了蛋白质原子的局部连接性和全局三维几何信息
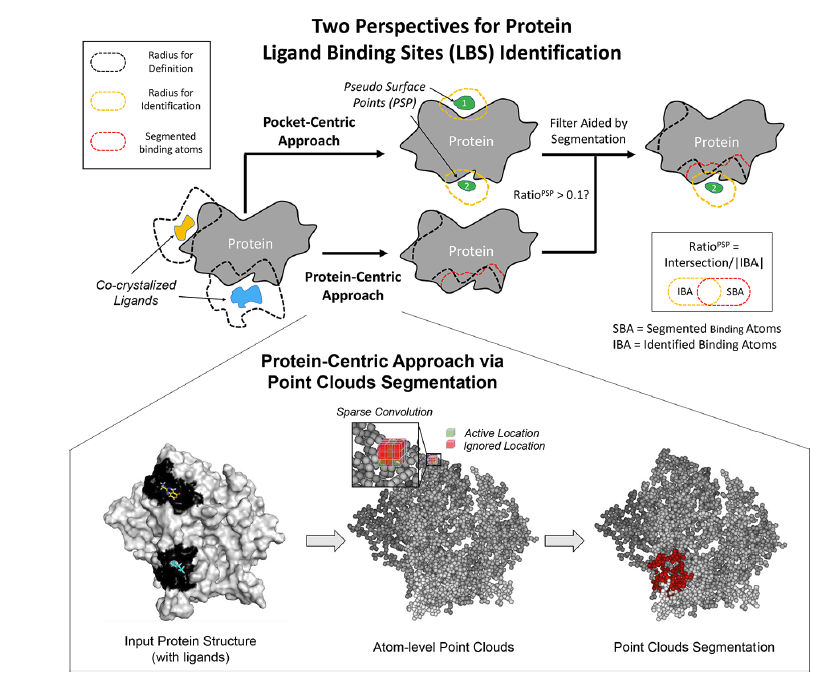
- 将原始的3D蛋白质结构转换为点云。这种转换可能有助于简化蛋白质结构的表示,同时保留了关键的空间信息。利用机器学习架构,特别是基于次流形稀疏卷积的U-Net,以原子级别进行分割。这种方法允许对蛋白质结构中的配体结合原子进行细粒度识别。
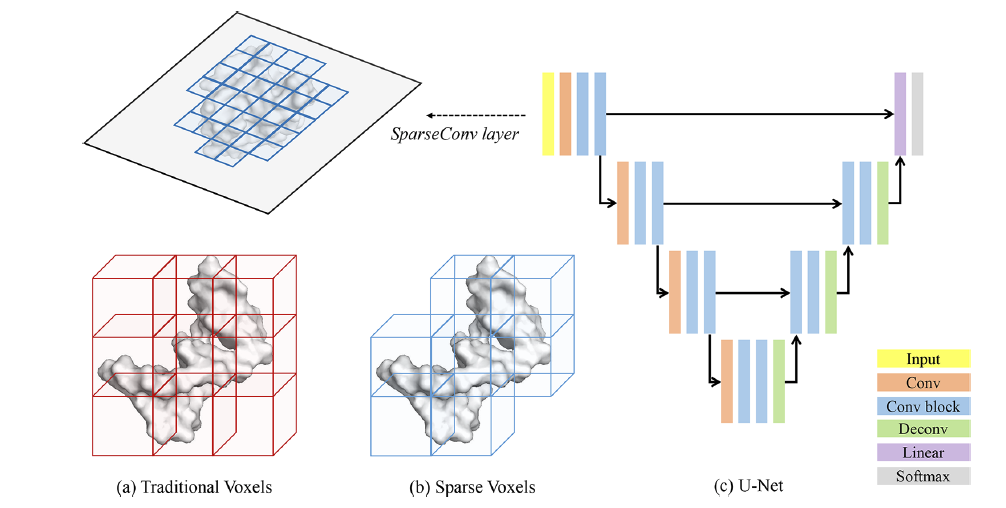
- PointSite通过考虑详细的原子级表示来增强特征学习。这种增强有助于在准确识别配体结合原子方面表现出更高的性能。PointSite在原子交集联合(atom-IoU)方面显著优于先前的方法,表明其在识别配体结合位点方面具有更高的准确性。通过使用预测概率较高的分段配体结合原子,PointSite作为先前以口袋为中心的方法预测的过滤器。这种过滤机制减少了识别LBS候选物中的假阳性。
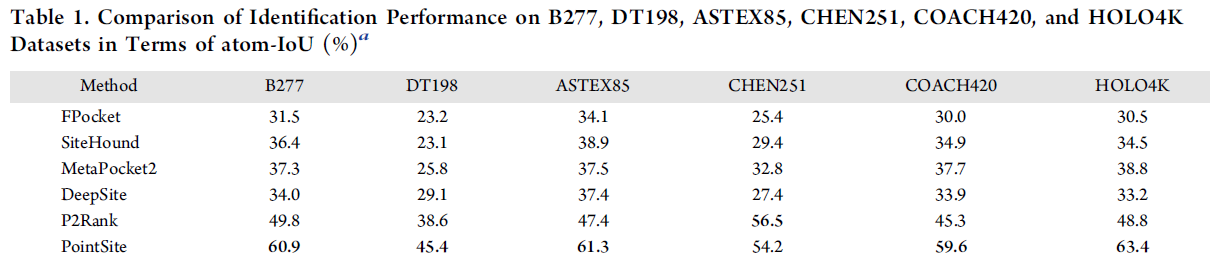
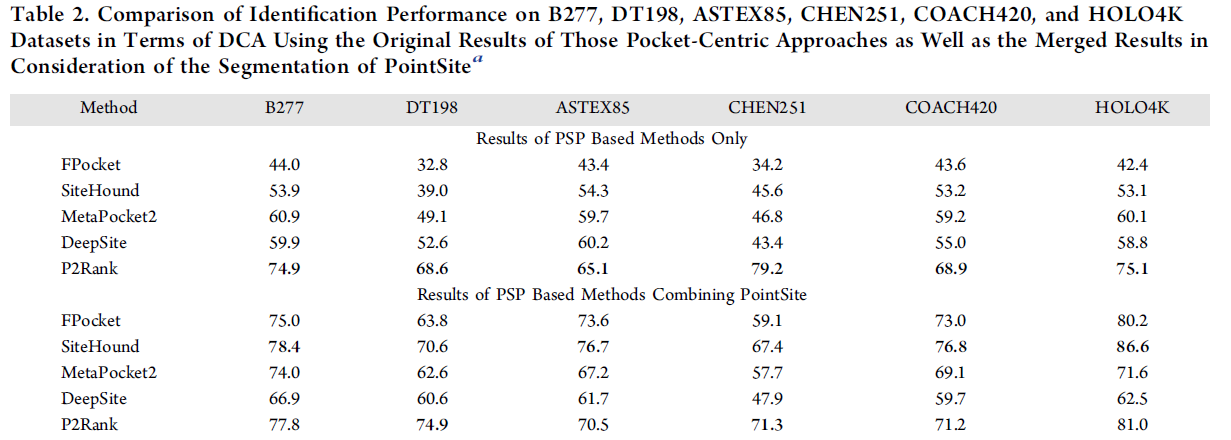
- PointSite在各种基准测试中实现了最先进的性能
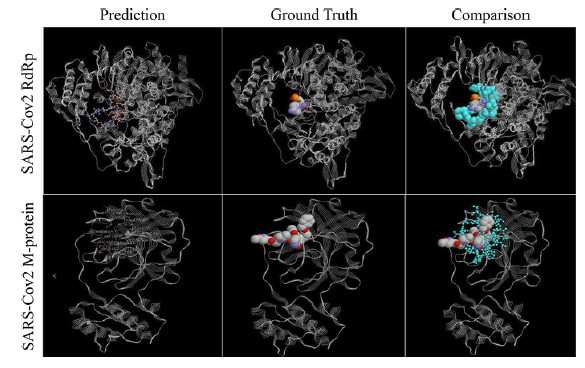
总的来说,PointSite通过采用蛋白质为中心的方法,利用先进的机器学习技术进行分割,并在准确识别配体结合原子方面表现出优越性能,解决了先前以口袋为中心的方法的局限性。其泛化到未结合蛋白质的能力以及减少假阳性的特点使其成为了解蛋白质功能并辅助基于结构的药物设计的有前景工具。