健康饮食指数 (HEI) 是评估一组食物是否符合美国人膳食指南 (DGA) 的指标。Dietindex包提供用户友好的简化方法,将饮食摄入数据标准化为基于指数的饮食模式,从而能够评估流行病学和临床研究中对这些模式的遵守情况,从而促进精准营养。
该软件包可以计算以下饮食模式指数:
• 2020 年健康饮食指数(HEI2020 和 HEI-Toddlers-2020)
• 2015 年健康饮食指数 (HEI2015)
• 另类健康饮食指数(AHEI)
• 控制高血压指数 (DASH) 的饮食方法
• DASH 试验中的 DASH 份量指数 (DASHI)
• 替代地中海饮食评分 (aMED)
• PREDIMED 试验中的 MED 份量指数 (MEDI)
• 膳食炎症指数 (DII)
• 美国癌症协会 2020 年饮食评分(ACS2020_V1 和 ACS2020_V2)
• EAT-Lancet 委员会 (PHDI) 的行星健康饮食指数
上一期咱们咱们初步介绍了dietaryindex包计算膳食指数,这期咱们继续介绍。
先导入需要的R包
library(ggplot2)
library(dplyr)
library(tidyr)
library(dietaryindex)
library(survey)
options(survey.lonely.psu = "adjust") ##解决了将调查数据分组到小组的孤独psu问题
导入R包自带的数据,其中DASH_trial和PREDIMED_trial是临床试验的数据,NHANES_20172018数据属于临床流行病学的研究。
假设咱们想研究临床试验数据的膳食指数和流行病学中的膳食指数由什么不同?
data("DASH_trial")
data("PREDIMED_trial")
data("NHANES_20172018")
先设置一下咱们的目录
setwd(“E:/公众号文章2024年/dietaryindex包计算营养指数”)
导入R包关于设置好的营养指数的数据,这个数据需要到作者的主页空间下载,如果需要我下载好的,公众号回复:代码
# Load the NHANES data from 2005 to 2018
## NHANES 2005-2006
load("NHANES_20052006.rda")## NHANES 2007-2008
load("NHANES_20072008.rda")## NHANES 2009-2010
load("NHANES_20092010.rda")## NHANES 2011-2012
load("NHANES_20112012.rda")## NHANES 2013-2014
load("NHANES_20132014.rda")## NHANES 2015-2016
load("NHANES_20152016.rda")
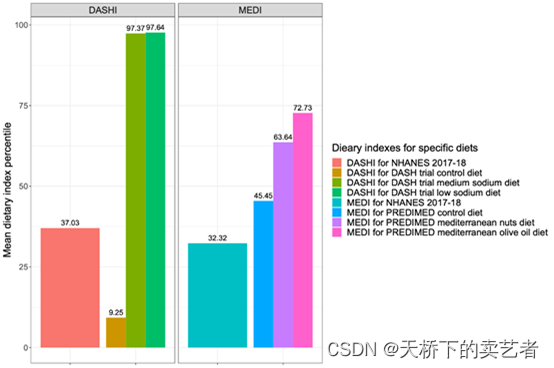
这里咱们以DASHI饮食指数和地中海 MEDI 膳食指数为例子, 利用DASH和MEDI饮食指数,对2017-2018年临床试验(即DASH和PREDIMED)的结果与流行病学研究(即NHANES)的结果进行对比分析。
计算DASHI饮食指数(基于营养素),即停止高血压的饮食方法,使用每1天摄入的营养素。
所有营养素将除以(总能量/2000 kcal)以调整能量摄入
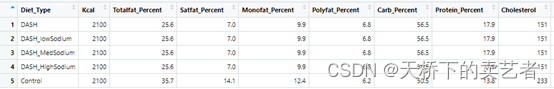
DASHI_DASH = DASHI(SERV_DATA = DASH_trial, #原始数据文件,包括所有份量的食物和营养素RESPONDENTID = DASH_trial$Diet_Type, #每个参与者的唯一参与者IDTOTALKCAL_DASHI = DASH_trial$Kcal, #总能量摄入TOTAL_FAT_DASHI = DASH_trial$Totalfat_Percent, #总脂肪摄入量SAT_FAT_DASHI = DASH_trial$Satfat_Percent, #饱和脂肪摄入量PROTEIN_DASHI = DASH_trial$Protein_Percent, #蛋白质摄入量CHOLESTEROL_DASHI = DASH_trial$Cholesterol, #胆固醇摄入量FIBER_DASHI = DASH_trial$Fiber, #纤维摄入量POTASSIUM_DASHI = DASH_trial$Potassium, #钾摄入量MAGNESIUM_DASHI = DASH_trial$Magnesium, #镁摄入量CALCIUM_DASHI = DASH_trial$Calcium, #钙摄入量SODIUM_DASHI = DASH_trial$Sodium) #钠摄入量
#计算地中海 MEDI 膳食指数(基于食用量),使用给定的 1 天摄入的食物和营养素的食用量
MEDI_PREDIMED = MEDI(SERV_DATA = PREDIMED_trial, #数据文件RESPONDENTID = PREDIMED_trial$Diet_Type, #每位参与者的唯一 IDOLIVE_OIL_SERV_MEDI = PREDIMED_trial$Virgin_Oliveoil,#橄榄油的食用分量FRT_SERV_MEDI = PREDIMED_trial$Fruits, #所有全果的食用分量VEG_SERV_MEDI = PREDIMED_trial$Vegetables, #除马铃薯和豆类以外的所有蔬菜的食用量LEGUMES_SERV_MEDI = PREDIMED_trial$Legumes, #豆类蔬菜的食用分量 NUTS_SERV_MEDI = PREDIMED_trial$Total_nuts, #坚果和种子的食用量FISH_SEAFOOD_SERV_MEDI = PREDIMED_trial$Fish_Seafood, #鱼类海产品的食用分量ALCOHOL_SERV_MEDI = PREDIMED_trial$Alcohol, #酒精的食用量,包括葡萄酒、啤酒、"淡啤酒"、白酒SSB_SERV_MEDI = PREDIMED_trial$Soda_Drinks, #所有含糖饮料的食用量SWEETS_SERV_MEDI = PREDIMED_trial$Sweets, #所有甜食(包括糖果、巧克力、冰淇淋、饼干、蛋糕、派、糕点)DISCRET_FAT_SERV_MEDI = PREDIMED_trial$Refined_Oliveoil, #酌定脂肪食用分量,包括黄油、人造黄油、蛋黄酱、沙拉酱 REDPROC_MEAT_SERV_MEDI = PREDIMED_trial$Meat) #红肉和加工肉类
接下来就是对NHANES进行计算
对2017-2018 年第 1 天和第 2 天 NHANES 数据设置调查设计,先去掉缺失值
##过滤掉权重变量 WTDR2D 的缺失值
NHANES_20172018_design_d1d2 = NHANES_20172018$FPED %>%filter(!is.na(WTDR2D))
计算各个指数
##NHANES 2017年至2018年
#DASHI第1天和第2天,NUTRIENT为第一天数据,NUTRIENT2为第二天数据
#在 1 个步骤内计算 NHANES_FPED 数据(2005 年以后)的 DASHI(基于营养素)
DASHI_NHANES = DASHI_NHANES_FPED(NUTRIENT_PATH=NHANES_20172018$NUTRIENT, NUTRIENT_PATH2=NHANES_20172018$NUTRIENT2)
MEDI for 第1天和第2天
MEDI_NHANES = MEDI_NHANES_FPED(FPED_IND_PATH=NHANES_20172018$FPED_IND, NUTRIENT_IND_PATH=NHANES_20172018$NUTRIENT_IND, FPED_IND_PATH2=NHANES_20172018$FPED_IND2, NUTRIENT_IND_PATH2=NHANES_20172018$NUTRIENT_IND2)
DASH for 第1天和第2天
DASH_NHANES = DASH_NHANES_FPED(NHANES_20172018$FPED_IND, NHANES_20172018$NUTRIENT_IND, NHANES_20172018$FPED_IND2, NHANES_20172018$NUTRIENT_IND2)
MED for 第1天和第2天
MED_NHANES = MED_NHANES_FPED(FPED_PATH=NHANES_20172018$FPED, NUTRIENT_PATH=NHANES_20172018$NUTRIENT, DEMO_PATH=NHANES_20172018$DEMO, FPED_PATH2=NHANES_20172018$FPED2, NUTRIENT_PATH2=NHANES_20172018$NUTRIENT2)
#AHEI for 第1天和第2天
AHEI_NHANES = AHEI_NHANES_FPED(NHANES_20172018$FPED_IND, NHANES_20172018$NUTRIENT_IND, NHANES_20172018$FPED_IND2, NHANES_20172018$NUTRIENT_IND2)
DII for 第1天和第2天
DII_NHANES = DII_NHANES_FPED(FPED_PATH=NHANES_20172018$FPED, NUTRIENT_PATH=NHANES_20172018$NUTRIENT, DEMO_PATH=NHANES_20172018$DEMO, FPED_PATH2=NHANES_20172018$FPED2, NUTRIENT_PATH2=NHANES_20172018$NUTRIENT2)
HEI2020 for 第1天和第2天
HEI2020_NHANES_1718 = HEI2020_NHANES_FPED(FPED_PATH=NHANES_20172018$FPED, NUTRIENT_PATH=NHANES_20172018$NUTRIENT, DEMO_PATH=NHANES_20172018$DEMO, FPED_PATH2=NHANES_20172018$FPED2, NUTRIENT_PATH2=NHANES_20172018$NUTRIENT2)
通过SEQN将所有先前的这些数据合并为一个数据,
NHANES_20172018_dietaryindex_d1d2 = inner_join(NHANES_20172018_design_d1d2, DASHI_NHANES, by = "SEQN") %>%inner_join(MEDI_NHANES, by = "SEQN") %>%inner_join(DASH_NHANES, by = "SEQN") %>%inner_join(MED_NHANES, by = "SEQN") %>%inner_join(AHEI_NHANES, by = "SEQN") %>%inner_join(DII_NHANES, by = "SEQN") %>%inner_join(HEI2020_NHANES_1718, by = "SEQN")
对这个合并数据生成调查设计
NHANES_design_1718_d1d2 <- svydesign(id = ~SDMVPSU, strata = ~SDMVSTRA, weight = ~WTDR2D, data = NHANES_20172018_dietaryindex_d1d2, #set up survey design on the full dataset #can restrict at time of analysis nest = TRUE)
从这个调查对象中提取出相关指标的平均值
# 生成 DASHI_ALL 的 svymean 对象
DASHI_1718_svymean = svymean(~DASHI_ALL, design = NHANES_design_1718_d1d2, na.rm = TRUE)
# 从 svymean 对象中提取平均值
DASHI_1718_svymean_mean = DASHI_1718_svymean[["DASHI_ALL"]]# 生成 MEDI_ALL 的 svymean 对象
MEDI_1718_svymean = svymean(~MEDI_ALL, design = NHANES_design_1718_d1d2, na.rm = TRUE)
# 从 svymean 对象中提取平均值
MEDI_1718_svymean_mean = MEDI_1718_svymean[["MEDI_ALL"]]#为DASH_ALL生成svymean对象
DASH_1718_svymean = svymean(~DASH_ALL, design = NHANES_design_1718_d1d2, na.rm = TRUE)
#从svymean对象中提取均值
DASH_1718_svymean_mean = DASH_1718_svymean[["DASH_ALL"]]#为MED_ALL生成svymean对象
MED_1718_svymean = svymean(~MED_ALL, design = NHANES_design_1718_d1d2, na.rm = TRUE)
#从svymean对象中提取均值
MED_1718_svymean_mean = MED_1718_svymean[["MED_ALL"]]# generate the svymean object for AHEI_ALL
AHEI_1718_svymean = svymean(~AHEI_ALL, design = NHANES_design_1718_d1d2, na.rm = TRUE)
# extract the mean from the svymean object
AHEI_1718_svymean_mean = AHEI_1718_svymean[["AHEI_ALL"]]# generate the svymean object for DII_ALL
DII_1718_svymean = svymean(~DII_ALL, design = NHANES_design_1718_d1d2, na.rm = TRUE)
# extract the mean from the svymean object
DII_1718_svymean_mean = DII_1718_svymean[["DII_ALL"]]# generate the svymean object for HEI2020_ALL
HEI2020_1718_svymean = svymean(~HEI2020_ALL, design = NHANES_design_1718_d1d2, na.rm = TRUE)
# extract the mean from the svymean object
HEI2020_1718_svymean_mean = HEI2020_1718_svymean[["HEI2020_ALL"]]
先设置X轴标签名
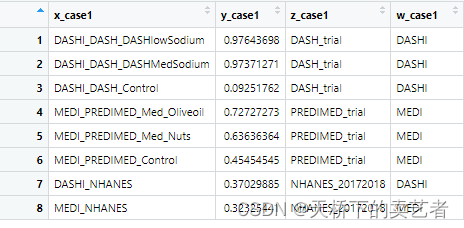
x_case1 = c("DASHI_DASH_DASHlowSodium", "DASHI_DASH_DASHMedSodium", "DASHI_DASH_Control", "MEDI_PREDIMED_Med_Oliveoil", "MEDI_PREDIMED_Med_Nuts", "MEDI_PREDIMED_Control", "DASHI_NHANES", "MEDI_NHANES")
提取DASHI_DASH数据中DASHI_ALL指标,MEDI_PREDIMED数据中的MEDI_ALL指标,MEDI_PREDIMED数据中的MEDI_ALL指标,还有从NHANES_design_1718_d1d2提取的平均值
y_case1 = c(DASHI_DASH$DASHI_ALL[2]/9, DASHI_DASH$DASHI_ALL[3]/9, DASHI_DASH$DASHI_ALL[5]/9, MEDI_PREDIMED$MEDI_ALL[1]/11, MEDI_PREDIMED$MEDI_ALL[2]/11, MEDI_PREDIMED$MEDI_ALL[3]/11, DASHI_1718_svymean_mean/9, MEDI_1718_svymean_mean/11)
生产一个Z的向量,等下绘图用于分组
z_case1 = c("DASH_trial", "DASH_trial", "DASH_trial", "PREDIMED_trial", "PREDIMED_trial", "PREDIMED_trial", "NHANES_20172018", "NHANES_20172018")
创建饮食索引类型的向量
w_case1 = c("DASHI", "DASHI", "DASHI", "MEDI", "MEDI", "MEDI", "DASHI", "MEDI")
将所有y值乘以100得到百分比
y_case1 = y_case1*100
将相关指标合并成一个数据
df_case1 = data.frame(x_case1, y_case1, z_case1, w_case1)
把分类变量转成因子
df_case1$z_case1 = factor(df_case1$z_case1, levels = c("NHANES_20172018", "DASH_trial", "PREDIMED_trial"))
df_case1$x_case1 = factor(df_case1$x_case1, levels = c("DASHI_NHANES", "DASHI_DASH_Control", "DASHI_DASH_DASHMedSodium", "DASHI_DASH_DASHlowSodium", "MEDI_NHANES", "MEDI_PREDIMED_Control", "MEDI_PREDIMED_Med_Nuts", "MEDI_PREDIMED_Med_Oliveoil"))
最后绘图
ggplot(df_case1, aes(x=z_case1, y=y_case1, fill=x_case1)) + geom_bar(stat = "identity", position = "dodge") +theme_bw() +# create a facet grid with the dietary index typefacet_wrap(. ~ w_case1, scales = "free_x") +labs(y = "Mean dietary index percentile", fill = "Dieary indexes for specific diets") +theme(# increase the plot title sizeplot.title = element_text(size=18),# remove the x axis titleaxis.title.x = element_blank(),# remove the x axis textaxis.text.x = element_blank(),# increase the y axis title and text sizeaxis.title.y = element_text(size=18),axis.text.y = element_text(size=14),# increase the legend title and text sizelegend.title = element_text(size=18),legend.text = element_text(size=16),# increase the facet label sizestrip.text = element_text(size = 16)) +# add numeric labels to the bars and increase their sizegeom_text(aes(label = round(y_case1, 2)), vjust = -0.5, size = 4.5, position = position_dodge(0.9)) +# add custom fill labelsscale_fill_discrete(labels = c("DASHI_DASH_DASHlowSodium" = "DASHI for DASH trial low sodium diet","DASHI_DASH_DASHMedSodium" = "DASHI for DASH trial medium sodium diet","DASHI_DASH_Control" = "DASHI for DASH trial control diet","MEDI_PREDIMED_Med_Oliveoil" = "MEDI for PREDIMED mediterranean olive oil diet","MEDI_PREDIMED_Med_Nuts" = "MEDI for PREDIMED mediterranean nuts diet","MEDI_PREDIMED_Control" = "MEDI for PREDIMED control diet","DASHI_NHANES" = "DASHI for NHANES 2017-18","MEDI_NHANES" = "MEDI for NHANES 2017-18"))
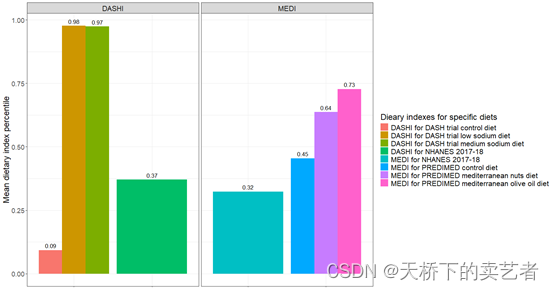
最后得到上图,y表示平均膳食指数百分位,不同颜色的柱子分别表示各个指数。