基因调控网络GRN是包含转录因子TFs与其下游靶基因之间的调控相互作用的可解释图模型。了解GRN的拓扑结构和动力学是解释疾病病因机制和将相应发现转化为新疗法的基础。单细胞多组学技术的最新进展促使从单细胞转录组学和表观基因组学数据中以前所未有的分辨率推断GRN。在这里,作者提出scGRN,一个全面的单细胞多组学GRN平台,包含人类和小鼠。目前版本的scGRN收录了237,051个细胞类型特异性GRN(62,999,692个TF-靶基因对),涵盖160个组织/细胞系。scGRN是第一个记录从单细胞多组学数据推断的不同人类和小鼠条件下大规模细胞类型特异性GRN信息的资源。作者已经实现了多种有效的GRN分析在线工具,包括差异TF-target网络分析,TF富集分析和pathway下游分析。作者还详细介绍了TF与GRN中靶基因的启动子(promoters)、超增强子(super-enhancers)和典型增强子(typical enhancers)的结合情况。综上所述,scGRN是一个搜索、浏览、分析、可视化和下载感兴趣GRN的综合平台,使人们能够深入了解不同条件下调控机制的差异。
来自:scGRN: a comprehensive single-cell gene regulatory network platform of human and mouse
在线网页:https://bio.liclab.net/scGRN/

目录
- 背景概述
- 数据和方法
- 数据收集与管理
- 数据预处理
- scRNA和scATAC数据的细胞聚类和注释
- GRN重建
- 基本知识
- 从RNA模态中重建
- 从ATAC模态中重建
- GRN中target genes的注释
- 平台使用和访问
- 整体概述
- 检索scGRN数据集的接口
- GRN在线分析工具
- 局限
- Reference
背景概述
复杂的基因表达模式在很大程度上是由转录因子TFs的调控所控制和塑造的,转录因子是位于顺式调控元件CREs(cis-regulatory elements)内部或周围的序列特异性DNA结合蛋白[1]。TFs丰度或活性的变化可导致其下游target转录的增加或减少[2,3]。TFs的基因调控不是一个线性过程,而是发生在复杂网络的背景下,该网络包括多个TFs与其下游靶基因之间的调控相互作用[4],称为基因调控网络GRN,可以控制基因表达的时间、条件和数量。揭示GRN的拓扑结构和动力学是理解细胞身份的建立和重编程以及细胞命运的基础[5,6]。这些网络也可以用来模拟不同条件下基因表达的变化,从而深入了解差异基因表达的机制[4]。
重建GRN是生物学中一个长期存在的目标,并且已经创建了各种方法来挖掘TF-target调控关系并将其映射到graph上。早期重建GRN的研究利用了数据库中经过实验验证的调控事件。下一代测序技术的出现促进了从大量TF-binding或转录组学数据中推断出GRN。然而,由于这些高通量数据的bulk性质,组织样本中跨细胞类型混合测量引起的限制尚未被克服。
在过去的十年中,单细胞技术引起了一场革命,使用单细胞组学数据的GRN重建方法已逐渐应用于推断细胞类型特异性TF-gene相互作用。scRNAseq是目前最常用的推断单细胞GRN(scGRNs)的方法。例如,SCENIC将scRNA-seq共表达网络与motif binding信息整合,以推断GRN[12]。单细胞转座酶可及染色质测序(scATACseq)的出现使得在单细胞分辨率和全基因组范围内分析开放染色质成为可能,使用scATACseq数据生成的大规模表观基因组信息进行GRN重建成为完善TF-gene调控关系推断的补充选择。最近,Zhang等人[19]提出了DIRECTNET,其中仅使用scATAC数据就可以重构GRN。
构建GRN是系统生物学的一个主要研究热点,因为它可以为药物设计和医学研究提供重要的信息。例如,kragsteen等人通过基因调控分析确定了Norn TFs的靶基因,发现Tcf21是Norn细胞的关键转录调控因子[20]。Jain等通过研究SSc_ILD和健康对照肺样本的GRN,发现了治疗SScILD的几个新靶点[21]。
在过去的几年中,为了描述全基因组调控事件,已经创建了多个致力于提供TF-gene关系的资源。然而,预测的TF-target gene调控关系来自bulk omics data,因此无法表征特定于细胞类型的全基因组调控事件。TRRUST[22,25]是第一个使用基于句子的文本挖掘构建的基于文献的TF-target interactions数据库,使其成为GRN计算推理和重建的有用基准。基因调控网络数据库GRAND的开发是为了提供特定样本的GRN资源,其中考虑了患者之间的表型差异,如性别、年龄和种族。hTFtarget数据库通过整合ChIP-seq数据和TF binding site prediction,提供了人类的TF-target gene调控关系。
虽然上述数据库为检索TF-target gene关系提供了全面的资源,但由于数据来自bulk组学数据,因此无法在单细胞分辨率下对GRN进行深入分析。GRNdb已经部分解决了这一限制[24],GRNdb使用从公共数据库收集的不同人类和小鼠条件的scRNA-seq数据重建了GRN。然而,单细胞转录组数据的不断积累促使我们创建一个更全面的资源来记录scGRN(scGRN与GRNdb的比较见补充表S1)。此外,在推断TFs和它们的target genes的调控关系时,单独使用单细胞转录组数据可能会忽略表观基因组信息的影响,例如开放的染色质区域。

因此,作者开发了scGRN,这是一个跨越多种人类和小鼠条件的综合单细胞多组学基因调控网络平台,旨在记录从scRNA-seq和scATACseq数据中计算推断的大量GRN信息,并提供target genes的详细表观遗传学注释。目前,scGRN共收录了62,999,692对细胞类型特异性TF-target gene对,涵盖160个组织/细胞系。这些样本来自NCBI GEO/SRA、ENCODE、Arrayexpress数据库,包括多个单细胞测序平台。为了提供更好的用户体验,scGRN提供了多种GRN相关信息的可视化方法,并对TF结合区域的启动子、超级增强子和典型增强子进行了详细的功能标注。作者还开发了三种在线分析工具,包括差异GRN分析、TF富集分析和pathway下游分析。
数据和方法
数据收集与管理
作者从NCBI GEO、ENCODE和ArrayExpress等多个公共数据库中收集scRNA-seq样本。GEO数据库中的scRNA-seq数据集通过基于文本挖掘的pipeline自动抓取和下载,其中用于query数据的关键字包括“scRNA-seq”、“single cell RNA sequencing”、“single-cell RNA-seq”、“Homo sapiens”和“Mus musculus”。在手动审核收集到的每个记录后,过滤掉那些没有原始基因表达谱的样本。作者还从ENCODE和ArrayExpress下载了人和小鼠的scRNA-seq样本。对于scATAC-seq数据,作者从NCBI GEO/SRA中收集了原始fastq文件,其获取方式与从GEO数据库中获取的方式相似。此外,手工整理和校对了每个scRNA-seq和scATAC-seq的元信息,包括物种、数据类型、组织类型、测序平台、细胞数和相关出版文献。
数据预处理
为了克服技术差异和噪声的影响,作者分别对scRNA-seq和scATAC-seq数据采用统一的质量控制流水线进行预处理。首先,使用Seurat软件包(4.3.0)对scRNA-seq样品的原始基因表达谱进行质量控制。然后,使用带有默认比例因子的全局缩放的归一化方法“LogNormalize”对表达进行归一化,并采用PCA线性降维。对于scATAC-seq数据,首先使用cellranger-atac处理原始fastq文件,然后使用Signac (1.9.0) 进行严格的质量控制,以去除低质量的细胞。此外,作者还基于TF-IDF方法对cell-peak矩阵进行了归一化。最后对变换后的ATAC矩阵进行奇异值分解,提取每个细胞的低维表示。
scRNA和scATAC数据的细胞聚类和注释
数据预处理后,使用Seurat对scRNA-seq数据进行细胞聚类。随后使用自动标注方法SingleR (2.2.0) 识别每个簇的细胞类型。对于scATACseq数据,使用Signac R包进行细胞聚类。还使用MAESTRO R包(1.5.1) 中的“ATACCalculateGenescore”函数将peak矩阵转换为基因活性评分矩阵,并使用SingleR进行细胞类型注释。scRNA-seq和scATAC-seq的聚类结果使用UMAP降维方法进行可视化。
GRN重建
基本知识
TF-motif注释
- TF-motif注释的目的是识别和预测这些转录因子的结合位点。"TF"代表转录因子,而"motif"代表转录因子结合位点的序列模式。因此,TF-motif注释数据包含了转录因子结合位点的位置信息以及与之对应的基序。
从基因共表达网络到TF
- 首先,利用基因共表达网络的模块化分析方法,将基因分组成共表达模块。然后,通过TF基因的信息,如基因的注释或数据库中已知的TF信息,检查每个模块中的基因是否富集有TF。如果某个模块中的基因富集有TF,则可以将这些TF视为调控该模块的候选TF。
- TF结合位点预测:利用已知的TF结合位点信息,可以在基因组中预测TF的结合位点。然后,将这些结合位点与共表达网络中的基因进行比较,以确定哪些TF可能直接调控这些基因的表达。这需要结合TF-motif注释数据。
- 基于TF-基因共表达模式的预测:利用机器学习方法,例如基于深度学习的方法,从基因共表达网络中学习TF与其调控基因之间的共表达模式。这种方法可以帮助识别TF与其调控基因之间的潜在关系,并预测新的TF-基因调控关系。
- 实验验证:最终,为了验证预测的TF-基因调控关系,可以进行实验验证,例如染色质免疫沉淀(ChIP)实验,以确定TF是否真正调控共表达网络中的基因表达。

- 补充图1:GRN重建管道。
从RNA模态中重建
作者采用pySCENIC pipeline (version 0.11.2),利用scRNA-seq数据集的基因表达矩阵和已知的TF-motif注释推断GRN (补充图1A)。首先,使用GENIE3和GRNBOOST2分别基于TF-gene共表达模块识别潜在的TF targets。然而,单独使用共表达预测的GRN含有许多假阳性和间接的targets。为了克服这一限制,作者使用Rcistarget对每个共表达模块进行修剪,以确定具有motif支持的直接targets (regulons,调控子)。这些motif来自两个数据库(位于转录起始位点TSS周围10 kb和TSS上游500 bp),作者保留了那些注释了相应TFs且标准化富集分数(NES) >3.0的motif。最后,利用AUCell对每个细胞中这些调控子的活性进行量化,并利用调控子活性评分(RAS)矩阵和细胞类型条形码信息计算每个调控子的细胞类型特异性评分。细胞类型特异性调控子定义为RSS >0.1且在每种细胞类型中排名前10位的调控子。
从ATAC模态中重建
作者使用DIRECT-NET(1.0.0)从单细胞染色质可及性数据(即scATAC数据)(补充图1B)。首先,使用MAESTRO将每个scATAC样本的细胞峰矩阵转化为基因活性评分矩阵,从该矩阵中检测到每个细胞型簇的差异表达基因(DEGs)。对于每个DEG,转录起始位点(TSS)上游500bp被视为启动子,TSS上游和下游250kb内的peak被定义为候选功能区。然后,作者构建了一个新的特征矩阵,其中行表示相似的细胞,列表示启动子和候选调节区域,聚合来自KNN graph (默认k = 50) 的相似细胞的信号。第三,通过远端候选功能区域的可达性对启动子表达水平的可达性进行回归,发现重要度得分高于重要度得分中位数最大值且值为0.001的区域为高置信度CREs(HC CREs)。最后,利用ChromVAR软件包(1.20.2)中的motifmatchr函数,通过基序富集分析鉴定与启动子和HC CREs结合的TF,重构细胞类型特异性GRNs。此外,还计算了TF与靶基因之间的Spearman相关评分,以提供参考。
GRN中target genes的注释
作者首先从SEdb 2.0中获得了1739个人和931个小鼠 H3K27ac ChIP-seq样本中鉴定的1,717,744个超级增强子 (SE) 区域和79,709,120个典型增强子 (TE) 区域。然后,利用最近的活性基因、重叠基因、近端基因和最近基因四种连接策略绘制了这些SE和TE区域的靶基因图谱。作者将TSS上游和下游2kb的基域定义为靶基因的启动子区域。
为了鉴定与靶基因启动子、SE和TE结合的TFs,作者从ReMap 2022中收集了817个人和648个小鼠TFs的51,616,973个和32,985,444个非冗余结合区,这些TFs来自不同细胞系和组织类型。使用BEDTools (2.25.0) 鉴定了所有GRNs中与靶基因的启动子、SE或TE区域重叠的TF结合峰。
平台使用和访问
整体概述
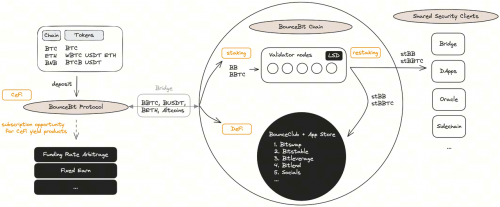
scGRN的主要框架和功能如图1所示。scGRN目前包含1324份来自10X Genomics、Drop-seq、Microwell-seq、inDrop、Smart-seq、HyDrop、sciATAC-seq等多个测序平台的scRNAseq和scATAC-seq数据。这些数据包括来自160个组织/细胞系的6,808,724个细胞,包括疾病和健康状况的样本。作者分别从scRNA-seq和scATAC-seq数据中使用统一的管道和软件参数进行GRN推断。scGRN提供可视化,如聚类、细胞类型注释、转录因子活性热图,以及推断每个样本的细胞类型特异性GRN。此外,scGRN还提供了TF结合区启动子、超级增强子和典型增强子的详细功能注释。此外,scGRN还为用户提供了三种在线分析工具,包括TF富集分析、差异网络分析和通路下游分析。总的来说,scGRN是一个用户友好的平台,可以查询、分析和可视化与scGRN相关的信息。

- 图1:scGRN收集了大量的人和小鼠单细胞多组学数据,并使用统一的流水线来推断GRNs。scGRN支持多种浏览、搜索、分析、可视化和下载GRNs相关信息。

- 图2:scGRN的主要功能和使用方法。A. scGRN导航条。B. 浏览scGRN页面。C. 提供四种查询模式,包括“按TF搜索”、“按靶基因搜索”、“按组织类型搜索”和“按细胞类型搜索”。D. 每个样本的详细信息,包括“样本概述”、“可视化”和“TF-target网络”。E. 每个靶基因的详细信息。F. 提供了三种在线分析工具,包括“差异TF-target网络分析”、“TF富集分析”和“Pathway downstream分析”。G. scGRN允许用户下载TF-target list和注释信息。
检索scGRN数据集的接口
scGRN使用户能够搜索、浏览、分析、可视化和下载感兴趣的GRN(图2A)。作者在“Search”页面上提供了四种不同的查询方法来搜索scGRN相关信息,包括“按TFs搜索”、“按靶基因搜索”、“按组织类型搜索”和“按细胞类型搜索”(图2C)。比如第一种查询模式,“按TFs搜索”,是为对某些特定TFs感兴趣的生物学家设计的。按target基因搜索是专门为对scGRN中特定target基因感兴趣的用户设计的。唯一的区别是用户应该输入感兴趣的靶基因名称,而不是TF名称。当用户对特定组织类型或细胞类型感兴趣时,“按组织类型搜索”和“按细胞类型搜索”应该是最佳查询模式。最后,点击“搜索”按钮将用户引导到所搜索的组织类型或细胞类型的相应数据集。
结果页面给出了相关样本的总结表,并在该表中显示了每个样本的简要信息,比如样本ID,物种和平台。在点击特定的样本ID后,用户被引导至详细页面(图2D),包括“样本概述”、“可视化”和“TF -target网络”。作者在“样本概述”面板的左侧部分提供了所选样本的基本描述,而详细信息,如细胞分布、网络统计数据和质量控制结果,在右侧部分表示。对于可视化面板中的scRNA-seq样本,分别为SCENIC (GENIE3)和SCENIC (GRNBOOST2)提供了多种可视化方法,包括“Cell cluster and TF activity”、“Regulon module”、“Regulon特异性”和“Regulon activity”。GRN信息在TF-target网络面板中显示。
GRN在线分析工具
作者已经实现了三种有效的基因调控网络分析在线工具,包括“差异TF-target网络分析”、“TF富集分析”和“Pathway downstream分析” (图2F)。“差异TF-target网络分析”用于比较任意两个网络的差异。首先,用户确定种类、数据类型和网络推理方法。然后,用户应该选择两个样本进行分析。最后,点击“Analyze”将返回差异网络分析结果,包括“Sample Overview”、“Global differential network”和“cell types之间的差异网络”。使用“TF富集分析”,用户首先需要提供基因列表或上传包含基因名称的文件,然后设置测试使用的参数。同时,也要选择富集TF的样本。运行分析将返回用户提交的所有基因的富集结果,这些结果被组织成一个汇总表,其中包含了这些基因的详细TF富集信息。为了提高直观性,还提供了浓缩结果的气泡图和条形图。“通路下游分析”的功能是在选定的样本中识别TF或靶基因的富集通路。点击“Analyze”将返回所选通路数据库中的富集通路。
局限
局限性是scGRN目前只记录了使用scRNA-seq或scATACseq数据预测的scGRN,由于现阶段可用样本有限,不包括同时使用scRNA-seq和scATACseq数据推断的scGRN。然而,单细胞多组学技术的不断进步导致了使用多模态分析样本进行更可靠的scGRN推断的新型计算方法的发展,例如ANANSE, CellOracle [67], DeepMAPS [68]和SCENIC+[69]。
Reference
[1]The evolution of gene regulation by transcription factors and microRNAs
[2]Gene regulation by transcription factors and microRNAs
[3]KnockTF: a comprehensive human gene expression profile database with knockdown/knockout of transcription factors
[4]Gene regulatory networks and the role of robustness and stochasticity in the control of gene expression
[5]Gene regulatory networks in the evolution and development of the heart
[6]Gene regulatory networks and the evolution of animal body plans
[12]SCENIC: single-cell regulatory network inference and clustering
[19]DIRECT-NET: an efficient method to discover cis-regulatory elements and construct regulatory networks from single-cell multiomics data
[20]The transcriptional and regulatory identity of erythropoietin producing cells
[21]Epigenetic regulation of profibrotic macrophages in systemic sclerosis-associated interstitial lung disease
[22]TRRUST v2: an expanded reference database of human and mouse transcriptional regulatory interactions
[24]GRNdb: decoding the gene regulatory networks in diverse human and mouse conditions
[25]TRRUST: a reference database of human transcriptional regulatory interactions
[67]Dissecting cell identity via network inference and in silico gene perturbation
[68]Single-cell biological network inference using a heterogeneous graph transformer
[69]SCENIC+: single-cell multiomic inference of enhancers and gene regulatory networks