技术背景
在前面的一篇博客中,我们介绍过CudaSPONGE的基础使用方法、CudaSPONGE调用Python接口函数以及CudaSPONGE结合增强采样软件PySAGES的使用方法。在这篇文章中,我们将介绍CudaSPONGE和PySAGES相结合的初步性能测试结果。

测试案例
我们沿用这一篇文章中的测试案例,对一个简单的多肽体系的两个二面角参数空间执行MetaDynamics增强采样方法:

我们的预期结果,在普通的NVT下,二面角的采样空间较为集中:

而加上PySAEGES的增强采样之后,理论上采样点可以均匀的遍布在整个采样空间:

这是我们对于这个采样结果的预期,其实图片内容也就是实际结果。但是本文主要侧重于性能测试,而我们的性能测试,就基于这个案例来开展。
调用逻辑
其实对于PySAGES来说,他们有自己的一套函数调用逻辑:把不同的MD软件作为Backend,由PySAGES来进行封装和调用,并且用于控制MD模拟的进行。所以如果按照PySAGES的调用逻辑来说,应该用这么一个框架来结合PySAGES和CudaSPONGE:

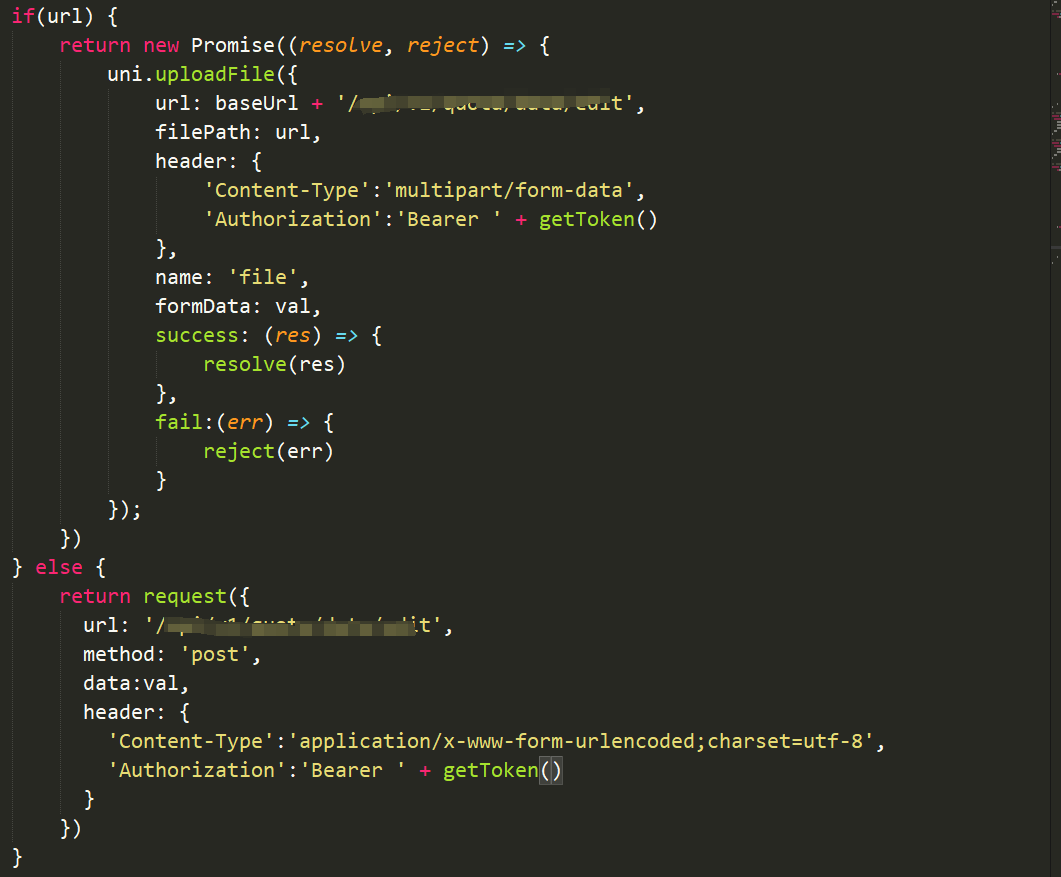
但是因为CudaSPONGE主要基于CUDA-C开发,为了发挥更多的性能优势,CudaSPONGE也有自己的一套调用逻辑,结合PySAGES之后是这样的:

其实这个问题的本质就在于,以哪一种软件为核心。如果以PySAGES为核心,就是采用上面的一种交互形式。如果是以MD软件为核心,就是采用下面的这种形式。不过对于CudaSPONGE v1.4版本来说,因为提供的API有限,其实只能使用下面的这种调用形式,也就是以MD软件为核心的调用形式。本文所使用到的测试案例代码,都可以参考参考链接1中的内容。
MetaDynamics说明
关于MetaDynamics的原理,建议还是翻阅一下Parrinello组的原始文章《Escaping free-energy minima》。简单的来说就是,我们在一个定义好的序参量空间,每隔固定的MD步数,就施加一个高斯势,这部分的总和组成一个偏置势:
如果使用一个well-tempered来加速自由能面的收敛,那这个参数\(\omega\)需要从一个常数变成一个随偏置势增长而逐渐减小的一个可变量:
这里PySAGES中的MetaDynamics就是用的这个增强采样方法,也是我们重点测试的方法对象。需要提及的是,虽然每隔固定的步数我们才会施加一个高斯势,但其实历史累积的高斯势的作用力一直都存在。所以,只要在MD过程中添加了MetaDynamics方法,就意味着每一步都需要计算一个序参量的值,及其对应的偏置势产生的偏置作用力。在很多情况下,偏置作用力的计算有可能会成为整个分子模拟过程的瓶颈部分。因此,MetaDynamics方法的性能至关重要。
初步测试数据
经过初步的测试,分别运行了2000、4000、6000、8000、10000个Step的NVT和Meta NVT,运行时长和Meta时长占比如下:

总结概要
接前一篇关于PySAGES结合CudaSPONGE使用方法的文章,本文主要还是使用了一样的测试案例。仅通过不同的测试步长,来定性的分析PySAGES的MetaDynamics实现方案结合MD软件之后的性能数据。
版权声明
本文首发链接为:https://www.cnblogs.com/dechinphy/p/sponge_ext_01.html
作者ID:DechinPhy
更多原著文章:https://www.cnblogs.com/dechinphy/
请博主喝咖啡:https://www.cnblogs.com/dechinphy/gallery/image/379634.html
参考链接
- https://www.cnblogs.com/dechinphy/p/18615556/pysages-sponge
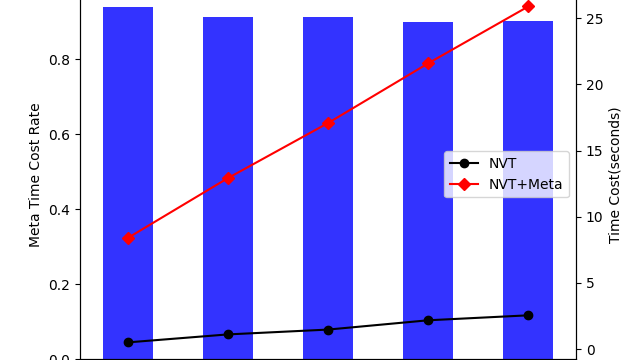 接前一篇关于PySAGES结合CudaSPONGE使用方法的文章,本文主要还是使用了一样的测试案例。仅通过不同的测试步长,来定性的分析PySAGES的MetaDynamics实现方案结合MD软件之后的性能数据。
接前一篇关于PySAGES结合CudaSPONGE使用方法的文章,本文主要还是使用了一样的测试案例。仅通过不同的测试步长,来定性的分析PySAGES的MetaDynamics实现方案结合MD软件之后的性能数据。