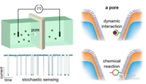
纳米孔测序(Nanopore Sequencing)是一种基于单分子检测的第三代测序技术,其核心原理是通过纳米孔结构实时监测分子(如DNA、RNA)通过孔道时引起的电信号变化,从而实现碱基序列的识别。与传统的测序技术(如Illumina的合成测序)相比,纳米孔测序具有长读长、实时分析、无需扩增等独特优势,在基因组学、病原体检测和精准医学领域展现出巨大潜力。
一、纳米孔测序的核心原理
- 基本结构:
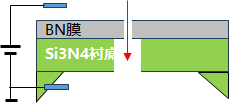
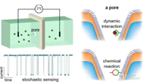
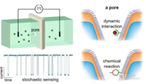
- 纳米孔:固态材料(如氮化硅、石墨烯)或生物蛋白孔(如α-溶血素)形成的孔径为1-3 nm的通道。
- 跨膜电压:在孔两侧施加电压(通常50-200 mV),驱动带电分子(如DNA)穿过纳米孔。
- 电信号检测:分子通过纳米孔时,阻塞离子电流(或产生隧穿电流),信号变化与分子特性(碱基序列)相关。
- 信号解码:
- 每个碱基(A/T/C/G)因其化学结构和体积不同,导致离子电流阻塞程度和持续时间差异。
- 机器学习算法(如隐马尔可夫模型、深度学习)将电信号转换为碱基序列。
二、技术分类**
-
生物纳米孔测序

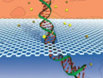
- 代表技术:Oxford Nanopore Technologies(ONT)的MinION/GridION平台。
- 核心材料:蛋白质孔(如CsgG、α-溶血素)。
- 优势:天然生物相容性,易修饰(如连接核酸外切酶)。
- 挑战:蛋白孔稳定性差,易受环境(pH、温度)影响。
-
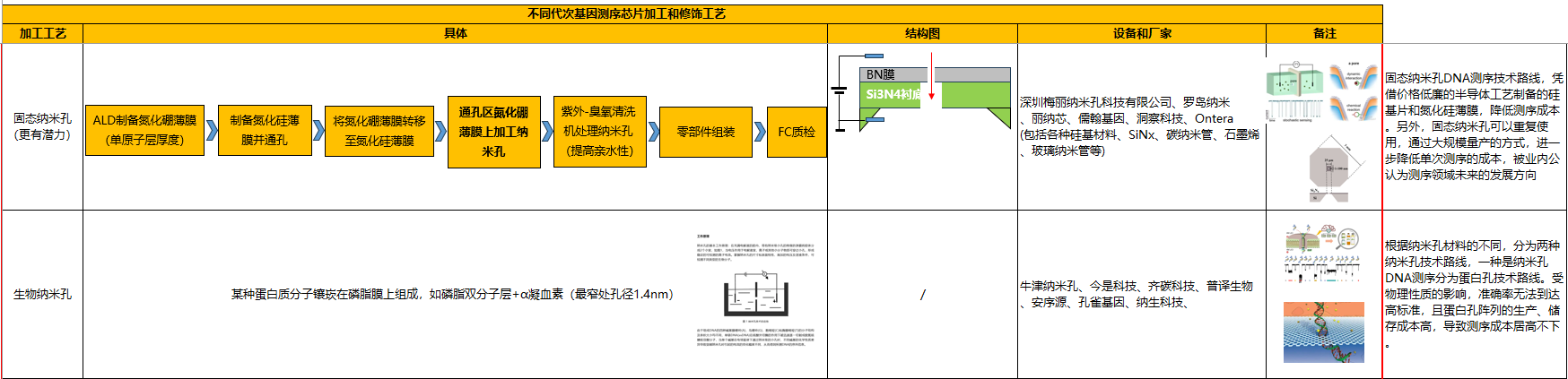
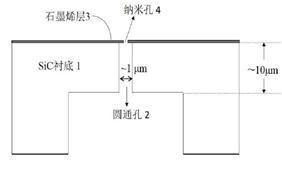
固态纳米孔测序
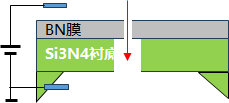
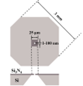
- 代表技术:基于SiN、石墨烯等材料的固态芯片。
- 核心材料:氮化硅(SiN)、二硫化钼(MoS₂)、石墨烯。
- 优势:机械强度高、孔径可调、耐极端条件。
- 挑战:表面修饰复杂,信噪比低(需高精度电极集成)。

三、技术优势
- 超长读长:可读取数十kb至Mb级DNA片段,解决重复序列和结构变异的检测难题。
- 实时分析:数据即时输出,适用于现场快速诊断(如传染病监测)。
- 直接测序:无需PCR扩增或荧光标记,保留表观遗传修饰(如甲基化)。
4.*便携性:掌上设备(如MinION)支持野外或临床床边检测。
四、关键技术挑战
- 信号噪声:
- 分子通过速度过快(微秒级),信号采集频率需达MHz级。
- 热噪声、电容耦合干扰需通过低噪声电路设计抑制。
- 碱基识别精度:
- 相邻碱基信号重叠(如AA与AT),需算法优化。
- 单碱基分辨率需达到<1 nm的空间识别能力。
- 通量与成本:
- 单孔通量低,依赖纳米孔阵列并行化(如牛津纳米孔的“孔芯片”含2048孔)。
- 固态纳米孔加工成本高(电子束刻蚀、ALD工艺)。
五、典型应用场景
- 基因组测序:
- 长读长组装复杂基因组(如人类、植物多倍体)。
- 检测结构变异(缺失、重复、倒位)。
- 病原体快速检测:
- 实时识别病毒(如新冠病毒、埃博拉)、耐药菌。
- 应用于机场、医院等场景的即时诊断(POCT)。
- 表观遗传学:
- 直接检测DNA甲基化(5mC)、RNA修饰(m6A)。
- 单分子蛋白质分析:
- 解析蛋白质折叠状态或翻译后修饰(如磷酸化)。
六、主流技术对比
| 技术指标 | Oxford Nanopore(生物孔) | 固态纳米孔 | Illumina(二代测序) |
|---|---|---|---|
| 读长 | 10 kb - 2 Mb | 1-100 kb(发展中) | 50-300 bp |
| 测序速度 | 实时(分钟级) | 实时(需优化) | 数小时至数天 |
| 设备成本 | 低(MinION约$1000) | 高(依赖微加工设备) | 高(仪器数百万美元) |
| 表观检测 | 支持(直接读取) | 潜在支持 | 需亚硫酸氢盐处理 |
| 主要应用 | 野外检测、长读长组装 | 高精度单分子分析 | 高通量短读长测序 |
七、未来发展方向
- 材料创新:
- 二维材料(如石墨烯、h-BN)提升空间分辨率。
- 混合孔(生物-固态复合结构)兼顾稳定性和灵敏度。
- 加工技术突破:
- 电子束/离子束刻蚀实现亚纳米级孔径控制。
- 纳米孔阵列的大规模并行化(如百万级孔芯片)。
- 算法与硬件协同优化:
- 深度学习模型实时解析复杂信号(如RNA二级结构)。
- 集成CMOS芯片实现信号原位放大与降噪。
- 临床应用拓展:
- 癌症早筛(循环肿瘤DNA检测)。
- 抗生素耐药基因的快速鉴定。
八、总结
纳米孔测序凭借其独特优势正在颠覆传统测序技术,尤其在长读长、实时性和便携性方面表现突出。尽管面临信号噪声、成本等挑战,随着材料科学、微纳加工和人工智能的进步,纳米孔测序有望成为基因组学研究和临床诊断的核心工具。固态纳米孔与生物孔的融合、高通量芯片的成熟,将推动该技术从实验室走向大规模应用。
★★★文章来源
网络、新闻采访、专利等公开信息,仅用于学习
文章由芯晨微纳(河南)光电科技有限公司搜集整理



![[I.2][个人作业 软件案例分析]](https://s2.loli.net/2025/03/13/NhMm2I4fUtAi3lX.jpg)